Генетика, 2021, T. 57, № 12, стр. 1396-1402
Генетическое разнообразие 21 аутосомного STR-маркера системы CODIS в популяциях Восточной Европы
В. Н. Харьков 1, *, С. А. Котова 2, Н. А. Колесников 1, Е. А. Спивак 2, К. В. Вагайцева 1, Т. В. Забавская 2, В. И. Рыбакова 2, А. С. Парфенова 2, А. Н. Верчук 2, Н. К. Янковский 3, 4, В. А. Степанов 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
2 Научно-практический центр Государственного комитета судебных экспертиз
Республики Беларусь
220114 Минск, Беларусь
3 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
4 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: vladimir.kharkov@medgenetics.ru
Поступила в редакцию 07.06.2021
После доработки 15.06.2021
Принята к публикации 17.06.2021
Аннотация
Впервые получены генетические характеристики 15 локальных популяций восьми народов Восточной Европы по расширенной панели, включающей 21 аутосомный STR-маркер, применяемый в судебно-экспертной практике для ДНК-идентификации и установления родства. Проведена оценка полиморфизма маркеров в российских, белорусских и молдавских популяциях, охарактеризованы уровни внутри- и межпопуляционной генетической дифференциации исследованных групп населения. Рассчитаны параметры информационной значимости системы аутосомных микросателлитов для экспертной ДНК-идентификации и установления родства. Создана база данных частот аллелей аутосомных STR-локусов, которая может быть использована для проведения вероятностно-статистических расчетов при оценке уровня достоверности экспертного исследования в странах Союзного государства. Обоснована необходимость учета популяционно-генетической структуры населения при вероятностной оценке достоверности результатов судебно-экспертного ДНК-анализа.
ВВЕДЕНИЕ
Каждый индивидуум несет уникальный геном, который характеризуется неповторяющимся сочетанием аллелей множества полиморфных ДНК-маркеров. Основным методом криминалистической идентификации на базе анализа ДНК является исследование аутосомных коротких тандемных повторов (STR), обладающих высоким уровнем аллельного полиморфизма. Генотипирование аллельного состояния 15–20 локусов таких повторов обладает достаточным уровнем информативности для установления индивидуального тождества. Для анализа аутосомных STR-локусов используются наборы реагентов для генотипирования, основными производителями которых являются компании Thermo Fisher Scientific (США) и Promega Corporation (США). В основе наборов реагентов указанных производителей лежит панель маркеров стандарта CODIS (англ. Combined DNA Index System), предложенная в 2015 г. и утвержденная к использованию для криминалистического ДНК-анализа в 2017 г. Федеральным Бюро Расследований Министерства юстиции США. Несмотря на то что система CODIS разработана и функционирует на территории США, многие другие страны, в том числе Республика Беларусь и Российская Федерация, опираются на стандарт данной системы при проведении ДНК-идентификации личности в судебной экспертизе. В 2017 г. панель маркеров стандарта CODIS, до этого включавшая 13 микросателлитов, была расширена. В настоящее время в состав так называемого ядра системы входят 20 аутосомных STR-маркеров: CSF1PO, FGA, THO1, TPOX, VWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51, D21S11, D1S1656, D2S441, D2S1338, D10S1248, D12S391, D19S433, D22S1045. Эти изменения привели к необходимости обновления референсных баз данных, содержащих информацию, необходимую для вероятностно-статистической оценки результатов экспертного исследования, а также актуализации информации об уровне генетической дифференциации локальных популяций по маркерам расширенной системы CODIS и информационной значимости нового ядра системы для ДНК-идентификации в ряде локальных популяций Восточной Европы. Большинство публикаций, посвященных этому вопросу, на настоящий момент содержат информацию только по 16 STR-маркерам [1–14].
В настоящей работе мы охарактеризовали частоты аллелей, уровень генетической дифференциации популяций и оценили идентификационный потенциал системы из 21 аутосомного STR-маркера (D1S1656, D2S441, D2S1338, D3S1358, D5S818, D7S820, D8S1179, D10S1248, D12S391, D13S317, D16S539, D18S51, D21S11, D22S1045, CSF1PO, FGA, Penta D, Penta E, TH01, TPOX, vWA) в популяционных выборках восьми этнических групп Восточной Европы – у русских, белорусов, молдаван, гагаузов, поляков, коми, марийцев и украинцев.
МАТЕРИАЛЫ И МЕТОДЫ
Материал исследования составили образцы ДНК из 15 различных локальных популяций восьми народов Восточной Европы. Общая численность выборок составила 1093 индивида. Образцы ДНК были взяты у неродственных лиц из следующих популяций, проживающих в России (русских – 205 образцов, коми – 83, марийцев – 76), Молдавии (молдаван – 117, гагаузов – 57), Беларуси (русских – 57, белорусов – 413, поляков – 32, украинцев – 110). Материал из популяций Беларуси содержит шесть выборок из историко-этнографических регионов республики: Восточного Полесья (N = 41), Западного Полесья (N = 56), Центрального региона (N = 135), Поднепровья (N = 80), Поозерья (N = 55), Понеманья (N = 46). Коми представлены двумя выборками: коми-зыряне (N = 43), коми-ижемцы (N = 40).
Забор первичного биологического материала (венозной крови) у доноров проводили с соблюдением процедуры письменного информированного согласия на проведение исследования. На каждого донора составлялась анкета с его родословной, указанием этнической принадлежности и мест рождения предков. В исследование включены только образцы ДНК доноров, по результатам анкетирования отрицавших факт метисации с представителями других этносов минимум в трех поколениях. Индивида относили к данной этнической группе на основании его собственной этнической идентификации, его родителей и места рождения.
Генотипирование образцов по аутосомным STR-маркерам проводили методом мультиплексной полимеразной цепной реакции с последующим анализом продуктов амплификации на автоматическом генетическом анализаторе как описано ранее [10]. Экспериментальные исследования проведены на базе Центра коллективного пользования научно-исследовательским оборудованием “Медицинская геномика” Томского НИМЦРАН и НПЦ Государственного комитета судебных экспертиз Республики Беларусь.
Оценку соответствия распределений генотипов равновесию Харди–Вайнберга, генетической вариабельности STR-локусов и межпопуляционное сравнение проводили с использованием программного средства Arlequin v. 3.11 [15]. Для анализа генетической дифференциации популяций применяли расчет попарных значений Fst и анализ молекулярной дисперсии (AMOVA), используя в качестве матрицы генетических расстояний матрицу среднеквадратичных различий в числе повторов Rst.
Дискриминационный потенциал системы из 21 STR-маркера оценивали с помощью стандартных криминалистических показателей, а именно: вероятности случайного совпадения генотипов (MP, matching probability), вероятности дискриминации неродственных индивидов (PD, power of discrimination), исключающей способности (PE, power of exclusion), и индекса отцовства (PI, paternity index) [16]. Частота аллелей и криминалистические параметры рассчитывали с помощью программного пакета MS Excel.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Генетическая вариабельность 21 STR
Частоты аллелей 21 STR демонстрируют существенное разнообразие в исследованных популяциях. Наиболее частые аллели по большинству локусов во всех исследованных популяциях совпадают. Наибольшее суммарное число аллелей по 21 аутосомному маркеру было обнаружено в белорусской популяции (250), чуть меньшее число аллелей показано в российской и украинской популяциях (228, 214 соответственно), 204 аллеля было обнаружено в популяции молдаван. В популяционных выборках гагаузов (195), марийцев (192) и поляков (184) показано менее 200 аллелей. Наименьшее количество аллелей было обнаружено в коми-ижемской популяции (163).
После применения коррекции Бонферрони (р = 0.002) не наблюдалось существенных отклонений от равновесия Харди–Вайнберга, за исключением четырех маркеров в трех популяциях. Маркер FGA показал отклонения в российской популяции (р = 0.00082 ± 0.00002). Маркер Penta Е показал отклонения в общей выборке белорусов (р = 0.00175 ± 0.00004). В подвыборке Восточного Полесья отклонение от равновесия было показано по локусу D1S1656 (р = 0.00095 ± 0.00003). Наконец, D16S539 (р = 0.00095 ± 0.00003) показал отклонения в выборке коми-ижемцев.
Анализ генетических расстояний для шести выборок белорусов не показал между ними статистически значимой генетической дистанции. Таким образом, для данной популяции нет необходимости учитывать территориальное происхождение индивида внутри страны при идентификационном анализе. Тем не менее в дальнейших расчетах мы приводим результаты как для тотальной выборки белорусов, так и для территориальных выборок.
Обратная ситуация показана для выборок коми. Анализ генетических расстояний по Нею обнаружил наличие значимой генетической дистанции между коми-зырянами и коми-ижемцами (0.0054, р = 0.004). Эти результаты дают основание для рассмотрения этих выборок отдельно друг от друга при формировании референсных популяционных данных при проведении генетической экспертизы.
Также значимые генетические расстояния по 21 анализируемому STR-маркеру отделяют популяции коми-зырян, коми-ижемцов, марийцев, молдаван друг от друга и от остальных анализируемых популяций. Напротив, между русскими, белорусами, поляками и украинцами значительная генетическая дистанция не показана (табл. 1).
Таблица 1.
Генетические расстояния по Нею
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Русские (1) | * | |||||||||||||
| Гагаузы (2) | 0.0010 (0.125) | * | ||||||||||||
| Коми-ижемцы (3) | 0.0075 (0.000) | 0.0103 (0.000) | * | |||||||||||
| Коми-зыряне (4) | 0.0020 (0.042) | 0.0051 (0.001) | 0.0054 (0.004) | * | ||||||||||
| Марийцы (5) | 0.0156 (0.000) | 0.0186 (0.000) | 0.0254 (0.000) | 0.0148 (0.000) | * | |||||||||
| Молдаване (6) | 0.0017 (0.002) | 0.0008 (0.182) | 0.0107 (0.000) | 0.0032 (0.004) | 0.0169 (0.000) | * | ||||||||
| BELvp (7) | –0.0001 (0.505) | 0.0002 (0.378) | 0.0095 (0.000) | 0.0018 (0.118) | 0.0168 (0.000) | –0.0006 (0.594) | * | |||||||
| BELzp (8) | 0.0016 (0.030) | 0.0029 (0.025) | 0.0097 (0.000) | 0.0027 (0.044) | 0.0189 (0.000) | 0.0032 (0.001) | –0.0017 (0.860) | * | ||||||
| BELp (9) | 0.0003 (0.363) | 0.0002 (0.406) | 0.0073 (0.000) | 0.0014 (0.211) | 0.0154 (0.000) | 0.0008 (0.190) | –0.0007 (0.637) | –0.0011 (0.804) | * | |||||
| BELpd (10) | 0.0007 (0.149) | 0.0017 (0.052) | 0.0099 (0.000) | 0.0042 (0.003) | 0.0215 (0.000) | 0.0015 (0.023) | –0.0005 (0.628) | –0.0001 (0.537) | 0.0003 (0.404) | * | ||||
| BELpz (11) | 0.0001 (0.453) | 0.0020 (0.088) | 0.0093 (0.001) | 0.0031 (0.037) | 0.0159 (0.000) | –0.0001 (0.464) | –0.0009 (0.698) | 0.0008 (0.269) | 0.0012 (0.218) | 0.0002 (0.460) | * | |||
| BELc (12) | –0.0004 (0.811) | 0.0017 (0.040) | 0.0098 (0.000) | 0.0023 (0.033) | 0.0165 (0.000) | 0.0017 (0.004) | 0.0004 (0.337) | 0.0016 (0.057) | –0.0000 (0.502) | 0.0002 (0.418) | –0.0002 (0.603) | * | ||
| Поляки (13) | 0.0006 (0.289) | 0.0034 (0.028) | 0.0075 (0.001) | 0.0050 (0.011) | 0.0205 (0.000) | 0.0026 (0.035) | –0.0011 (0.627) | 0.0002 (0.408) | –0.0010 (0.663) | 0.0001 (0.462) | –0.0012 (0.748) | –0.0004 (0.583) | * | |
| Белорусы (14) | 0.0001 (0.327) | 0.0012 (0.065) | 0.0092 (0.000) | 0.0027 (0.006) | 0.0173 (0.000) | 0.0011 (0.007) | –0.0017 (0.982) | –0.0010 (0.928) | –0.0013 (0.950) | –0.0012 (0.993) | –0.0011 (0.953) | –0.0010 (0.998) | –0.0005 (0.659) | * |
| Украинцы (15) | –0.0006 (0.845) | 0.0019 (0.030) | 0.0077 (0.000) | 0.0020 (0.044) | 0.0188 (0.000) | 0.0011 (0.030) | –0.0000 (0.439) | –0.0001 (0.509) | –0.0006 (0.668) | –0.0002 (0.543) | –0.0006 (0.727) | –0.0007 (0.921) | 0.0002 (0.393) | –0.0005 (0.901) |
Стоит отметить, что маркером с наибольшим числом аллелей является локус D12S391, для которого показано 18 аллельных вариантов в белорусской популяции и 15 аллелей в польской популяции. В остальных анализируемых популяциях наибольшее количество аллелей отмечено для локуса Penta E. Данные о частотах аллелей доступны по запросу у авторов.
Генетическая дифференциация популяций
Оценку генетической дифференциации исследованных выборок по совокупности всех STR-маркеров проводили с помощью анализа молекулярной дисперсии. При сравнении всего массива выборок различия между популяциями составили 0.35%. В случае объединения выборок до уровня этнических групп дифференциация на уровне этносов возросла до 0.39%. При сравнении двух выборок русских и белорусов различия полностью отсутствовали. Сравнение популяций русских с коми и марийцами продемонстрировало небольшой отрицательный уровень генетической дифференциации (0.03%), свидетельствующий о несколько большем генетическом сходстве индивидов из различных популяций по сравнению с индивидами из той же популяции. Дифференциация популяций русских, молдаван и гагаузов составила 0.13%.
При анализе частот аллелей для всех популяций из Республики Беларусь (всех выборок белорусов, русских, поляков и украинцев) доля различий между ними составила 0.1%. При сравнении выборок белорусов из разных историко-этнографических регионов республики уровень дифференциации между ними был 0.1%. При сравнении группы белорусов с русскими, поляками и украинцами различие достигло значения 0.8%. Эти результаты вполне ожидаемы в силу мультиаллельности STR-маркеров и их относительно высокой степени мутирования и рекомбинации. Также закономерно увеличение показателей генетической дифференциации при сравнении различных по происхождению этнических групп. Результаты дисперсионного анализа показывают отсутствие значительных генетических различий между русскими и белорусами, и славянскими популяциями в целом, а также очень низкий уровень различий между русскими, коми и марийцами по STR-генотипам. Это означает, что, несмотря на имеющуюся информацию о значительной антропологической дифференциации и существующие современные различия между исследованными этнотерриториальными группами, часть их генофонда по аутосомным STR-маркерам является очень мало дифференцированной.
Генетические взаимоотношения между популяциями
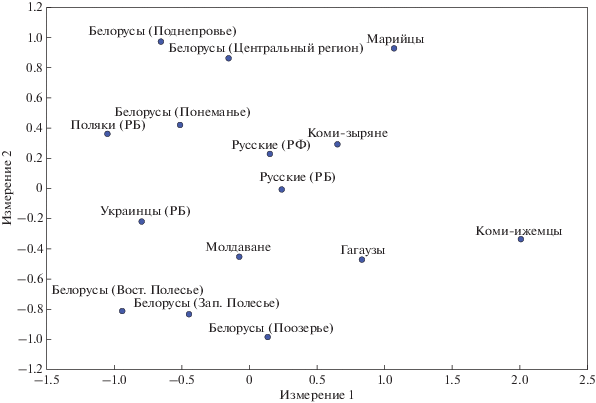
Визуализация матрицы генетических расстояний между популяционными выборками методом многомерного шкалирования (рис. 1) показывает, что исследованные выборки распределены в соответствии с их этнической близостью и географической локализацией. Наиболее генетически близкими друг другу являются выборки русских из России и Беларуси. Максимально удалены друг от друга наиболее территориально разобщенные выборки коми-ижемцев и поляков. Коми-зыряне и коми-ижемцы демонстрируют значительные различия по первому измерению, что вероятно связано с относительно сильным сдвигом частот аллелей в выборке коми-ижемцев в связи с их относительно недавним расселением на эту территорию и наличием эффекта основателя в этой субэтнической группе. В соответствии с географической локализацией близки друг другу и выборки белорусов из Восточного и Западного Полесья, Центральной Белоруссии, Поднепровья и Понеманья. Выбивается только выборка Поозерья, которая является самой северной, но при этом группируется с южными выборками из Полесья. Анализ подчеркивает генетическую близость между собой молдаван и гагаузов.
Идентификационный потенциал системы аутосомных STR-локусов
Для оценки возможности использования системы маркеров при ДНК-идентификации в судебной экспертизе, рассчитывали стандартные криминалистические параметры информативвности: вероятность случайного совпадения генотипов (MP, matching probability), вероятность дискриминации неродственных индивидов (PD, power of discrimination), исключающая способность (PE, power of exclusion), индекс отцовства (PI, paternity index). Показатели MP и PD используются при ДНК-идентификации личности, а значения PE и PI рассчитываются при определении отцовства.
Криминалистические параметры для каждого локуса представлены в дополнительных материалах. Наибольшая информативность расширенного набора маркеров для ДНК-идентификации показана для выборок коми (коми-зыряне, коми-ижемцы) (табл. 2). В популяции русских значение вероятности дискриминации неродственных индивидов (PD) возросло до 0.999999999994978, а показатель исключающей способности (PE) изменился от 0.99999999989 до 0.999999998679. Наиболее низкая идентификационная информативность набора из 21 STR показана для выборки поляков. Показатели информативности для идентификации и определения отцовства во всех популяциях на несколько порядков превышают значения, установленные нормативными актами, действующими на территории Российской Федерации. Данные о частотах аллелей 21 STR-локуса в популяциях, исследованных в настоящей работе, включены в разработанную авторами базу данных. Представленные частоты могут быть референсными (для соответствующей популяции или этнической группы) для вероятностно-статистической оценки результатов экспертного исследования при идентификации личности, установлении родства и др. Кроме того, данные могут использоваться для популяционно-генетических сравнительных исследований.
Таблица 2.
Идентификационная информативность совокупности 21 аутосомного STR-локуса в популяциях Восточной Европы
| Популяция | PD | PE |
|---|---|---|
| Гагаузы | 0.999999999954539 | 0.999999997608839 |
| Коми-зыряне | 1.0 | 0.999999999658466 |
| Коми-ижемцы | 1.0 | 0.999999999137624 |
| Марийцы | 0.999999999972582 | 0.999999998174319 |
| Молдаване | 0.999999999994137 | 0.999999999509155 |
| Поляки | 0.999999999789335 | 0.999999999974522 |
| Русские | 0.999999999994978 | 0.999999998679004 |
| Украинцы | 0.999999999996165 | 0.999999999893009 |
| Белорусы | 0.999999999997410 | 0.999999999263556 |
| Восточное Полесье | 0.999999999952167 | 0.999999999989964 |
| Днепр | 0.999999999952167 | 0.999999999133377 |
| Центральная | 0.999999999996428 | 0.999999999233081 |
| Озерное плато | 0.999999999969731 | 0.999999998645556 |
| Неман | 0.999999999961487 | 0.999999999765122 |
| Западное Полесье | 0.999999999980485 | 0.999999999810011 |
Таким образом, были установлены частоты аллелей и криминалистических параметров 21 аутосомного STR-локуса для 15 популяций восьми народов Восточной Европы. Показан значимый дискриминирующий потенциал используемого набора для ДНК-идентификации и его эффективность при определении родства. Оценка генетической дифференциации различных популяций выявила необходимость учета популяционной принадлежности при создании баз данных генотипов для сравнения референсных частот аллелей.
Работа выполнена в рамках Научно-технической программы Союзного государства “Разработка инновационных геногеографических и геномных технологий идентификации личности и индивидуальных особенностей человека на основе изучения генофондов регионов Союзного государства” (“ДНК-идентификация”), Государственный контракт № 011-17 от 26.09.2017.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Szczerkowska Z., Kapińska E., Wysocka J., Cybulska L. Northern Polish population data and forensic usefulness of 15 autosomal STR loci // Forensic Sci. Int. 2004. V. 144. № 1. P. 69–71. https://doi.org/10.1016/j.forsciint.2004.02.020
Jacewicz R., Jedrzejczyk M., Ludwikowska M., Berent J. Population database on 15 autosomal STR loci in 1000 unrelated individuals from the Lodz region of Poland // Forensic Sci Int Genet. 2008. V. 2. № 1. P. e1–e3. https://doi.org/10.1016/j.fsigen.2007.08.002
Малярчук Б.А., Wozniak M., Czarny J. и др. Вариабельность 15 аутосомных микросателлитных локусов ДНК в русской популяции // Мол. биология. 2007. Т. 41. № 1. С. 3–7.
Piatek J., Jacewicz R., Ossowski A. et al. Population genetics of 15 autosomal STR loci in the population of Pomorze Zachodnie (NW Poland) // Forensic Sci. Int. Genet. 2008. V. 2. № 3. P. e41–e43. https://doi.org/10.1016/j.fsigen.2007.09.002
Rebała K., Wysocka J., Kapińska E. et al. Belarusian population genetic database for 15 autosomal STR loci // Forensic Sci. Int. 2007. V. 173. № 2–3. P. 235–237. https://doi.org/10.1016/j.forsciint.2007.02.002
Pepiński W., Skawrońska M., Niemcunowicz-Janica A. et al. Polymorphism of 11 autosomal STRS in a population sample of Belarussian minority residing in the region of Podlasie (Northeastern Poland) – an extension of the STR core set // Arch. Med. Sadowej Kryminol. 2008. V. 58. № 2–3. P. 65–68.
Zhivotovsky L.A., Malyarchuk B.A., Derenko M.V. et al. Developing STR databases on structured populations: The native South Siberian population versus the Russian population // Forensic Sci. Int. Genet. 2009. V. 3. № 4. P. e111–e116. https://doi.org/10.1016/j.fsigen.2008.08.001
Parys-Proszek A., Kupiec T., Wolańska-Nowak P., Branicki W. Genetic variation of 15 autosomal STR loci in a population sample from Poland // Leg. Med. (Tokyo). 2010. V. 12. № 5. P. 246–248. https://doi.org/10.1016/j.legalmed.2010.05.002
Stepanov V.A., Melnikov A.V., Lash-Zavada A.Y. et al. Genetic variability of 15 autosomal STR loci in Russian populations // Leg. Med. (Tokyo). 2010. V. 12. № 5. P. 256–258. https://doi.org/10.1016/j.legalmed.2010.05.006
Степанов В.А., Балановский О.П., Мельников А.В. и др. Характеристика популяций Российской Федерации по панели пятнадцати локусов, используемых для ДНК-идентификации и в судебно-медицинской экспертизе // Acta Naturae. 2011. Т. 3. № 2. С. 59–71.
Soltyszewski I., Pepinski W., Wolanska-Nowak P. et al. Polish population data on 15 autosomal STRs of AmpFlSTR NGM PCR kit // Forensic Sci. Int. Genet. 2014. V. 9. P. 142–149. https://doi.org/10.1016/j.fsigen.2013.12.002
Цыбовский И.С., Вермейчик В.М., Котова С.А. и др. Создание судебной референтной базы данных по 18 аутосомным STR для ДНК-идентификации в Республике Беларусь // Генетика. 2017. Т. 53. № 2. С. 249–258.
Benvisto A., Messina F., Finocchio A. et al. A genetic portrait of the South-Eastern Carpathians based on autosomal short tandem repeats loci used in forensics // Am. J. Hum. Biol. 2018. V. 30. № 5. P. e23139. https://doi.org/10.1002/ajhb.23139
Rębała K., Veselinović I., Siváková D. et al. Northern Slavs from Serbia do not show a founder effect at autosomal and Y-chromosomal STRs and retain their paternal genetic heritage // Forensic Sci. Int. Genet. 2009. V. 3. № 4. P. e133–e136. https://doi.org/10.1016/j.fsigen.2008.11.001
Excoffier L., Laval G., Schneider S. Arlequin ver. 3.0: An integrated software package for population genetics data analysis // Evol. Bioinformatics Online. 2005. V. 1. P. 47–50.
Desmarais D., Zhong Y., Chakraborty R. et al. Development of a highly polymorphicSTR marker for identity testing purposes at the humanandrogen receptor gene (HUMARA) // J. Forensic Sci. 1998. V. 43. P. 1046–1049.
Дополнительные материалы
- скачать ESM.docx
- Идентификационная информативность 21 аутосомного STR локуса в популяциях Восточной Европы