Генетика, 2023, T. 59, № 3, стр. 345-355
Применение ДНК-штрихкодирования для изучения зеленых ящериц (Sauria: Lacertidae: Lacerta)
М. А. Доронина 1, И. В. Доронин 1, *, С. А. Луконина 2, Л. Ф. Мазанаева 3, К. Ю. Лотиев 4, 5, Н. Б. Ананьева 1
1 Зоологический институт Российской академии наук
199034 Санкт-Петербург, Россия
2 Пензенский государственный университет
440026 Пенза, Россия
3 Дагестанский государственный университет
367025 Махачкала, Россия
4 Сочинский национальный парк,
354002 Сочи, Россия
5 Комплексный научно-исследовательский институт
им. Х.И. Ибрагимова Российской академии наук
364051 Грозный, Россия
* E-mail: igor.doronin@zin.ru
Поступила в редакцию 02.05.2022
После доработки 28.05.2022
Принята к публикации 03.06.2022
- EDN: INYJAE
- DOI: 10.31857/S0016675823030037
Аннотация
ДНК-штрихкодирование остается признанным и широко используемым методом идентификации таксонов при инвентаризации и мониторинге биоразнообразия. В публикации мы приводим сведения о результатах использования этого метода при изучении зеленых ящериц рода Lacerta. Всего было проанализировано 67 последовательностей фрагмента гена COI трех видов (L. agilis, L. media, L. strigata). Впервые c помощью ДНК-штрихкодирования изучены все известные на Кавказе подвиды L. agilis, предложена филогеографическая гипотеза для L. strigata в кавказской части ареала. Генетическая идентификация была успешной на видовом уровне. В составе L. agilis в пределах Кавказа мы можем диагностировать только подвиды L. a. boemica и L. a. exigua, тогда как L. a. brevicaudata, L. a. grusinica и L. a. mzymtensis по выбранному маркеру неотличимы от L. a. exigua, а L. a. ioriensis от L. a. boemica.
Систематика и филогения зеленых ящериц рода Lacerta Linnaeus 1758, включающего по современным представлениям десять видов [1], в настоящее время претерпевает значительные изменения. Перспективным районом для исследования данных вопросов, по нашему мнению, является Кавказ [2, 3]. В пределах этого региона обитают три вида рода – прыткая, L. agilis Linnaeus 1758, представленная здесь шестью подвидами (L. a. boemica Suchow, 1929, L. a. brevicaudata Peters, 1958, L. a. exigua Eichwald, 1831, L. a. grusinica Peters, 1960, L. a. ioriensis Peters et Muskhelischwili, 1968 и L. a. mzymtensis Tuniyev et Tuniyev, 2008), номинативный подвид средней ящерицы – L. m. media Lantz et Cyrén, 1920, и монотипический вид полосатая ящерица – L. strigata Eichwald, 1831 ([4], www.lacerta.de). Филогенетические исследования L. agilis в Кавказском регионе не получили развития после работы С.А. Калябиной-Хауф [5] и выхода ее совместной с Н.Б. Ананьевой монографии [6]. В настоящее время статус L. a. boemica предлагается повысить до видового (как в первоначальной публикации Г.Ф. Сухова [7]), в то время как ряд других кавказских подвидов свести в младшие синонимы L. a. exigua [6, 8]. Молекулярно-генетические исследования L. a. ioriensis и L. a. mzymtensis не проводились вовсе. Выходя за пределы Кавказа, отметим, что остается не решенным вопрос о таксономическом положении закарпатских популяций прыткой ящерицы [6, 9–11]. Слабо изучены филогеография и филогенетические взаимоотношения популяций L. strigata [12] в Кавказском регионе. Это относится и к L. media [13], при том что данный вид входит в надвидовой комплекс Lacerta (trilineata), находящейся в фокусе исследований филогении рода Lacerta [1, 14].
Ранее в литературе были высказаны предположения о путях проникновения и расселения зеленых ящериц в Закавказье [15], реликтовом характере популяций полосатой ящерицы Терско-Кумской низменности, Кумо-Манычской впадины [16] и Черноморского побережья Кавказа в Абхазии [17]. Одной из задач настоящего исследования являлась верификация этих предположений. Немаловажным является то обстоятельство, что ряд обозначенных таксонов зеленых ящериц включен в региональные и национальные списки охраняемых таксонов животных (Красные книги); это подчеркивает актуальность данного исследования с позиции изучения и сохранения биоразнообразия. Молекулярно-генетические данные позволяют выделить действительно уникальные популяции (за рамками принятой в настоящее время подвидовой систематики, во многом устаревшей – в случае с L. agilis) и обосновать необходимость их охраны. Остается актуальным и диагностика видов, поскольку во многих опубликованных работах и в базах данных по их распространению нами отмечены ошибки [18].
Сказанное выше указывает на необходимость более обстоятельного анализа филогеографии, систематики и распространения ящериц рода Lacerta на Кавказе и сопредельных территориях. Действенным методом в этой области продолжает оставаться ДНК-штрихкодирование (Biological identifications through DNA barcodes). В герпетологических исследованиях его эффективность показана для исследований на глобальном [19] и региональном [20–22] уровнях. Данный инструмент с успехом применяется и в исследованиях cложных в систематическом отношении групп ящериц [23].
МАТЕРИАЛЫ И МЕТОДЫ
Для выделения геномной ДНК были использованы части регенерированных хвостов, фаланги пальцев передних конечностей или кожные покровы ящериц, фиксированные в 96%-ном этаноле. Ваучерные экземпляры хранятся в коллекции Зоологического института РАН (ZISP; поддержание коллекции осуществлено в рамках проекта Министерства науки и высшего образования РФ № 075-15-2021-1069) и Зоологическом музее МГУ (ZMMU). В филогенетический анализ включены 67 последовательностей фрагмента митохондриального гена первой субъединицы цитохром оксидазы (COI) L. agilis, L. media и L. strigata, собранных в 50 локалитетах (Приложение, рис. 1). Из них 62 получены в ходе проведения данного исследования, пять взяты из GenBank (http://www.ncbi.nlm.nih.gov/gene/) [20–22].
Рис. 1.
Пункты сбора экземпляров Lacerta, использованных для молекулярно-генетического анализа: a – все виды, б – L. agilis, в – L. media, г – L. strigata. Нумерация соответствует таковой в приложении, цвет кружка соответствует таксону на рис. 2 и 3.

Рис. 1.
Продолжение.

Рис. 1.
Продолжение.
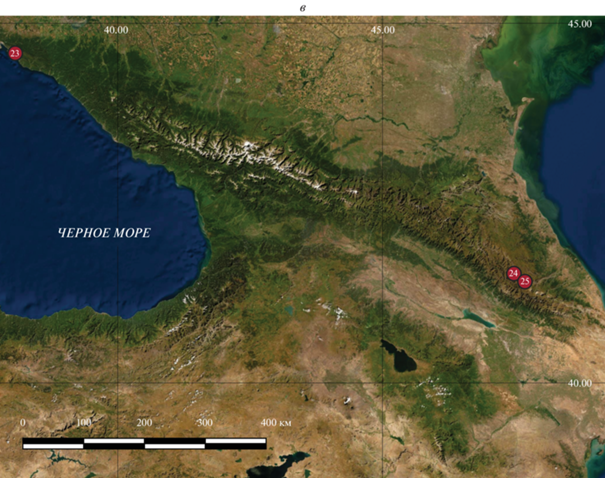
Рис. 1.
Окончание.

В ходе изучения внешних морфометрических признаков, на которых строится систематика прыткой ящерицы [24], мы столкнулись с отсутствием надежных маркеров для диагностики ряда ее подвидов на Кавказе. По этой причине идентификация подвидов проводилась на основе сведений об их ареалах, согласно существующим на сегодняшний день представлениям [6, 24–26]. Для сопоставления кавказских подвидов, относящихся к восточной группе [6], нами были использованы последовательности COI представителей западной группы подвидов – L. agilis argus (Laurenti, 1768) и закарпатской L. agilis ssp. Важно почеркнуть, что в анализе использованы типовые экземпляры (голотип L. a. mzymtensis – ZISP24648) и топотипы (L. a. boemica – ZISP31113, L. a. ioriensis – ZISP29878), что делает наши выводы о систематике надежными и обоснованными.
Выделение тотальной ДНК проведено стандартным солевым методом с лизированием протеиназой К [27]. Амплификация участка гена COI (643 пн) осуществлялась с использованием универсальных праймеров UTF 5'-TGT AAA ACG ACG GCC AGT TCT CAA CCA AYC AYA ARG AYA TYG G-3' и UTR 5'-CAG GAA ACA GCT ATG ACT ARA CTT CTG GRT GKC CRA ARA AYC A-3' [28] при следующих условиях: начальная денатурация 95°С – 3 мин; затем 30 циклов 95°С – 30 с, 50°С – 30 с, 72°С – 50 с; завершающий синтез 72°С – 5 мин.
Реакционная смесь для ПЦР (25 мкл) содержала 50–100 нг ДНК, 0.5 мкМ каждого праймера, 0.2 мМ dNTPs, 1.5 мМ MgCl2, 2.5 мкл 10-кратного ПЦР-буфера (10 мМ Tris-HCl, pH 8.3, 50 мМ KCl) и 2 ед. Taq-полимеразы (Thermo Scientific). Секвенирование проводилось на генетическом анализаторе ABI PRIZM 3500xL (Applied Biosystems). Полученные последовательности были депонированы в GenBank NCBI (OM267788–OM267849).
Выравнивание последовательностей проведено в программах Geneious Prime 2021.0.1 (https://www.geneious.com) и AliView 1.6 [29]. Филогенетические отношения реконструированы с использованием метода Байеса (Bayesian analysis, BA) в программе Mr. Bayes 3.1.2 [30–32] и методом максимального правдоподобия (Maximum Likelihood, ML) в программном пакете MEGA X [33]. Для BA использованы 1.5 млн генераций; статистическая надежность узлов ML-деревьев оценена путем бутстреп-анализа (2000 псевдореплик). Устойчивость узлов дендрограмм филогенетических отношений в случае BA была оценена с помощью расчета апостериорных вероятностей. Уровни поддержки узлов: 0.95 – высокий, 0.90–0.95 – средний, менее 0.90 – неподдержанный; значения бутстреп-поддержек в ML-анализе: более 75% – достоверная, 50–75% – тенденция, менее 50% – отсутствие поддержки [34]. В качестве внешней группы выбраны последовательности Zootoca vivipara (Lichtenstein, 1823) (MN015068) и Timon lepidus (Daudin, 1802) (MN015075). Кроме того, в анализе задействована L. trilineata Bedriaga, 1886 (MN015090).
Выбор модели эволюции для нуклеотидных последовательностей проведен в программе MrModeltest 2.4 [35] c использованием информационного критерия Акаике (AIC) – GTR (General Time Reversible) с параметрами I = 0.3691 и G = 0.1844. Графические изображения деревьев получены с помощью программы FigTree 1.4.4 (http://tree.bio.ed.ac.uk/software/figtree). Построение сети гаплотипов осуществлялось методом TCS в программе PopART [36]. При анализе генетической изменчивости с использованием программы DnaSP v.5.10.01 [37] вычислены следующие параметры: общее количество полиморфных позиций (S), общее количество замен (η), количество гаплотипов (H), разнообразие гаплотипов (h), разнообразие нуклеотидов на сайт (π), среднее количество нуклеотидных замен (K), значение теста Таджимы (Tajima’s D) и тест на нейтральность Фу (Fu’s Fs). Генетические дистанции (p-дистанции) вычисляли в программе MEGA X.
Для проведения анализа таксономических границ по молекулярно-генетическим признакам использованы алгоритмы ASAP (Automated simultaneous analysis phylogenetics) [38] и ABGD (Automatic barcode gap discovery) [39]. Они позволяют выявить условные молекулярные операционные таксономические единицы (Molecular phylogenetic taxonomic units, MOTU), представляющие собой монофилетические клады неопределенного ранга. Анализ проводили с использованием следующих параметров: Pmin (минимальная априорная дистанция) = 0.01, Pmax (максимальная априорная дистанция) = 0.1, X (относительная ширина интервала) = 0.5; была использована матрица попарных К80-дистанций.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
В ходе исследования получено 62 последовательности гена COI зеленых ящериц (OM267788–OM267849); ранее опубликованные пять последовательностей (KP697820, MN993135, MN993136, MN993138, MG815780) имели длину от 630 до 657 пн. 30 изученных последовательностей L. agilis образуют 17 гаплотипов (Hap 5–8, 10–15, 20, 22, 24–28); выявлены 54 (8.4% от общей длины фрагмента) вариабельные позиции, из них 43 (6.7%) парсимони информативные; соотношение транзиций к трансверсиям составило 7.6. Четыре последовательности гена COI L. media образуют три гаплотипа (Hap 16–18); выявлены 14 (2.2% от общей длины фрагмента) вариабельных позиций, из них – одна (0.2%) парсимони информативная; соотношение транзиций к трансверсиям составило 2.1. 33 последовательности L. strigata образуют семь гаплотипов (Hap 1–4, 9, 20, 22); выявлены 13 (2% от общей длины фрагмента) вариабельных позиций, из них – четыре (0.6%) парсимони информативные; соотношение транзиций к трансверсиям составило 7.7.
Показатели генетического разнообразия в выборке представлены в табл. 1. Относительно высокие значения гаплотипического и нуклеотидного разнообразия у прыткой ящерицы свидетельствуют о стабильной популяции с высокой численностью или о возможном вторичном контакте исторически разделенных популяций [40]. О возможном недавнем расселении полосатой ящерицы из рефугиума могут говорить низкие значения показателей генетического разнообразия, а отрицательные достоверные показатели D – о наличии в прошлом внезапного роста популяции (экспансии) и/или положительного отбора [41].
Таблица 1.
Показатели генетического разнообразия в изученных выборках зеленых ящериц рода Lacerta
| Вид | n | S/η | h | π | K | Tajima’s D | Fu’s Fs |
|---|---|---|---|---|---|---|---|
| L. agilis | 30 | 54/54 | 0.94 ± 0.02 | 0.029 ± 0.002 | 17.72 | 1.12 (p > 0.1) | 0.89 (p > 0.1) |
| L. media | 4 | 14/14 | 0.83 ± 0.02 | 0.011 ± 0.005 | 7.16 | 0.62 (p > 0.1) | 2.08 (p > 0.1) |
| L. strigata | 33 | 13/13 | 0.38 ± 0.11 | 0.002 ± 0.001 | 1.32 | –1.90 (p < 0.05) | –1.62 (p < 0.05) |
Примечание. n – объем выборки, S – общее количество полиморфных позиций, η – общее количество замен, h – разнообразие гаплотипов, π – разнообразие нуклеотидов на сайт, K – среднее количество нуклеотидных замен, значение Tajima’s D – значение теста Таджимы и Fu’s Fs – значение теста на нейтральность Фу.
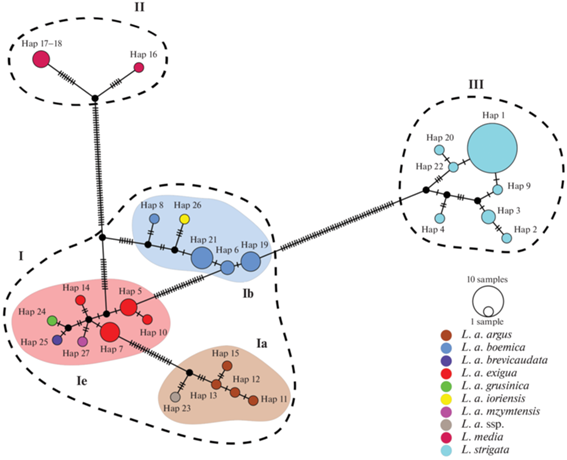
Минимальное значение генетической дистанции (p-дистанции) найдено в паре видов L. agilis–L. strigata (10.1%), максимальное – L. media–L. strigata (11.8%) (табл. 2). По результатам реконструкции филогенетических взаимоотношений представителей рода Lacerta получены дендрограммы с идентичной топологией, на которых можно выделить пять клад с высокими бутстреп-поддержками (рис. 2). На полученной медианной сети гаплотипов (рис. 3) можно обозначить три гаплогруппы: I – L. agilis с тремя подгруппами (Ia – argus-группа, Ib – boemica-группа, Ie – exigua-группа), II – L. media и III – L. strigata. Наиболее удаленной от всех прочих является гаплогруппа III. От гаплогруппы I она отдалена на 25 нуклеотидных замен, от II – на 22. Анализ ASAP/ABGD показал наличие четкого разрыва между значениями подвидовой и видовой изменчивости (treshold distance = = 2.9%, barcoding gap = 3.0%). Применение данных алгоритмов позволило выявить в объеме изученной выборки пять монофилетических MOTU.
Таблица 2.
Средние генетические p-дистанции (%) (под диагональю) и их стандартные отклонения (над диагональю) между видами рода Lacerta по данным анализа последовательностей фрагмента гена COI (643 пн)
| Вид | L. strigata | L. agilis | L. media |
|---|---|---|---|
| L. strigata | 1.1 | 1.3 | |
| L. agilis | 10.1 | 1.2 | |
| L. media | 11.8 | 11.1 |
Рис. 2.
Дендрограмма филогенетических отношений представителей рода Lacerta по результатам анализа фрагмента гена COI (643 пн). В узлах указаны байесовы апостериорные вероятности и бутстрэп-поддержки (BA/BS). Цветовое обозначение таксонов соответствует таковому на рис. 1. В двух столбцах разными цветами указаны выделенные молекулярные операционные таксономические единицы (MOTU).

Рис. 3.
Медианная сеть гаплотипов COI (643 пн) представителей рода Lacerta Кавказа и сопредельной территории. Каждый цветной кружок – уникальный гаплотип, чей размер пропорционален встречаемости в выборке; цвет кружка соответствует подвидам на рис. 1 и 2. Соединительные линии – вероятные эволюционные связи, засечки – нуклеотидные замены, черные кружки в узлах линий – предсказанные гаплотипы. Пунктирной линией ограничены группы гаплотипов (I–III), цветной заливкой обозначены подгруппы гаплотипов L. agilis (Ia – argus-группа, Ib – boemica-группа, Ie – exigua-группа).
ОБСУЖДЕНИЕ
Изученные виды зеленых ящериц демонстрируют высокий уровень дифференциации по гену COI, т.е. на нашем материале была подтверждена его видоспецифичность. На полученной дендрограмме (рис. 2) первыми обособляются L. triliniata и L. media, традиционно включаемые в Lacerta (triliniata) [14]. Это не соответствует опубликованным данным, полученным с использованием фрагментов как митохондриальных (cytb, 12S, 16S), так и ядерных (C-mos, β-fib) генов [12, 14]. Так, было показано, что отделение L. strigata произошло на более ранних этапах радиации рода Lacerta [12]. Это говорит о малой эффективность ДНК-баркода для изучения филогенетических взаимоотношений видов рода Lacerta. Следующими на дендрограмме обособляются L. strigata, и, наконец, L. agilis.
Последовательности прыткой ящерицы, в свою очередь, сформировали три клады. Внутри L. agilis первой обособляется клада L. a. boemica, в которую вошла и L. a. ioriensis (p-дистанция 0.8%). Это подтверждает гипотезу о генетической близости иорской ящерицы и ящерицы Беме [15, 24, 25, 42]. Примечательно, что наиболее дистанцирована в этой выборке особь из расположенного в долине р. Терек г. Владикавказа (Hap 8). Вероятно в прошлом, в холодных перигляциальных условиях на Осетинской наклонной равнине существовали изолированные микрорефугиумы этого таксона, приуроченные к долинам крупных рек. Косвенным подтверждением этого предположения может служить особь из близлежащего локалитета – г. Алагира в долине р. Ардон, также обладающая уникальным гаплотипом (Hap 6). Как и в предшествующих публикациях [6, 8] L. a. boemica продемонстрировала максимальные различия с другими подвидами L. agilis – 4.0–4.6% (табл. 3). В то же время отличия L. a. boemica от других подвидов не достигают уровня, сопоставимого с таковым между признаваемыми видами рода.
Таблица 3.
Средние генетические p-дистанции (%) (под диагональю) и их стандартные отклонения (над диагональю) между подвидами Lacerta agilis по данным анализа последовательностей фрагмента гена COI (643 пн)
| Подвид | L. a. argus | L. a. boemica | L. a. brevicaudata | L. a. exigua | L. a. grusinica | L. a. ioriensis | L. a. mzymtensis | L. a. ssp. |
|---|---|---|---|---|---|---|---|---|
| L. a. argus | 0.8 | 0.8 | 0.8 | 0.8 | 0.8 | 0.8 | 0.4 | |
| L. a. boemica | 4.6 | 0.8 | 0.8 | 0.8 | 0.3 | 0.8 | 0.8 | |
| L. a. brevicaudata | 4.3 | 4.5 | 0.3 | 0.2 | 0.8 | 0.4 | 0.8 | |
| L. a. exigua | 4.1 | 4.0 | 0.8 | 0.3 | 0.7 | 0.3 | 0.8 | |
| L. a. grusinica | 4.3 | 4.5 | 0.3 | 0.8 | 0.8 | 0.4 | 0.8 | |
| L. a. ioriensis | 4.7 | 0.8 | 4.2 | 3.9 | 4.2 | 0.8 | 0.8 | |
| L. a. mzymtensis | 3.9 | 4.3 | 0.8 | 0.6 | 0.8 | 4.1 | 0.8 | |
| L. a. ssp. | 1.2 | 4.5 | 4.2 | 3.8 | 4.2 | 4.6 | 3.7 |
Закарпатская L. agilis ssp. сгруппировалась с L. a. argus из Австрии и Германии (p-дистанция 1.2%). Это согласуется с выводом К.Д. Мильто [43], основанном на изучении внешней морфологии. Публикации, в которых авторы отстаивают таксономическую самостоятельность закарпатских популяций на основе морфометрических признаков [9–11], не включали ее сравнение с подвидами западной группы (за исключением L. a. chersonensis Andrzejowski, 1832), в том числе с L. a. argus.
Эндемичные кавказские подвиды L. a. brevicaudata, L. a. grusinica и L. a. mzymtensis вошли в одну кладу с L. a. exigua (p-дистанция 0.3–0.8%). В ней же, по сравнению с другими кладами, наименее выражена структурированность. Ранее уже было показано, что популяции L. a. exigua характеризуются настолько низкой изменчивостью митохондриального гена cytb, что географически удаленные популяции от Кавказа до Восточного Казахстана не имеют по этому маркеру значимых отличий [2, 6]. Это может свидетельствовать о быстром заселении прыткой ящерицей огромного пространства степной и лесостепной зон Евразии в конце позднего плейстоцена – голоцене [2].
Как и в специальной работе о филогеографии L. media по результатам анализа гена cytb [13], выборки с территории Большого Кавказа и Загроса, относимые к номинативному подвиду, продемонстрировали довольно значительную генетическую дистанцию друг от друга (p-дистанция 2.1%), превышающую межпопуляционную у других изученных таксонов зеленых ящериц.
Обращает на себя внимание выраженная звездчатая структура сети гаплотипов у L. strigata, характеризующаяся преобладанием массового гаплотипа Hap 1, доминирующего на Северном Кавказе (Приложение). Гаплотипы с территории Северной Осетии (Hap 9) и Дагестана (Hap 20, Hap 22) незначительно отличаются от него – на одну и три нуклеотидных замены соответственно. На территории и в окрестностях с. Буглена выявлено присутствие трех из них (Hap 1, 20 и 22). Последовательность особи из г. Сухуми идентична таковой из г. Мцхета (Hap 3), притом что данные локалитеты находятся на удалении более 320 км друг от друга (соответственно, в границах Колхидской низменности и на южном склоне Большого Кавказа) и характеризуются совершенно различными физико-географическими условиями. Это говорит о расселении L. strigata на Черноморское побережье с юго-востока, а не с севера в обход Главного Кавказского хребта. Закавказье превосходит по гаплотипическому разнообразию Северный Кавказ, который был заселен, по-видимому, лишь на поздних этапах формирования видового ареала. В целом, несмотря на наличие изолированных популяций, значительно дистанцированных гаплотипов здесь не обнаружено, что, вероятно, может рассматриваться как свидетельство молодости ареала полосатой ящерицы в регионе.
Молекулярные операционные таксономические единицы строго соответствуют признаваемым видам рода Lacerta и не согласуются с подвидовой структурой прыткой ящерицы. Выявленные противоречия между принятой в настоящее время внутривидовой систематикой L. agilis и дифференциацией подвидов по COI, указывают на необходимость ее пересмотра с использованием более информативных генетических маркеров. В то же время ДНК-штрихкодирование может быть использовано для видовой диагностики зеленых ящериц Кавказа и сопредельных территорий.
Авторы признательны К.Д. Мильто, В.Ф. Орловой и Б.С. Туниеву за ценные консультации и помощь в работе.
Молекулярно-генетические исследования выполнены при финансовой поддержке гранта РНФ (№ 22-24-00079).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Kornilios P., Thanou E., Lymberakis P. et al. A phylogenomic resolution for the taxonomy of Aegean green lizards // Zool. Scripta. 2019. V. 49. № 1. P. 14–27. https://doi.org/10.1111/zsc.12385
Кукушкин О.В., Ермаков О.А., Иванов А.Ю. и др. Филогеография прыткой ящерицы в Крыму по результатам анализа гена цитохрома b: древний рефугиум на полуострове, поздняя экспансия с севера и первые свидетельства гибридизации подвидов Lacerta agilis tauridica и L. a. exigua (Lacertidae: Sauria) // Тр. Зоол. ин-та РАН. 2020. Т. 324. № 1. С. 56–99. https://doi.org/10.31610/trudyzin/2020.324.1.56
Доронина М.А., Доронин И.В., Луконина С.А. и др. Филогеография и систематика ящериц рода Lacerta (Lacertidae: Sauria) на Кавказе и сопредельных территориях // Вопр. герпетологии: VIII съезд Герпетологического о-ва им. А.М. Никольского при РАН “Современные герпетологические исследования Евразии”. Программа и тезисы докладов. Москва: КМК, 2021. С. 147–149.
Uetz P., Cherikh S., Shea G. et al. A global catalog of primary reptile type specimens // Zootaxa. 2019. V. 4695. № 5. P. 438–450. https://doi.org/10.11646/zootaxa.4695.5.2
Калябина-Хауф С.А. Филогеография и внутривидовая структура широкоареального вида ящериц, Lacerta agilis L. 1758. Дисс. … канд. биол. наук. СПб.: ЗИН РАН, 2003. 176 с.
Калябина-Хауф С.А., Ананьева Н.Б. Филогеография и внутривидовая структура широкоареального вида ящериц Lacerta agilis L., 1758 (Lacertida, Sauria, Reptilia) (опыт использования митохондриального гена цитохрома b // Тр. Зоол. ин-та РАН. Т. 302. СПб.: ЗИН РАН, 2004. 108 с.
Sookhov [Suchow] G.F. Description of a new species of lizards from the environs of Vladicaucasus (Lacerta boemica sp. nov.) // Zbirnyk prats’ Zoolohichnogo muzeyu. 1929. I. 7. P. 117–119.
Andres C., Franke F., Bleidorn C. et al. Phylogenetic analysis of the Lacerta agilis subspecies complex // Syst. and Biodiversity. 2014. V. 12. № 1. P. 43–54. https://doi.org/10.1080/14772000.2013.878000
Песков В.Н., Бровко А.Ю. Изменчивость меристических признаков фолидоза и морфологическая дифференциация Lacerta agilis (Lacertidae, Sauria, Reptilia) на территории Украины // Матер. Першої конференції Українського герпетологічного товариства. Київ, 2005. С. 128–134.
Песков В.Н., Свириденко Е.Ю., Малюк А.Ю., Котенко Т.И. Половой диморфизм и определение пола по меристическим признакам фолидоза у прыткой ящерицы, Lacerta agilis Linnaeus, 1758 (Reptilia, Sauria, Lacertidae) // Науковий вісник Ужгородського університету Серія Біологія. 2010. Вып. 27. С. 140–144.
Песков В.Н., Свириденко Е.Ю., Малюк А.Ю., Котенко Т.И. Изменчивость фолидоза и дифференциация подвидов прыткой ящерицы, Lacerta agilis (Reptilia, Lacertidae), на территории Украины // Вестник зоологии. 2011. Т. 45. № 6. С. 541–554.
Saberi-Pirooz R., Rajabi-Maham H., Ahmadzadeh F. et al. Pleistocene climate fluctuations as the major driver of genetic diversity and distribution patterns of the Caspian green lizard, Lacerta strigata Eichwald, 1831 // Ecol. Evol. 2021. V. 11. № 11. P. 6927–6940. https://doi.org/10.1002/ece3.7543
Доронина М.А., Доронин И.В., Луконина С.А. и др. Филогеография Lacerta media Lantz et Cyrén, 1920 (Lacertidae: Sauria) по результатам анализа митохондриального гена цитохрома b // Генетика. 2022 Т. 58. № 2. С. 177–186. https://doi.org/10.31857/S0016675822020035
Ghane-Ameleh S., Khosravi M., Saberi-Pirooz R. et al. Mid-Pleistocene transition as a trigger for diversification in the Irano-Anatolian region: Evidence revealed by phylogeography and distribution pattern of the eastern three-lined lizard // Global Ecol. and Conservation. 2021. V. 31. P. 1–13. https://doi.org/10.1016/j.gecco.2021.e01839
Мусхелишвили Т.А. Возможные пути проникновения пресмыкающихся на территорию Грузии и некоторые характерные особенности их распространения // Сообщения Академии наук Грузинской ССР. 1970а. Т. 58. № 1. С. 209–212.
Тертышников М.Ф., Бадмаева В.И., Горовая В.И., Иванов В.Б. Материалы о распространении и экологии ящерицы полосатой на Северном Кавказе // Фауна и экология животных Калмыкии и сопредельных районов. Межвузовский сб. науч. трудов. Элиста: Калмыцкий гос. ун-т, 1984. С. 85–92.
Рудик А.М. О находке полосатой ящерицы (Lacerta strigata Eichwald) на Черноморском побережье Кавказа // Тр. Зоол. ин-та АН СССР. Герпетологические исследования на Кавказе. Л.: ЗИН АН СССР, 1986. С. 187–188.
Доронина М.А., Доронин И.В. Анализ распространения зеленых ящериц рода Lacerta на Кавказе и сопредельных территориях // Совр. герпетология: пробл. и пути их решения. Матер. Второй междунар. молодежной конф. герпетологов России и сопред. стран, посвященной 100-летию отделения герпетологии Зоол. ин-та РАН. СПб.: ЗИН РАН, 2019. С. 48.
Murphy R.W., Crawford A.J., Bauer A.M. et al. Cold Code: the global initiative to DNA barcode amphibians and nonavian reptiles // Mol. Ecol. Res. 2013. V. 13. № 2. P. 161–167. https://doi.org/10.1111/1755-0998.12050
Hawlitschek O., Morinière J., Dunz A. et al. Comprehensive DNA barcoding of the herpetofauna of Germany // Mol. Ecol. Resources. 2016. V. 16. № 1. P. 242–253. https://doi.org/10.1111/1755-0998.12416
Drohvalenko M., Mykhailenko A., Rekrotchuk M. et al. Application of DNA barcoding in taxonomy and phylogeny: An individual case of COI partial gene sequencing from seven animal species // Vestnik Zoologii. 2019. V. 53. № 5. P. 375–384. https://doi.org/10.2478/vzoo-2019-0034
Zangl L., Daill D., Schweiger S. et al. A reference DNA barcode library for Austrian amphibians and reptiles // PLoS One. 2020. V. 15. № 3. P. e0229353. https://doi.org/10.1371/journal.pone.0229353
Дунаев Е.А., Соловьева Е.Н., Поярков Н.А. Таксономия, филогения и распространение Phrynocephalus (superspecies guttatus) (Reptilia: Agamidae) // Совр. герпетология. 2020. Т. 20. Вып. 1/2. С. 16–34. https://doi.org/10.18500/1814-6090-2020-20-1-2-16-34
Даревский И.С., Щербак Н.Н., Петерс Г. и др. Систематика и внутривидовая структура // Прыткая ящерица. Монографическое описание вида / Отв. ред.: Яблоков А.В. М.: Наука, 1976. С. 53–95.
Мусхелишвили Т.А. Пресмыкающиеся Восточной Грузии. Тбилиси: Мецниереба, 1970. 235 с.
Tuniyev S.B., Tuniyev B.S. Intraspecific variation of the Sand lizard (Lacerta agilis) from the Western Caucasus and description of a new subspecies Lacerta agilis mzymtensis ssp. nov. (Reptilia: Sauria) // Russ. J. Herpetology. 2008. V. 15. № 1. P. 55–66. https://doi.org/10.30906/1026-2296-2008-15-1-55-66
Aljanabi S.M., Martinez I. Universal and rapid salt-extraction of high genomic DNA for PCR-based techniques // Nucl. Ac. Res. 1997. V. 25. № 22. P. 4692–4693. https://doi.org/10.1093/nar/25.22.4692
Lissovsky A.A., Obolenskaya E.V., Abramson N.I. et al. Geographic variation of Microtus middendorffii (Cricetidae, Arvicolinae, Rodentia) sensu lato studied by craniometrical and mitochondrial features // Russ. J. Theriol. 2010. V. 9. № 2. P. 71–81. https://doi.org/10.15298/rusjtheriol.09.2.03
Larsson A. AliView: A fast and lightweight alignment viewer and editor for large data sets // Bioinformatics. 2014. V. 30. I. 22. P. 3276–3278. https://doi.org/10.1093/bioinformatics/btu531
Huelsenbeck J., Ronquist F. MRBAYES: Bayesian inference of phylogenetic trees // Bioinformatics. 2001. V. 17. № 8. P. 754–755. https://doi.org/10.1093/bioinformatics/17.8.754
Ronquist F., Huelsenbeck J.Ph. MrBayes 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Altekar G., Dwarkadas S., Huelsenbeck J.P., Ronquist F. Parallel metropolis coupled Markov chain Monte Carlo for Bayesian phylogenetic inference // Bioinformatics. 2004. V. 20. № 3. P. 407–415. https://doi.org/10.1093/bioinformatics/btg427
Kumar S., Stecher G., Li M. et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. and Evol. 2018. V. 35. № 6. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Felsenstein J. Inferring Phylogenies. Sunderland: Sinauer Associates, 2004. 664 p.
Nylander J.A.A. MrModeltest v2. Program distributed by the author. Evolutionary Biology Centre, Uppsala University. 2004. https://github.com/nylander/MrModeltest2
Leigh J.W., Bryant D. PopART: Full-feature software for haplotype network construction // Meth. in Ecol. and Evol. 2015. V. 6. № 9. P. 1110–1116. https://doi.org/10.1111/2041-210X.12410
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
Sarkar I.N., Egan M.G., Coruzzi G. et al. Automated simultaneous analysis phylogenetics (ASAP): An enabling tool for phlyogenomics // BMC Bioinformatics. 2008. V. 9. № 103. P. 1–5. https://doi.org/10.1186/1471-2105-9-103
Puillandre N., Lambert A., Brouillet S., Achaz G. ABGD, Automatic Barcode Gap Discovery for primary species delimitation // Mol. Ecol. 2012. V. 21. № 8. P. 1864–1877. https://doi.org/10.1111/j.1365-294X.2011.05239.x
Абрамсон Н.И. Филогеография: итоги, проблемы, перспективы // Вестник ВОГиС. 2007. Т. 11. № 2. С. 307–331.
Fu Y.-X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection // Genetics. 1997. V. 147. P. 915–925. https://doi.org/10.1093/genetics/147.2.915
Peters G., Muskheliswili T.A. Lacerta agilis ioriensis – eine neue Subspecies der kaukasischen Zauneidechsen // Zool. Jb. Syst. 1968. V. 95. P. 213–228.
Milto K.D. On the Systematic Position of the Sand Lizard (Lacerta agilis Linnaeus, 1758) in the Western Ukraine // Russ. J. Herpetology. 1996. V. 3. № 2. P. 199–200.
Дополнительные материалы
- скачать ESM.docx
- Приложение 1.
Таблица 1. Коллекционные экземпляры зеленых ящериц рода Lacerta, использованные при молекулярно-генетическом анализе


