Генетика, 2023, T. 59, № 4, стр. 403-416
Генетически изолированная популяция дрожжей Saccharomyces bayanus в Новой Зеландии и Австралии
А. Н. Боровкова 1, 2, Г. И. Наумов 1, А. В. Шнырева 2, Е. С. Наумова 1, *
1 Национальный исследовательский центр “Курчатовский институт”, Курчатовский комплекс генетических исследований (ГосНИИгенетика)
123098 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
119234 Москва, Россия
* E-mail: lena_naumova@yahoo.com
Поступила в редакцию 11.07.2022
После доработки 05.09.2022
Принята к публикации 20.09.2022
- EDN: ATSSFD
- DOI: 10.31857/S0016675823040021
Аннотация
С помощью методов молекулярной и классической генетики изучено генетическое родство дрожжей комплекса Saccharomyces bayanus и обнаружена дивергентная популяция этих дрожжей в Новой Зеландии и Австралии. Комплекс S. bayanus включает четыре генетические популяции: S. bayanus var. bayanus, S. bayanus var. uvarum, S. eubayanus и новозеландская. Штаммы новозеландской популяции существенно отличаются по нуклеотидным последовательностям ядерных (FSY1, HIS3, MET2) и митохондриальных (FUN14, COX2) генов и образуют полустерильные гибриды с остальными популяциями: 6.2–23.3%. Между S. bayanus var. bayanus, S. bayanus var. uvarum, S. eubayanus и новозеландской популяцией нет полной межвидовой постзиготической изоляции: все гибриды имели регулярное мейотическое расщепление контрольных ауксотрофных маркеров. Согласно полученным результатам указанные генетические популяции относятся к одному биологическому виду, обладая дивергенцией геномов на уровне таксономических разновидностей.
Генофонд культурных дрожжей-сахаромицетов представлен видами S. cerevisiae, S. bayanus (син. S. uvarum) и их гибридом S. pastorianus (син. S. carlsbergensis) [1, 2]. Помимо двух указанных род Saccharomyces включает еще шесть видов: S. arboricola, S. cariocanus, S. jurei, S. kudriavzevii, S. mikatae и S. paradoxus [2–6]. Благодаря общей системе типов спаривания восемь биологических видов рода Saccharomyces могут скрещиваться во всех комбинациях, но за счет постзиготической изоляции образующиеся гибриды стерильны и имеют нежизнеспособные аскоспоры. Естественные межвидовые гибриды S. cerevisiae × S. bayanus, S. cerevisiae × S. kudriavzevii и S. cerevisiae × S. bayanus × S. kudriavzevii обнаружены среди коммерческих винных, пекарских и пивных дрожжей, используемых во Франции, Испании, Австрии, Швейцарии и Австралии [7–11].
Растущий интерес к изучению дрожжей S. bayanus связан не только с его возможной ролью в качестве одного из родительских геномов пивных дрожжей низового брожения S. pastorianus, но и с его значением как нового генофонда культурных дрожжей Saccharomyces. С помощью различных молекулярных методов была показана гетерогенность вида S. bayanus, включающего две группы штаммов, которые различаются по рибосомным последовательностям (ITS1 и IGS2) и молекулярным кариотипам: “bayanus” и “uvarum” [7, 12, 13]. Гибридологическим анализом установлена частичная генетическая изоляция указанных групп: их гибриды полустерильны (15–34% выживаемости аскоспор), имея регулярную мейотическую сегрегацию ауксотрофных контрольных маркеров [7, 14]. В то же время штаммы обеих групп образуют стерильные гибриды с дрожжами S. cerevisiae. На основании генетических и молекулярных данных были предложены две разновидности вида S. bayanus: var. bayanus и var. uvarum, принятые в современных монографиях по систематике дрожжей [2, 7, 14].
Специфической экологической нишей S. bayanus var. uvarum является виноделие и виноградарство при пониженных температурах: эти дрожжи ассоциированы с производством белых, сладких и игристых вин, а также сидра [15–23]. Природные изоляты S. bayanus var. uvarum обнаружены в Испании, Словакии, Венгрии, Португалии, на Дальнем Востоке России, в США, Аргентине, Чили, Австралии и Новой Зеландии [1, 20, 24]. Дрожжи S. bayanus var. bayanus представлены в основном штаммами, загрязняющими пивоварение, включая типовую культуру CBS 380 [7, 13]. Некоторые авторы возводят указанные разновидности в ранг отдельных видов: S. bayanus и S. uvarum [25–27]. В отличие от S. bayanus var. uvarum дрожжи S. bayanus var. bayanus обладают субтеломерными последовательностями S. cerevisiae [7, 28]. На этом основании было высказано предположение о гибридной природе этих дрожжей и было предложено закрыть вид S. bayanus как “неправильный” (not proper) и восстановить не содержащий чужеродных последовательностей вид S. uvarum как таксономически “чистый” [26, 28, 29]. В качестве типовой культуры был выбран штамм CBS 7001 (MCYC 623), у которого определена полная нуклеотидная последовательность генома [30]. С помощью ПДРФ-анализа 48 генов и частичного секвенирования 16 из них Rainieri et al. [27] подтвердили гомогенность дрожжей S. bayanus var. uvarum. Среди дрожжей S. bayanus var. bayanus авторами также была обнаружена “чистая” линия: штамм NBRC 1948, выделенный из испорченного бочкового пива в Европе. Этот штамм был предложен в качестве новой типовой культуры вида S. bayanus.
Более детальное молекулярное изучение выявило мозаичность генома штамма NBRC 1948, содержащего последовательности uvarum и второго вида S. baynus-типа, условно названного авторами S. lagerae, а также интрогрессивные субтеломерные фрагменты S. cerevisiae [31]. Родственные дрожжи S. eubayanus были описаны на изолятах из Аргентины, а позже обнаружены в Китае, США, Канаде, Австралии и Новой Зеландии [24, 32–35]. В Европе геном S. eubayanus обнаружен только у гибридных дрожжей S. pastorianus [36, 37]. Гибридологическим анализом показано, что гибриды S. bayanus var. bayanus × S. eubayanus обладают пониженной выживаемостью аскоспор: 55–62% [38]. Гибриды S. bayanus var. uvarum × S. eubayanus практически стерильны: 11% выживаемости аскоспор. В то же время во всех скрещиваниях наблюдалась рекомбинация родительских маркеров. Полногеномное секвенирование нескольких штаммов S. eubayanus обнаружило их большое сходство с холодоустойчивым родителем пивных дрожжей S. pastorianus [36, 37, 39]. Выявлено большое сходство генома штамма NBRC 1948 с типовой культурой S. eubayanus CBS 12357 и холодоустойчивым геномом пивного коммерческого штамма Weihenstephan 34/70 [13, 24]. Еще больше усложнило понимание таксономического статуса дрожжей S. bayanus обнаружение новозеландской популяции, штаммы которой по ряду молекулярных маркеров отличаются от S. bayanus var. bayanus, S. bayanus var. uvarum и S. eubayanus [20].
Цель исследования – изучение генетического родства дрожжей комплекса S. bayanus, включая географически изолированную популяцию из Новой Зеландии и Австралии.
МАТЕРИАЛЫ И МЕТОДЫ
Среды и штаммы
Изучаемые штаммы дрожжей S. bayanus и их происхождение приведены в табл. 1. Дрожжи культивировали при 28°С на полной агаризованной среде YPD следующего состава (г/л): глюкоза – 20, пептон – 20, дрожжевой экстракт – 10, агар – 20.
Таблица 1.
Происхождение изученных штаммов Saccharomyces
| Штамм | Источник и место выделения | Регистрационный номер в GenBank | ||||
|---|---|---|---|---|---|---|
| MET2 | HIS3 | FSY1 | FUN14 | COX2 | ||
| S. cerevisiae | ||||||
| YNN295 | Генетическая линия | – | – | – | – | – |
| S288C | Генетическая линия | NM_001183115 | NM_001183621 | – | NM_001178153 | NC_001224 |
| ВКМ Y–502 | Виноград, Дальний Восток, Россия | – | – | – | – | – |
| S. bayanus var. bayanus | ||||||
| CBS 380 | Пиво | AJ627635.1 | JF786624.1 | GCA_013180675.1 | GCA_013180675.1 | AF442211 |
| CBS 425 | Яблочный сок, Швейцария | OP355544 | JF786626 | JF786693 | JF786618 | OP355555 |
| CBS 424 | Грушевый сок, Швейцария | FR774000 | OP355535 | OP355524 | OP355516 | OP355554 |
| NBRC 1948 | Испорченное пиво, Европа | JF786641.1 | GCA_013180125.1 | GCA_013180125.1 | GCA_013180125.1 | EF639726 |
| CBS 378 | Пиво, Европа | JF786659 | OP355533 | JF786695 | JF786658 | OP355553 |
| S. bayanus var. uvarum | ||||||
| CBS 395 | Сок черной смородины Ribes nigrum, Нидерланды | AJ627638 | OP355534 | HE858456 | KF892125 | KX657742 |
| CBS 7001 | Ручейник Mesophylax adopersus, Испания | GCA_019953615.1 | GCA_019953615.1 | GCA_019953615.1 | GCA_019953615.1 | KF530350 |
| ВКМ Y–1146 | Виноград, Мичуринск, Россия | GCA_013180055.1 | GCA_013180055.1 | GCA_013180055.1 | GCA_013180055.1 | GCA_013180055.1 |
| CBS 377 | Грушевое вино, Германия | GCA_013265775.1 | GCA_013265775.1 | GCA_013265775.1 | GCA_013265775.1 | GCA_013265775.1 |
| M488 | Виноград, Молдавия | GCA_013180195.1 | GCA_013180195.1 | GCA_013180195.1 | GCA_013180195.1 | GCA_013180195.1 |
| ВКМ Y–361 | Токайское вино, Словакия | GCA_013180345.1 | GCA_013180345.1 | GCA_013180345.1 | GCA_013180345.1 | GCA_013180345.1 |
| ВКМ Y–508 | Токайское вино, Словакия | GCA_013180255.1 | GCA_013180255.1 | GCA_013180255.1 | GCA_013180255.1 | GCA_013180255.1 |
| PJS2.95 | Бродящая мезга, Сансер, Франция | GCA_013179965.1 | GCA_013179965.1 | GCA_013179965.1 | GCA_013179965.1 | GCA_013179965.1 |
| 148.01 | Экссудат вяза Ulmus pumila, Благовещенск, Россия | GCA_013265745.1 | GCA_013265745.1 | GCA_013265745.1 | GCA_013265745.1 | GCA_013265745.1 |
| NCAIM Y.00676 | Алкогольный напиток, Венгрия | GCA_013180065.1 | GCA_013180065.1 | GCA_013180065.1 | GCA_013180065.1 | GCA_013180065.1 |
| PYCC 7082 | Cyttaria sp. на Nothofagus dombeyi, Патагония, Аргентина | OP355536 | OP355525 | OP355517 | OP355509 | OP355545 |
| PYCC 7083 | Кора Nothofagus pumillio, Патагония, Аргентина | OP355537 | OP355526 | OP355518 | OP355510 | OP355546 |
| PYCC 6330 | Плодовое тело Cyttaria hariotii, Патагония, Аргентина | JF786645 | JF786630 | JF786699 | JF786622 | OP355552 |
| UWO(PS) 99-808 | Сокотечение бука Nothofagus sp., Патагония, Аргентина | OP355543 | OP355532 | JF786703 | – | OP355551 |
| Новозеландская популяция | ||||||
| PYCC 6864 | Cyttaria gunni на Nothofagus menziesii, Новая Зеландия | OP355538 | OP355527 | OP355519 | OP355511 | OP355547 |
| PYCC 6865 | Кора Nothofagus cunninghamii, Тасмания, Австралия | OP355539 | OP355528 | OP355520 | OP355512 | – |
| PYCC 6867 | Кора Nothofagus solandri var. solandri, Новая Зеландия | OP355540 | OP355529 | OP355521 | OP355513 | OP355548 |
| PYCC 6868 | Кора Nothofagus solandri var. solandri, Новая Зеландия | OP355541 | OP355530 | OP355522 | OP355514 | OP355549 |
| PYCC 6869 | Кора Nothofagus solandri var. solandri, Новая Зеландия | OP355542 | OP355531 | OP355523 | OP355515 | OP355550 |
| S. eubayanus | ||||||
| СBS 12357 | Cyttaria hariotti, Аргентина | GCA_003327605.1 | GCA_003327605.1 | GCA_003327605.1 | GCA_003327605.1 | CP030961 |
| PYCC 7084 | Cyttaria harioti на Nothofagus dombeyi, Аргентина | KF530488 | KF530444 | KF530380 | KF530401 | KF530338 |
| PYCC 7085 | Кора Nothofagus antarctica, Аргентина | KF530492 | KF530448 | KF530384 | KF530405 | KF530341 |
| PYCC 7087 | Почва под Nothofagus pumilio, Аргентина | KF530495 | KF530451 | KF530387 | KF530408 | – |
| PYCC 7088 | Почва под Nothofagus pumilio, Аргентина | KF530496 | KF530452 | KF530388 | KF530409 | KF530345 |
| PYCC 7089 | Почва под Nothofagus oblique, Аргентина | KF530497 | KF530453 | KF530389 | KF530410 | KF530346 |
| yHKS210 | Кора Fagus grandifolia, Висконсин, США | KF530498 | KF530454 | KF530390 | KF530411 | KF530347 |
| yHKS211 | Кора Fagus grandifolia, Висконсин, США | KF530499 | KF530455 | KF530391 | KF530412 | KF530348 |
| yHKS212 | Кора Acer saccharum, Висконсин, США | KF530500 | KF530456 | KF530392 | KF530413 | KF530349 |
Примечание. ВКМ – Всероссийская коллекция микроорганизмов (Пущино, Россия); М – Коллекция микроорганизмов виноделия “Магарач” (КМВ “Магарач”), ФГБУН ВННИИВиВ “Магарач” РАН (Ялта, Россия); CBS – The Westerdijk Fungal Biodiversity Institute (Утрехт, Нидерланды); NBRC/IFO – National Institute of Technology and Evaluation (Tокио, Япония); NCAIM – National Collection of Agricultural and Industrial Microorganisms (Будапешт, Венгрия); PYCC – Portuguese Yeast Culture Collection (Лиссабон, Португалия); UWO (PS) – Culture Collection of the Department of Biology, University of Western Ontario (Онтарио, Канада); PJS2.95 – штамм из коллекции Institut des Sciences De la Vigne et du Vin (ISVV) (Вильнав-д’Орнон, Франция). Остальные штаммы из коллекции лаборатории молекулярной генетики дрожжей “ГосНИИгенетика”, НИЦ “Курчатовский институт” (Москва, Россия). Соответствия некоторых номеров коллекций: PYCC 7082 = CRUB 1586; PYCC 7083 = CRUB 1778.
Полимеразная цепная реакция (ПЦР)
Полимеразную цепную реакцию осуществляли на ДНК-амплификаторе “Bio-Rad” (США). Дрожжевую ДНК выделяли согласно Lõoke et al. [40]. Праймеры, использованные в работе, приведены в табл. 2. ПЦР проводили в 30 мкл буфера, содержащего 2.5 мМ MgCl2, 0.1 мМ каждого dNTP, 50 пмоль каждого праймера, 2.5 единицы Taq-полимеразы (Синтол, Россия) и 20–200 нг ДНК. Для амплификации генов MET2, HIS3, FSY1 и FUN14 использовали следующую программу: начальная денатурация ДНК при 94°C в течение 3 мин; затем 30 циклов в режиме: денатурация в режиме 94°С – 30 с, отжиг праймеров при 56°С – 30 с, синтез ДНК при 72°С – 60 с; конечная достройка при 72°С – 10 мин. Амплификацию межгенного спейсера IGS2 проводили в следующем режиме: начальная денатурация ДНК при 94°C в течение 4 мин; затем 25 циклов: денатурация при 94°С – 60 с, отжиг праймеров при 48°С – 30 с, синтез ДНК при 72°С – 60 с; конечная достройка при 72°С – 10 мин. Митохондриальный ген COX2 амплифицировали по следующей программе: начальная денатурация ДНК при 94°C в течение 5 мин; затем 45 циклов в режиме: денатурация при 94°С – 40 с, отжиг праймеров при 45°С – 35 с, синтез ДНК при 72°С – 35 с; конечная достройка при 72°С – 10 мин. Продукты амплификации подвергали электрофорезу в 1%-ном агарозном геле при 60–65 В в 0.5× TBE буфере (45 мМ трис, 10 мМ ЭДТА, 45 мМ борная кислота, рН 8.0) в течение 1–1.5 ч. Гель окрашивали бромистым этидием, промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете на трансиллюминаторе Vilber Lourmat (Франция). В качестве маркера молекулярных масс использовали 1kb DNA Ladder (Fermentas, Литва).
Таблица 2.
| Ген или район амплификации | Последовательность праймера (5'–3') | Размер амплифицированного фрагмента, пн |
|---|---|---|
| IGS2 рДНК | NTS2: AACGGTGCTTTCTGGTAG | 1300 |
| ETS1: TGTCTTCAACTGCTTT | ||
| FSY1 | FSY11: GGATCYTCRACAAGCGTTTCTC | 1247 |
| FSY12: AAGGCAAACAYGTAAAGCAAAG | ||
| MET2 | MET21: CGAAAACGCTCCAAGAGCTGG | 415 |
| MET22: GACCACGATATGCACCAGGCAG | ||
| HIS3 | HIS31: ATGTCAGAGCAAAAGGCCCTA | 579 |
| HIS32: CATGAGAACACCCTTTGTGGA | ||
| COX2 | COX21: GGTATTTTAGAATTACATGA | 564 |
| COX22: ATTTATTGTTCRTTTAATCA | ||
| FUN14 | FUN14D: TATTAAGCTGGGAGTGCCCTT | 429 |
| FUN14R: TTATTGGCGTTTAGGCTTGA |
Определение нуклеотидных последовательностей и филогенетический анализ
Амплифицированные фрагменты элюировали из геля с помощью набора GeneJET Gel Extraction Kit (Thermo Scientific, США), согласно протоколу фирмы-изготовителя. Нуклеотидные последовательности генов определяли по двум цепям с помощью прямого секвенирования по методу Сенгера на автоматическом секвенаторе Applied Biosystems 3730 (США). Поиск гомологии с известными нуклеотидными последовательностями проводили с помощью программы BLAST в базе данных GenBank (http://www.ncbi.nlm.nih.gov/genbank/). Множественные выравнивания изученных нуклеотидных и аминокислотных последовательностей проводили с помощью программы BioEdit (http://www.mbio.ncsu.edu/BioEdit/bioedit.html). Филогенетические деревья строили методом объединения соседей (Neighbor-Joining) в программе MEGA7 [41].
ПДРФ-анализ ПЦР-амплифицированных IGS2-участков рДНК
Рестрикционный анализ осуществляли с помощью эндонуклеазы AluI (Fermentas) в течение 12 ч при 37°С. Разделение фрагментов рестрикции проводили в 2.5%-ном агарозном геле при 50–60 В в 0.5× ТВЕ-буфере в течение 2.5–3 ч. Гель окрашивали бромистым этидием (0.5 мкг/мл) в течение 2–3 ч, затем промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете на трансиллюминаторе Vilber Lourmat (Франция). В качестве маркера молекулярных масс использовали 100bp DNA Ladder (Fermentas).
Молекулярное кариотипирование и Саузерн-гибридизация
Выделение хромосомной ДНК проводили как описано ранее [15]. Для разделения хромосомной ДНК использовали аппарат CHEF-DR III (Bio-Rad, США). Образцы помещали в щели 1%-ного агарозного геля. Для разделения хромосомных полос использовали два режима кариотипирования: 1) 200 В, в течение 15 ч при времени переключения полей 60 с и 9 ч при времени переключения полей 90 с; 2) 200 В, в течение 24 ч при времени переключения полей 15–40 с. В качестве буфера использовали 0.5× TBE, охлажденный до 14°C. Штамм Saccharomyces cerevisiae YNN 295 (Bio-Rad), имеющий известный порядок и размеры хромосом, служил кариотипическим стандартом. После электрофореза гель окрашивали бромистым этидием в течение 2–3 ч, затем промывали в дистиллированной воде в течение 2 ч и фотографировали в УФ-свете. Использовали 1%-ную агарозу.
Хромосомную ДНК переносили на нитроцеллюлозную мембрану, используя аппарат Vacuum blotter (Bio-Rad). ДНК фиксировали на мембране путем отжига при 80°C в течение 2 ч. В качестве зонда использовали ПЦР-амплифицированный ген ACT1 дрожжей S. cerevisiae S288C. Метку вводили нерадиоактивным методом с использованием dUTP, меченного дигоксигенином (dig-II-dUTP) из набора DIG High Prime DNA Labeling and Detection Starter Kit I (Roche, Швейцария), согласно инструкции производителя. Гибридизацию и проявление гибридизационных полос также проводили по инструкции указанной фирмы.
Гибридологический анализ
Дрожжи скрещивали на полной агаризованной среде YPD; спорообразование индуцировали на ацетатной среде (г/л): CH3COONa – 10, KCl – 5, агар – 20. Спонтанные ауксотрофные мутации lys и ura отбирали на селективных средах, содержащих соответственно DL-аминоадипиновую и 5'-фтороротовую кислоты [42, 43]. Гибридизацию проводили методом “спора на спору” с использованием микроманипулятора или массовым скрещиванием спор на полной среде с последующим отбором гибридов на минимальных селективных средах. Состав минимальной среды (г/л): дрожжевая азотная основа без аминокислот (фирмы “Difco”, США) – 6.7, глюкоза – 20, агар – 20. Аскоспоры изолировали с помощью микроманипулятора, предварительно разрушив оболочки асков ферментативным препаратом из желудка виноградных улиток Helix pomatia.
РЕЗУЛЬТАТЫ
С помощью различных молекулярных методов и гибридологиического анализа мы изучили генетическое родство 33 штаммов комплекса S. bayanus. Штаммы выделены из ферментационных процессов и различных природных источников в разных регионах мира: Россия, Нидерланды, Швейцария, Испания, Германия, Франция, Словакия, Венгрия, Молдавия, Аргентина, США, Австралия и Новая Зеландия (табл. 1).
ПДРФ-анализ ПЦР-амплифицированных IGS2-участков рДНК
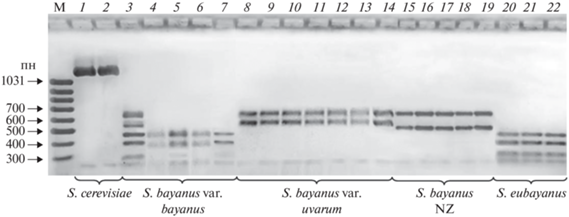
У изученных штаммов S. bayanus были проведены амплификация IGS2-участка рДНК и последующий ПДРФ-анализ с помощью рестриктазы AluI. На рис. 1 представлены ПДРФ-паттерны некоторых штаммов. С помощью этого молекулярного маркера можно четко дифференцировать виды S. cerevisiae и S. bayanus, а также внутривидовые популяции последнего (рис. 1, дорожки 1, 2 и 3–22 соответственно). По сходству AluI-профилей изученные штаммы S. bayanus разделились на четыре группы. Идентичные паттерны имели штаммы S. bayanus var. bayanus (CBS 378, CBS 424, CBS 425, NBRC 1948) и S. eubayanus (дорожки 4–7 и 20–22 соответственно). Вторую группу составили штаммы S. bayanus var. uvarum, имеющие три AluI-фрагмента размером 610, 520 и 170 пн (дорожки 8–14). В третью группу вошли пять штаммов S. bayanus, изолированных в Австралии (Тасмания) и Новой Зеландии (далее новозеландская популяция, NZ), у которых средний фрагмент был несколько меньшего размера: 500 пн (дорожки 15–19). Четвертая группа представлена типовой культурой S. bayanus var. bayanus CBS 380, в AluI-профиле которой объединены фрагменты, характерные для S. bayanus var. bayanus и S. bayanus var. uvarum (дорожка 3).
Рис. 1.
ПДРФ-анализ амплифицированных фрагментов межгенного спейсера IGS2 дрожжей комплекса Saccharomyces bayanus с помощью эндонуклеазы AluI. S. cerevisiae (контроль): 1 – S288C, 2 – ВКМ Y–502; S. bayanus var. bayanus: 3 – CBS 380, 4 – CBS 378, 5 – CBS 424, 6 – CBS 425, 7 – NBRC 1948; S. bayanus var. uvarum: 8 – ВКМ Y–1146, 9 – M488, 10 – NCAIM Y.00677, 11 – PJS 2.95, 12 – 148.01, 13 – UWO(PS) 99-808, 14 – M300; Новозеландская (NZ) популяция дрожжей S. bayanus: 15 – PYCC 6864, 16 – PYCC 6865, 17 – PYCC 6867, 18 – PYCC 6868, 19 – PYCC 6869; S. eubayanus: 20 – CBS 12357, 21 – PYCC 7085, 22 – yHKS 210. М – маркер молекулярных масс. 100 bp DNA Ladder (Fermentas, Литва).
Мультигенный филогенетический анализ
Для установления филогенетического родства изученных штаммов мы провели сравнительный анализ нуклеотидных последовательностей трех ядерных (FSY1, HIS3, MET2) и двух митохондриальных (FUN14, COX2) генов.
На филогенетическом дереве, построенном по нуклеотидным последовательностям генов FSY1, HIS3 и MET2, со 100%-ной статистической поддержкой выделяются два кластера (рис. 2,а). Первый включает два подкластера: S. bayanus var. uvarum и новозеландские штаммы S. bayanus, нуклеотидные последовательности которых отличаются 17–56 заменами. Наибольшие различия отмечены по последовательностям гена FSY1. Внутри каждого подкластера штаммы, как правило, имели идентичные последовательности или различались одной–восьмью заменами. К первому кластеру примыкает S. bayanus var. bayanus CBS 425, выделенный из яблочного сока в Швейцарии. Второй кластер также разделен на два подкластера: S. eubayanus и S. bayanus var. bayanus.
Рис. 2.
Филогенетические деревья, построенные по нуклеотидным последовательностям ядерных (FSY1, HIS3, MET2) (а) и митохондриальных (FUN14, COX2) (б) генов дрожжей комплекса Saccharomyces bayanus. Приводятся значения бутстрепа >70%. Шкала соответствует пяти нуклеотидным заменам на 1000 нуклеотидных позиций. NZ – новозеландская популяция дрожжей S. bayanus.
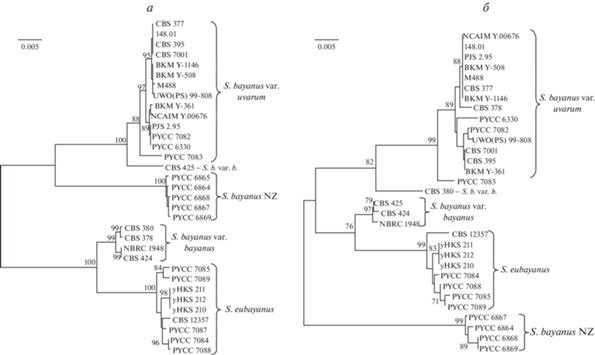
По последовательностям митохондриальных генов FUN14 и COX2 штаммы S. bayanus var. bayanus попали в два разных кластера: три штамма (CBS 424, CBS 425, NBRC 1948) вошли в один кластер с дрожжами S. eubayanus, а два (CBS 378 и CBS 380) – с S. bayanus var. uvarum (рис. 2,б). Третий кластер сформировали штаммы новозеландской популяции.
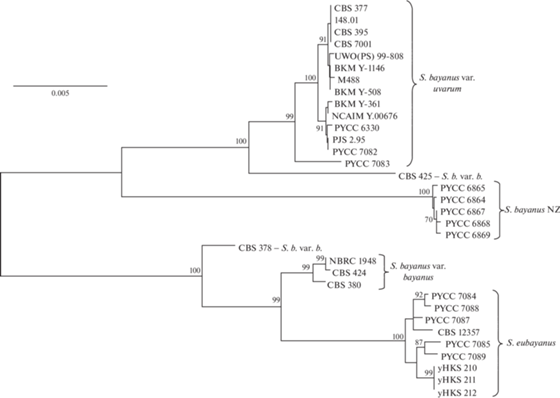
На рис. 3 представлено филогенетическое дерево, построенное по нуклеотидным последовательностям ядерных и митохондриальных генов. Следует отметить, что штаммы новозеландской популяции на всех трех филогенетических деревьях формируют отдельный кластер со статистической поддержкой 99–100%. Эти штаммы значительно отличаются от S. bayanus var. uvarum по нуклеотидным последовательностям всех пяти проанализированных генов: 55–56 замен (FSY1), 16–18 (MET2), 24–26 (HIS3), 21 замена (FUN14) и 43–46 (COX2). Различий с дрожжами S. eubayanus и S. bayanus var. bayanus было значительно больше.
Рис. 3.
Филогенетический анализ ядерных (FSY1, HIS3, MET2) и митохондриальных (FUN14, COX2) генов дрожжей комплекса Saccharomyces bayanus. Приводятся значения бутстрепа >70%. Шкала соответствует пяти нуклеотидным заменам на 1000 нуклеотидных позиций. NZ – новозеландская популяция дрожжей S. bayanus.
Молекулярное кариотипирование и Саузерн-гибридизация
Виды S. cerevisiae и S. bayanus имеют неколлинеарные кариотипы. В геноме последних дрожжей имеется три реципрокные транслокации, затрагивающие хромосомы XV/VIII, IV/II и X/VI [7, 44]. Последняя транслокация характерна для дрожжей S. bayanus var. uvarum и отсутствует у штаммов S. bayanus var. bayanus и S. eubayanus [7, 39]. Следует отметить, что молекулярное кариотипирование штаммов S. bayanus из новозеландской популяции ранее не проводилось.
Мы сравнили молекулярные кариотипы 33 изученных штаммов. Кариотипы некоторых из них представлены на рис. 4,а. Идентификацию отдельных хромосомных полос проводили по кариотипу стандартного штамма S. cerevisiae YNN 295, имеющего известные размеры и порядок хромосом (рис. 4,а, дорожка 1). Новозеландские штаммы имеют сходные кариотипические профили, незначительный полиморфизм размеров отмечен только для хромосомных полос размером 2200–770 тпн (дорожки 12–15). Эти штаммы имеют в своем кариотипе три хромосомные полосы размером 245–370 тпн, вместо двух у S. bayanus var. uvarum (дорожки 10 и 11). Молекулярные кариотипы S. eubayanus также характеризовались наличием хромосомы VI размером около 290 тпн (рис. 4,а, дорожки 7–9). Три штамма S. bayanus var. bayanus (CBS 380, CBS 424 и CBS 425) характеризовались тремя хромосомными полосами размером 245–370 тпн, тогда как у CBS 378 и NBRC 1948 имеется соответственно две и одна хромосомные полосы (дорожки 2–4, 5 и 6). Согласно интенсивности свечения указанные полосы штаммов CBS 378 и NBRC 1948, по-видимому, содержат несколько хромосом. Действительно, с помощью одноступенчатого режима кариотипирования удалось разделить на две хромосомы нижнюю полосу штамма NBRC 1948 и хромосомную полосу размером около 370 тпн у штамма CBS 378 (рисунок не приводится).
Рис. 4.
Пульс-электрофорез (а) и Саузерн-гибридизация (б) хромосомной ДНК дрожжей комплекса Saccharomyces bayanus с зондом ACT1 дрожжей S. cerevisiae. S. bayanus var. bayanus: 2 – CBS 380, 3 – CBS 424, 4 – CBS 425, 5 – CBS 378, 6 – NBRC 1948; S. eubayanus: 7 – CBS 12357, 8 – yHKS210, 9 – PYCC 7086; S. bayanus var. uvarum: 10 – CBS 7001, 11 – CBS 395; новозеландская популяция S. bayanus: 12 – PYCC 6864, 13 – PYCC 6867, 14 – PYCC 6868, 15 – PYCC 6869. Нумерация и размеры хромосом приводятся согласно стандартному штамму S. cerevisiae YNN295 (дорожка 1).
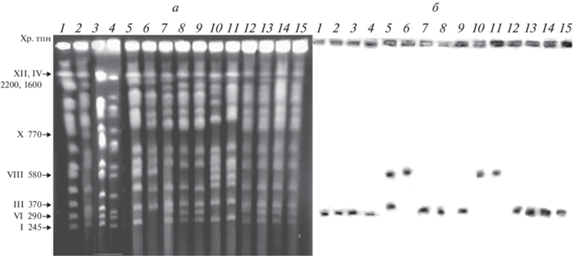
Хромосомные ДНК изученных штаммов были перенесены на нитроцеллюлозную мембрану для последующей Саузерн-гибридизации с зондом ACT1 (хромосома VI) дрожжей S. cerevisiae (рис. 4,б). У всех изученных штаммов S. eubayanus, новозеландской популяции и трех штаммов S. bayanus var. bayanus (CBS 380, CBS 424 и CBS 425) зонд ACT1 гибридизовался к хромосомной полосе размером около 290 тпн, соответствующей хромосоме VI кариотипического стандарта S. cerevisiae YNN 295 (рис. 2,б, дорожка 1). У штамма S. bayanus var. bayanus CBS 378 выявлено два гибридизационных сигнала: var. bayanus-типа и var. uvarum-типа (рис. 4,б, дорожка 5). У NBRC 1948 и всех изученных штаммов S. bayanus var. uvarum обнаружен только один гибридизационный сигнал в районе хромосомы размером около 580 тпн (рис. 4,б, дорожки 6, 10 и 11).
Таким образом, реципрокная транслокация между хромосомами VI и X характерна только для S. bayanus var. uvarum и отсутствует у штаммов новозеландской популяции и S. eubayanus. Среди дрожжей S. bayanus var. bayanus встречаются штаммы обоих типов.
Для определения генетического родства новозеландских и остальных популяций комплекса S. bayanus мы провели гибридологический анализ.
Гибридологический анализ
Были созданы гомозиготные моноспоровые линии штаммов PYCC 6867, PYCC 6868, PYCC 6869 (Новая Зеландия) и NBRC 1948 (S. bayanus var. bayanus) c высокой выживаемостью аскоспор: 83.3–91.7%. Несколько пониженную выживаемость аскоспор имела только моноспоровая культура новозеландского штамма PYCC 6868: 53.6%. Моноспоровые культуры были маркированы ауксотрофными мутациями lys и ura. В скрещиваниях использовали ранее полученные ауксотрофные мутанты штаммов CBS 424, CBS 7001, NCAIM Y.00677, UWO(PS) 99-808 и CBS 12357 [7, 38, 45].
Все полученные гибриды спорулировали и были пригодны для тетрадного анализа. Результаты гибридологического анализа представлены в табл. 3. Внутрипопуляционные гибриды PYCC 6867 × PYCC 6869 характеризовались 68.1%-ной выживаемостью аскоспор и регулярным мейотическим расщеплением контрольных ауксотрофных маркеров. С другой стороны, межпопуляционные гибриды новозеландских штаммов с S. bayanus var. bayanus (CBS 424, NBRC 1948), S. bayanus var. uvarum (CBS 7001, NCAIM Y.00677, UWO(PS) 99-808) и S. eubayanus (CBS 12357) обладали низкой выживаемостью аскоспор: 6.2–23.3%. Несмотря на полустерильность межпопуляционных гибридов, во всех гибридных комбинациях наблюдалась рекомбинация родительских маркеров (табл. 3). Заслуживает внимания гибрид S. bayanus var. bayanus (NBRC 1948) × S. bayanus var. uvarum (CBS 7001), имеющий достаточно высокую выживаемость аскоспор: 54.3% (табл. 3). Следует отметить, что ранее изученные нами гибриды штаммов CBS 380, CBS 424 и CBS 425 (S. bayanus var. bayanus) и CBS 7001 (S. bayanus var. uvarum) характеризовались пониженной выживаемостью спор: 9–39% [7, 14, 38].
Таблица 3.
Анализ гибридов генетических популяций комплекса S. bayanus: новозеландская (PYCC 6867, PYCC 6869), S. bayanus var. bayanus (CBS 424, NBRC 1948), S. bayanus var. uvarum (CBS 7001, NCAIM Y.00677, UWO(PS) 99-808 и PYCC 7083) и S. eubayanus (CBS 12357)
| Происхождение гибридов и их генотипы | Число изолированных тетрад | Жизнеспособность аскоспор, % | Мейотическое расщепление гибридов |
|---|---|---|---|
| 6867 × 6869 ura/lys |
36 | 68.1 | 3P:12N:4T* |
| 6867 × 7001 ura/lys |
136 | 11.4 | 11 ura LYS : 24 URA lys : 22 URA LYS : 5 ura lys |
| 6867 × 00677 ura/lys |
105 | 6.2 | 1 ura LYS : 12 URA lys : 9 URA LYS : 4 ura lys |
| 6867 × 99-808 ura/lys |
101 | 23.3 | 10 ura LYS : 19 URA lys : 37 URA LYS : 28 ura lys |
| 6867 × 424 ADE/ade |
35 | 18.6 | 10 ADE : 16 ade |
| 6869 × 1948 lys/ura |
29 | 19.0 | 6 ura LYS : 6 URA lys : 3 URA LYS : 7 ura lys |
| 6867 × 12357 ura/lys |
118 | 19.5 | 14 ura LYS : 34 URA lys : 31 URA LYS : 13 ura lys |
| 7083×12357 ura/lys |
20 | 2.5 | 2 URA lys |
| 1948 × 7001 LYS/lys |
35 | 54.3 | 44 LYS : 32 lys |
| 1948 × 424 ADE/ade |
31 | 23.4 | 17 ADE : 12 ade |
| 1948 × 12357 ura/lys |
19 | 17.1 | 2 ura LYS : 4 URA lys : 5 URA LYS : 2 ura lys |
Мы суммировали результаты гибридологического анализа генетических популяций комплекса S. bayanus, полученные в настоящей работе, и ранее опубликованные данные [7, 14, 16, 38, 46]. По выживаемости гибридных аскоспор изученные популяции можно разделить на две группы (рис. 5). Выживаемость аскоспор гибридов S. bayanus var. bayanus × S. eubayanus составила 55–62%, что сопоставимо с фертильностью гибридов при скрещивании разных штаммов S. bayanus var. bayanus: 64%. Новозеландские штаммы образовывали низко фертильные гибриды со всеми генетическими популяциями: 6.2–23.3%. Низкую выживаемость аскоспор также имели гибриды S. bayanus var. uvarum с S. bayanus var. bayanus и S. eubayanus: 9–39 и 2.5–11% (рис. 5). Исключением является достаточно фертильный гибрид CBS 7001 × NBRC 1948: 54.5%. Следует отметить, что гибриды штамма NBRC 1948 со штаммами новозеландской популяции, S. bayanus var. bayanus и S. eubayanus характеризовались низкой выживаемостью аскоспор: 17.1–23.4% (рис. 5).
ОБСУЖДЕНИЕ
Проведенное исследование подтвердило сложное строение комплекса S. bayanus, включающего, по крайней мере, четыре генетические популяции: S. bayanus var. bayanus, S. bayanus var. uvarum, S. eubayanus и новозеландская. Выживаемость аскоспор существенно зависела от родительских комбинаций и составила 55–62% у гибридов S. bayanus var. bayanus × S. eubayanus, 9–39% у S. bayanus var. bayanus × S. bayanus var. uvarum и 2.5–11% у S. bayanus var. uvarum × S. eubayanus (рис. 5). Результаты гибридологического и молекулярного анализов свидетельствуют о генетической дивергенции штаммов новозеландской популяции, которые значительно отличаются по всем пяти изученным молекулярным маркерам и образуют полустерильные гибриды с представителями остальных популяций: 6.2–23.3% (рис. 5). Независимо от выживаемости аскоспор у всех изученных гибридов наблюдалось регулярное мейотическое расщепление контрольных маркеров, включая двойные ауксотрофы. Внутрипопуляционные скрещивания были фертильны и также характеризовались мейотической рекомбинацией контрольных маркеров. Проведенный нами ранее гибридологический анализ более 100 штаммов S. bayanus var. uvarum, выделенных из различных ферментационных и природных источников в разных регионах мира (Западной, Центральной и Восточной Европы, Дальневосточной и Юго-восточной Азии, Северной и Южной Америки, Гавайских островов), выявил высокую выживаемость гибридных аскоспор: 89–100% [1, 15–18, 46]. Дрожжи S. eubayanus также обнаружены в разных регионах мира: Аргентине, Чили, США, Канаде, Китае, Австралии и Новой Зеландии [24, 32–35]. С другой стороны, все известные штаммы дрожжей S. bayanus var. bayanus были выделены исключительно в Европе, в основном из пивоварения [7, 13].
С помощью ПДРФ-анализа межгенного спейсера IGS2 рДНК можно четко дифференцировать S. bayanus var. uvarum и новозеландские изоляты от штаммов S. bayanus var. bayanus и S. eubayanus, имеющих идентичные AluI-паттерны. Сравнительный анализ ядерных и митохондриальных генов также выявил более близкое генетическое родство последних двух популяций. Характерная для S. bayanus var. uvarum реципрокная транслокация, затрагивающая хромосомы VI и X, отсутствует у штаммов S. bayanus var. bayanus, S. eubayanus и новозеландской популяции. Исключением являются выделенные из пивоварения штаммы S. bayanus var. bayanus NBRC 1948 и CBS 378, у которых также имеется указанная реципрокная транслокация. Следует отметить, что гибрид штамма NBRC 1948 с S. bayanus var. uvarum CBS 7001 имел 54%-ную выживаемость аскоспор, тогда как гибрид NBRC 1948 × S. bayanus var. bayanus CBS 424 был полустерильным: 23.4%. По-видимому, европейская популяция S. bayanus var. bayanus является связующим звеном между S. eubayanus и S. bayanus var. uvarum.
Таким образом, между S. bayanus var. bayanus, S. bayanus var. uvarum, S. eubayanus и новозеландской популяциями нет полной межвидовой постзиготической изоляции. Согласно полученным генетическим и молекулярным данным указанные таксоны относятся к одному биологическому виду, обладая дивергенцией геномов на уровне таксономических разновидностей.
Исследование выполнено в рамках государственного задания АААА-А20-120093090015-2.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Наумов Г.И., Наумова Е.С., Мартыненко Н.Н., Маснёф-Помаред И. Таксономия, экология и генетика дрожжей Saccharomyces bayanus – нового объекта в науке и практике // Микробиология. 2011. Т. 80. № 6. С. 723–730.
Vaughan-Martini A., Martini A. Saccharomyces Meyen ex Reess (1870) // The Yeast, a Taxonomic Study / Eds Kurtzman C.P., Fell J.W., Boekhout T. 5th ed. Amsterdam: Elsevier, 2011. V. 2. P. 733–746. https://doi.org/10.1016/B978-0-444-52149-1.00061-6
Naumov G.I., James S.A., Naumova E.S. et al. Three new species in the Saccharomyces sensu stricto complex: Saccharomyces cariocanus, Saccharomyces kudriavzevii and Saccharomyces mikatae // Int. J. Syst. Evol. Microbiol. 2000. V. 50. P. 1931–1942. https://doi.org/10.1099/00207713-50-5-1931
Kurtzman C.P. Phylogenetic circumscription of Saccharomyces, Kluyveromyces and other members of the Saccharomycetaceae, and the proposal of the new genera Lachancea, Nakaseomyces, Naumovia, Vandervatozyma and Zygotorulaspora // FEMS Yeast Res. 2003. V. 4. P. 233–245. https://doi.org/10.1016/S1567-1356(03)00175-2
Wang S.A., Bai F.Y. Saccharomyces arboricolus sp. nov., a yeast species from tree bark // Int. J. Syst. Evol. Microbiol. 2008. V. 58. P. 510–514. https://doi.org/10.1099/ijs.0.65331-0
Naseeb S., James S.A., Alsammar H. et al. Saccharomyces jurei sp. nov., isolation and genetic identification of a novel yeast species from Quercus robur // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 2046–2052. https://doi.org/10.1099/ijsem.0.002013
Naumova E.S., Naumov G.I., Masneuf-Pomarede I. et al. Molecular genetic study of introgression between Saccharomyces bayanus and S. cerevisiae // Yeast. 2005. V. 22. P. 1099–1115. https://doi.org/10.1002/yea.1298
Peris D., Pérez-Torrado R., Hittinger C.T. et al. On the origins and industrial applications of Saccharomyces cerevisiae × Saccharomyces kudriavzevii hybrids // Yeast. 2018. V. 35. P. 51–69. Epub 2017 Dec 6https://doi.org/10.1002/yea.3283
Morard M., Benavent-Gil Y., Ortiz-Tovar G. et al. Genome structure reveals the diversity of mating mechanisms in Saccharomyces cerevisiae × Saccharomyces kudriavzevii hybrids, and the genomic instability that promotes phenotypic diversity // Microbial. Genomics. 2020. V. 6(3). P. e000333. https://doi.org/10.1099/mgen.0.000333
Lopandic K. Saccharomyces interspecies hybrids as model organisms for studying yeast adaptation to stressful environments // Yeast. 2018. V. 35. P. 21–38. https://doi.org/10.1002/yea.3294
Bendixsen D.P., Frazão J.G., Stelkens R. Saccharomyces yeast hybrids on the rise // Yeast. 2022. V. 39. P. 40–54. https://doi.org/10.1002/yea.3684
Nguyen H.-V., Gaillardin C. Two subgroups within the Saccharomyces bayanus species evidenced by PCR amplification and restriction polymorphism of the non-transcribed spacer 2 in the ribosomal DNA unit // Syst. Appl. Microbiol. 1997. V. 20. P. 286–294. https://doi.org/10.1016/S0723-2020(97)80075-6
Pérez-Través L., Lopes C.A., Querol A., Barrio E. On the complexity of the Saccharomyces bayanus taxon: Hybridization and potential hybrid speciation // PLoS One. 2014. V. 9(4). P. e93729. https://doi.org/10.1371/journal.pone.0093729
Наумов Г.И. Новая разновидность S. bayanus var. uvarum, установленная генетическим анализом // Микробиология. 2000. Т. 69. № 2. С. 410–414.
Naumov G.I., Naumova E.S., Gaillardin C. Genetic and karyotypic identification of wine Saccharomyces bayanus yeasts isolated in France and Italy // Syst. Appl. Microbiol. 1993. V. 16. P. 274–279. https://doi.org/10.1016/S0723-2020(11)80480-7
Naumov G.I., Masneuf I., Naumova E.S. et al. Association of S. bayanus var. uvarum with some French wines: Genetic analysis of yeast populations // Res. Microbiol. 2000. V. 151. P. 683–691. https://doi.org/10.1016/S0923-2508(00)90131-1
Naumov G.I., Naumova E.S., Aigle M. et al. Genetic reidentification of the pectinolytic yeast strain SCPP as Saccharomyces bayanus var. uvarum // Appl. Microbiol. Biotechnol. 2001. V. 55. P. 108–111. https://doi.org/10.1007/s002530000480
Naumov G.I., Nguyen H.-V., Naumova E.S. et al. Genetic identification of Saccharomyces bayanus var. uvarum, a cider-fermenting yeast // Int. J. Food. Microbiol. 2001. V. 65. P. 163–171. https://doi.org/10.1016/S0168-1605(00)00515-8
Masneuf-Pomarede I., Le Jeune C., Durrens P. et al. Molecular typing of wine yeast strains Saccharomyces bayanus var. uvarum using microsatellite markers // System. Appl. Microbiol. 2007. V. 30. P. 75–82. https://doi.org/10.1016/j.syapm.2006.02.006
Almeida P., Gonçalves C., Teixeira S. et al. A gondwanan imprint on global diversity and domestication of wine and cider yeast Saccharomyces uvarum // Nat. Commun. 2014. V. 5. P. 4044–4055. https://doi.org/10.1038/ncomms5044
Zhang H., Richards K.D., Wilson S. et al. Genetic characterization of strains of Saccharomyces uvarum from New Zealand wineries // Food Microbiol. 2015. V. 46. P. 92–99. https://doi.org/10.1016/j.fm.2014.07.016
Rodríguez M.E., Pérez-Través L., Sangorrín M.P. et al. Saccharomyces uvarum is responsible for the traditional fermentation of apple chicha in Patagonia // FEMS Yeast Res. 2017. V. 17(1). P. fow109. https://doi.org/10.1093/femsyr/fow109
McCarthy G.C., Morgan S.C., Martiniuk J.T. et al. An indigenous Saccharomyces uvarum population with high genetic diversity dominates uninoculated Chardonnay fermentations at a Canadian winery // PLoS One. 2021. V. 16(2). P. e0225615. https://doi.org/10.1371/journal.pone.0225615
Libkind D., Hittinger C.T., Valério E. et al. Microbe domestication and the identification of the wild genetic stock of lager-brewing yeast // Proc. Natl Acad. Sci. USA. 2011. V. 108. P. 14539–14544. https://doi.org/10.1073/pnas.1105430108
Pulvirenti A., Nguyen H.V., Caggia C. et al. Saccharomyces uvarum, a proper species within Saccharomyces sensu stricto // FEMS Microbiol. Lett. 2000. V. 192. P. 191–196. https://doi.org/10.1111/j.1574-6968.2000.tb09381.x
Nguyen H.V., Gaillardin C. Evolutionary relationships between the former species Saccharomyces uvarum and the hybrids Saccharomyces bayanus and Saccharomyces pastorianus; reinstatement of Saccharomyces uvarum (Beijerinck) as a distinct species // FEMS Yeast Res. 2005. V. 5. P. 471–483. https://doi.org/10.1016/j.femsyr.2004.12.004
Rainieri S., Kodama Y., Kaneko Y. et al. Pure and mixed genetic lines of Saccharomyces bayanus and Saccharomyces pastorianus and their contribution to the lager brewing strain genome // Appl. Environ. Microbiol. 2006. V. 72. P. 3968–3974. https://doi.org/10.1128/AEM.02769-05
Nguyen H.-V., Lepingle A., Gaillardin C. Molecular typing demonstrates homogeneity of Saccharomyces uvarum strains and reveals the existence of hybrids between S. uvarum and S. cerevisiae, including the S. bayanus type strain CBS 380 // System. Appl. Microbiol. 2000. V. 23. P. 71–85. https://doi.org/10.1016/S0723-2020(00)80048-X
Rainieri S., Zambonelli C., Hallsworth J.E. et al. Saccharomyces uvarum, a distinct group within Saccharomyces sensu stricto // FEMS Microbiol. Lett. 1999. V. 177. P. 177–185. https://doi.org/10.1111/j.1574-6968.1999.tb13729.x
Bon E., Neuvéglise C., Casaregola S. et al. Genomic exploration of the hemiascomycetous yeasts: 5. Saccharomyces bayanus var. uvarum // FEBS Lett. 2000. V. 487. P. 37–41. https://doi.org/10.1016/s0014-5793(00)02276-6
Nguyen H.-V., Legras J.L., Neuvéglise C., Gaillardin C. Deciphering the hybridisation history leading to the Lager lineage based on the mosaic genomes of Saccharomyces bayanus strains NBRC 1948 and CBS 380 // PLoS One. 2011. V. 6(10). P. e25821. https://doi.org/10.1371/journal.pone.0025821
Bing J., Han P.J., Liu W.Q. et al. Evidence for a Far East Asian origin of lager beer yeast // Curr. Biol. 2014. V. 24. P. 380–381. https://doi.org/10.1016/j.cub.2014.04.031
Peris D., Langdon Q.K., Moriarty R.V. et al. Complex ancestries of lager-brewing hybrids were shaped by standing variation in the wild yeast Saccharomyces eubayanus // PLoS Genet. 2016. V. 12. P. 1–20. https://doi.org/10.1371/journal.pgen.1006155
Gayevskiy V., Goddard M.R. Saccharomyces eubayanus and Saccharomyces arboricola reside in North Island native New Zealand forests // Environ. Microbiol. 2016. V. 18. P. 1137–1147. https://doi.org/10.1111/1462-2920.13107
Nespolo R.F., Villarroel C.A., Oporto C.I. et al. An out-of-patagonia migration explains the worldwide diversity and distribution of Saccharomyces eubayanus lineages // PLoS Genetics. 2020. V. 16(5). e1008777. https://doi.org/10.1371/journal.pgen.1008777
Baker E., Wang B., Bellora N. et al. The genome sequence of Saccharomyces eubayanus and the domestication of Lager-brewing yeasts // Mol. Biol. Evol. 2015. V. 32(11). P. 2818–2831. https://doi.org/10.1093/molbev/msv168
Hebly M., Brickwedde A., Bolat I. et al. S. cerevisiae × × S. eubayanus interspecific hybrid, the best of both worlds and beyond // FEMS Yeast Res. 2015. V. 15. № 3. P. fov005.https://doi.org/10.1093/femsyr/fov005
Наумов Г.И. Генетическое родство и биологический статус индустриально важных дрожжей Saccharomyces eubayanus Sampaio et al. // ДАН. 2017. V. 473(5). P. 622–625. https://doi.org/10.7868/S0869565217110263
Sampaio J.P. Microbe Profile: Saccharomyces eubayanus, the missing link to lager beer yeasts // Microbiology. 2018. V. 164. P. 1069–1071. https://doi.org/10.1099/mic.0.000677
Lõoke M., Kristjuhan K., Kristjuhan A. Extraction of genomic DNA from yeasts for PCR based applications // Biotechniques. 2011. V. 50. P. 325–328. https://doi.org/10.2144/000113672
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Boek J., La Croute D., Fink G.R. A positive selection for mutants lacking orotidine-5'-phosphate decarboxilase activity // Mol. Gen. Genet. 1984. V. 197. P. 345–346.
Sherman F., Fink G.R., Hicks J.B. Laboratory course manual for methods in yeasts genetics // Methods in Yeast Genetics: A Gold Spring Harbor Laboratory Course Manual. N.Y.: Gold Spring Harbor Lab., 1986. P. 50–51.
Fischer G., James S.A., Roberts I.N. et al. Chromosomal evolution in Saccharomyces // Nature. 2000. V. 405. P. 451–454.
Kaneko Y., Banno I. Isolation and genetic characterization of auxotrophic mutants in Saccharomyces bayanus // IFO Res. Comm. 1989. V. 14. P. 104–110.
Наумов Г.И., Газдиев Д.О., Наумова Е.С. Обнаружение биологического вида Saccharomyces bayanus в Дальневосточной Азии // Микробиология. 2003. Т. 72. № 6. С. 834–839.
Дополнительные материалы отсутствуют.



