Кристаллография, 2021, T. 66, № 6, стр. 899-901
Моделирование комплекса N-метил–D-аспартат (NMDA) рецептора с липидами постсинаптической мембраны
А. С. Комолов 1, *, Д. Е. Петренко 1, В. И. Тимофеев 1, 2
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
2 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
* E-mail: askomolov@mail.ru
Поступила в редакцию 22.09.2020
После доработки 06.10.2020
Принята к публикации 06.10.2020
Аннотация
Методом молекулярной динамики промоделирован комплекс N-метил–D-аспартат (NMDA) рецептора с липидами постсинаптической мембраны. Проанализировано поведение составляющих системы во времени. Описаны взаимодействия NMDA-рецептора с липидами постсинаптической мембраны. Показано, что подобный комплекс может быть использован для исследования системы, содержащей NMDA-рецептор и липиды постсинаптической мембраны в различных концентрациях.
ВВЕДЕНИЕ
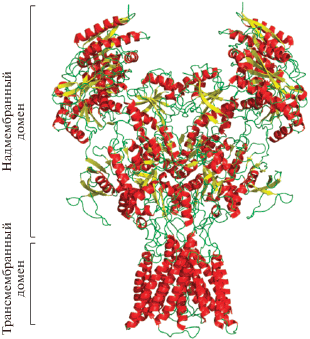
NMDA-рецептор – это ионотропный рецептор глутамата, селективно связывающий N-метил–D-аспартат (NMDA). Структурно рецептор является гетеродимером из четырех субъединиц (рис. 1). В неактивной форме в канале рецептора находится ион магния. Активация рецептора связана с деполяризацией постсинаптической мембраны, ион магния при этом уходит. Активация рецептора приводит к тому, что в клетку начинают поступать ионы натрия и кальция, а ионы калия, напротив, уходят из клетки. Ионы кальция активируют биохимический каскад, определяющий функцию данного рецептора. Процессы, происходящие при активации данного рецептора, играют ключевую роль в синаптической пластичности, а значит, в памяти и обучении [1]. Кроме того, существует гипотеза, что NMDA-рецепторы связаны с процессом сна. Известно, что функционирование рецептора связано с липидным составом постсинаптической мембраны. Основными компонентами липидной составляющей постсинаптической мембраны являются фосфатидилэтаноламин (DLiPE), фосфатидилхолин (DLiPC), фосфотидилсерин (LLPS) и фосфатидилинозитол (DLiPI) в определенном процентном составе [2]. Кроме того, существенную роль в структуре постсинаптической мембраны играет холестерин, т.е. холестерин является одним из основных компонентов, влияющих на вязкость постсинаптической мембраны [3]. Изменение процентного состава липидов в постсинаптической мембране негативно сказывается на работе рецептора, что приводит к снижению когнитивных способностей и ряду заболеваний [3]. Изучению причины данного явления посвящен ряд работ [4–6], однако на структурном уровне данное явление не было детально изучено. Изучить структурный механизм влияния процентного состава липидов постсинаптической мембраны на работу рецептора можно, используя различные методы компьютерного моделирования. В настоящей работе методами компьютерного моделирования получена модель комплекса NMDA-рецептора человека с липидами, входящими в состав постсинаптической мембраны. С помощью метода молекулярной динамики (МД) прослежена эволюция системы во времени. Показано, что описанная методика позволяет исследовать комплекс NMDA-рецептора с липидами постсинаптической мембраны.
МАТЕРИАЛЫ И МЕТОДЫ
В качестве модели для конструирования полноатомной модели NMDA-рецептора применяли пространственную структуру NMDA-рецептора, полученную методом криоэлектронной микроскопии (Protein Data Bank ID: 6IRA) [7]. Для построения отсутствующих аминокислотных оснований использовали программное обеспечение UCSF Chimera [8] с использованием функций достраивания аминокислот, предоставленных программой Modeller [9]. Отклонение конечной модели от изначальной имело среднеквадратичное отклонение (RMSD) = 0.001, что означает правильность и корректность построенной модели. Модель липидов вручную выбирали из архива, представленного на сайте Charmm-gui [10]. Состав мембраны выбран по аналогии с составом внутренней мембраны митохондрий, так как она почти в точности повторяет структуру постсинаптической мембраны. Процентный состав: DLiPE – 37, LLPS – 25.4, DLiPI – 4.5, DliPC – 26.5%.
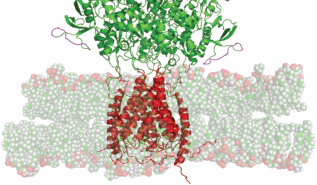
Все компоненты (вода, липиды, NMDA-рецептор) собирали вручную с применением программы PackMol [11]. Размер ячейки 155 × 155 × 210, количество липидов в одном слое DLiPE – 30 (38.4%), LLPS – 21 (26.9%), DLiPI – 5 (6.4%), DliPC – 22 (28.2%), количество молекул воды – 4000. Визуализированная модель представлена на рис. 2.
Рис. 2.
Модель комплекса пространственной структуры NMDA-рецептора и постсинаптической мембраны в начале моделирования. Элементами вторичной структуры показана молекула рецептора, палочками – молекулы липидов и молекулы воды.
В качестве ионов в раствор добавляли 0.15 M KCl. Для липидов использовали силовое поле Charmm36 [10], модель для воды – TIP3P. Дальнейшее моделирование методом МД проводили с использованием программного обеспечения AMBER [12]. Минимизацию потенциальной энергии и релаксацию системы осуществляли с шагом 1 фс. Затем проводили уравновешивание системы при помощи NVT- и NPT-моделей (шаг – 0.001 пс) с условиями 1 атм, 310 K. В качестве баростата использовали МК [13] с шагом 0.001 пс. В качестве термостата – термостат Ланжевена [14] с таким же шагом. Продуктивную МД проводили с шагом в 0.002 пс в течение 2 нс.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Методом компьютерного моделирования получена структурная модель комплекса NMDA-рецептора с моделью фрагмента постсинаптической мембраны. Полученная модель исследована методом МД. Протяженность МД-траектории составила 2 нс. Установлено, что система является стабильной и позволяет описать динамику взаимодействия рецептора с основными компонентами липидной фракции постсинаптической мембраны. Общий вид липидного бислоя с NMDA-рецептором показан на рис. 2. На рисунке представлено расположение трансмембранного домена NMDA-рецептора, а также большого надмембранного домена, отвечающего за связывание активатора рецептора (молекулы глутамата), относительно липидного бислоя. Картина взаимодействия с бислоем показана на рис. 3. Видно, что с бислоем взаимодействует как трансмембранный домен рецептора (остатки 546–658 и 809–847 каждой из четырех субъединиц), так и подвижные петли 416–427 надмембранного домена. Отметим, что в процессе 2 нс МД контакты подвижных петель с липидным бислоем не нарушались, что может свидетельствовать об их функциональном значении. Для анализа подвижности рецептора рассчитывали RMSF для его Сα-атомов. Из рис. 4 видно, что существенные движения, которые могут свидетельствовать об изменении конформации элементов вторичной структуры белка, практически отсутствуют. Исключение составляют C- и N-концевые участки субъединиц рецептора. Использованный в работе подход будет использован в дальнейшем для изучения влияния изменения липидного состава постсинаптической мембраны и содержания холестерина в последней на структуру и функцию рецептора.
Рис. 3.
Взаимодействие NMDA-рецептора и модели постсинаптической мембраны после 200 пс уравновешивания системы. Элементами вторичной структуры показана молекула рецептора (темным обозначен трансмембранный домен, светлым – надмембранный), сферами – атомы в молекулах липидов. Стрелками обозначены подвижные петли 416–427.

Работа выполнена при поддержке Министерства науки и высшего образования РФ в рамках выполнения работ по Государственному заданию ФНИЦ “Кристаллография и фотоника” РАН в части расчетов молекулярной динамики и при поддержке НИЦ “Курчатовский институт” (приказ № 1363 от 25.06.2019) в части анализа результатов компьютерного моделирования.
Список литературы
Van Dongen A.M. // Biology of the NMDA Receptor Frontiers in Neuroscience. Boca Raton: CRC, 2009. P. 57.
Cotman C., Blank M.L., Moehl A., Snyder F. // Biochemistry. 1969. V. 8. № 11. P. 4606. https://doi.org/10.1021/bi00839a056
Renner M., Choquet D., Triller A. // J. Neurosci. 2009. V. 29. № 9. P. 2926. https://doi.org/10.1523/JNEUROSCI.4445-08.2009
Dotti C.G., Esteban J.A., Ledesma M.D. // Front. Neuroanat. 2014. V. 8. P.76. https://doi.org/10.3389/fnana.2014.00076
Unwin N. // IUCrJ. 2020. V. 7. № 5. P. 852. https://doi.org/10.1107/S2052252520009446
Tulodziecka K., Diaz-Rohrer B.B., Farley M.M. et al. // Mol. Biol. Cell. 2016. V. 27. № 22. P. 3480. https://doi.org/10.1091/mbc.E16-06-0420
Zhang J.B., Chang S., Xu P. et al. // Cell Rep. 2018. V. 25. P. 3582. https://doi.org/10.1016/j.celrep.2018.11.071
Pettersen E.F., Goddard T.D., Huang C.C. et al. // J. Comput. Chem. 2004. V. 25. № 13. P. 1605. https://doi.org/10.1002/jcc.20084
Šali A., Blundell T.L. // J. Mol. Biol. 1993. V. 234. P. 779. https://doi.org/10.1006/jmbi.1993.1626
Jo S., Kim T., Iyer V.G. et al. // J. Comput. Chem. 2008. V. 29. № 11. P. 1859. https://doi.org/10.1002/jcc.20945
Martínez L., Andrade R., Birgin E.G. et al. // J. Comput. Chem. 2009. V. 30. № 13. P. 2157. https://doi.org/10.1002/jcc.21224
Case D.A., Belfon K., Ben-Shalom I.Y. et al. // AMBER 2020, University of California, San Francisco.
Steinbach P.J. // Proteins. 2004. V. 57. P. 665. https://doi.org/10.1002/prot.20247
Allen M.P.,Tildesley D.J. // Computer Simulation of Liquids. New York: Oxford University press, 1991. P. 532.
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография