Молекулярная биология, 2021, T. 55, № 3, стр. 362-391
Что могут рассказать белки WOX? Обзор мишеней, регуляторов, партнеров
В. Е. Творогова a, *, Е. Ю. Красноперова a, А. А. Кудряшов a, Э. А. Поценковская a, И. Е. Додуева a, Л. А. Лутова a
a Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: krubaza@mail.ru
Поступила в редакцию 18.08.2020
После доработки 13.10.2020
Принята к публикации 14.10.2020
Аннотация
WOX (WUSCHEL-RELATED HOMEOBOX) – это семейство гомеодоменсодержащих транскрипционных факторов растений. Белки WOX поддерживают активность меристем различного типа, а также регулируют формирование органов растений, влияя на пролиферацию и дифференцировку клеток в различных тканях. Изучение семейства WOX может оказаться полезным для разработки методик трансформации и редактирования генома растений. В настоящем обзоре рассмотрены функции и механизмы работы транскрипционных факторов WOX, их мишени, партнеры и регуляторы. Семейство WOX можно разделить на три филогенетически обособленные клады: так называемую древнюю, промежуточную и кладу WUS. Каждой из ветвей семейства посвящен отдельный раздел обзора. Наиболее подробно рассмотрены гены WOX Arabidopsis thaliana, описаны также их ортологи у других видов растений. Приведены сводные таблицы с известными мишенями, регуляторами и партнерами членов семейства WOX.
ВВЕДЕНИЕ
WOX (WUSCHEL-RELATED HOMEOBOX) – это семейство гомеодоменсодержащих транскрипционных факторов (ТФ), участвующих в регуляции роста и развития растений. В геноме Arabidopsis thaliana обнаружено 15 членов семейства WOX, включая ген WUSCHEL (WUS), а также гены WOX1–14 [1]. Семейство WOX можно разделить на три большие клады: так называемую древнюю, или T1, в которую попадают гены WOX10, 13 и 14 A. thaliana, промежуточную (T2), включающую WOX8, 9, 11 и 12, и современную кладу (T3, или кладу WUS), куда входят WUS и WOX1–7 [2, 3]. Согласно данным филогенетического анализа, ветвь WUS впервые появилась у общего предка Polypodiidae и Spermatophyta [4]. В то же время члены промежуточной клады можно встретить у растений, начиная с Lycopodiophyta [5], хотя, согласно одному из последних исследований, они встречаются только у семенных растений [3]. Гены WOX древней ветви обнаруживаются в геномах растений, начиная с Chlorophyta [6]. В настоящем обзоре рассмотрены функции членов данного семейства и регуляторные модули, в составе которых они работают.
СОВРЕМЕННАЯ ВЕТВЬ (T3/ВЕТВЬ WUS)
В состав ТФ WOX современной клады входит не только гомеодомен, но и так называемый WUS-мотив, обычно необходимый для функциональной активности ТФ, а некоторые факторы (WUS, WOX5 и WOX7) содержат и домен EAR (ERF-associated amphiphilic repression), обладающий репрессорной активностью [3]. В основном гены WOX современной клады участвуют в регуляции функционирования различных типов меристем.
WUS
Функции WUS в меристемах побега. Ген WUS A. thaliana известен в первую очередь как фактор, поддерживающий функционирование меристем побега за счет стимуляции пролиферации и подавления дифференцировки их стволовых клеток. WUS функционирует в апикальной меристеме побега (ПАМ), меристеме соцветия, пазушных меристемах и меристеме цветка. ПАМ растений с потерей функции гена WUS (wus-1) способна дать начало лишь небольшому числу листьев, после чего прекращает функционировать. На ее поверхности образуются новые меристемы, но любая из них через небольшой промежуток времени прекращает свою работу, и ситуация повторяется. Сходными дефектами обладают меристемы соцветий, а меристемы цветка также дифференцируются раньше, чем в норме, и вместо шести тычинок и двух плодолистиков формируют всего лишь одну тычинку в центре [7]. Потеря функции WUS также приводит к снижению частоты образования пазушных меристем. Напротив, стимуляция активности WUS приводит, в частности, к увеличению размеров ПАМ [8] и количества пазушных меристем [9].
WUS способен не только поддерживать функционирование меристем побега, но и обеспечивать их идентичность. Так, эктопическая экспрессия WUS в отдельных клетках корней A. thaliana приводит к развитию структур, подобных ПАМ, образующих листоподобные органы. При добавлении ауксина в среду на кончиках таких корней развиваются соматические эмбрионы, а на фоне сверхэкспрессии гена LEAFY, регулятора цветения, – неупорядоченно расположенные органы цветка [10]. В системе регенерации побегов из эксплантов гипокотилей A. thaliana потеря функции WUS делает невозможной регенерацию побегов in vitro, а его избыточная экспрессия может приводить к регенерации побегов на безгормональной среде [11].
WUS начинает экспрессироваться уже в эмбрионе на стадии 16 клеток во внутренних клетках апикального домена. В постэмбриональном развитии этот ген экспрессируется в ПАМ и меристеме соцветия в клетках организующего центра (ОЦ), а также в центральной части меристемы цветка. В ходе развития цветка уровень экспрессии WUS постепенно уменьшается, и его РНК не детектируется, когда стволовые клетки меристемы окончательно расходуются на образование плодолистиков [12]. Также экспрессия WUS наблюдается при заложении пазушной меристемы; в ходе дальнейшего развития домен экспрессии ограничивается ее ОЦ [13]. Таким образом, ген WUS в меристемах побега обычно оказывает влияние не на те клетки, в которых экспрессируется (клетки ОЦ), а на стволовые клетки, расположенные выше [12]. Белок WUS способен мигрировать из ОЦ в соседние клетки, и его присутствие в слоях L1 и L2 важно для выполнения его функций в ПАМ [14, 15]. WUS способен формировать гомодимеры, а его подвижность, вероятно, ограничивается в том числе за счет гомодимеризации [15].
Ортологи WUS обнаружены у множества видов растений, в том числе у голосеменных [16]. Обычно паттерны экспрессии и функции WUS A. thaliana и его ортологов сходны, однако встречаются и различия. Так, ортологи WUS у Medicago truncatula, Oryza sativa и Zea mays экспрессируются в листовых примордиях [17–19]. При этом потеря функции ортолога WUS у риса, гена MONOCULM3, или TILLERS ABSENT1 (TAB1 ), не влияет на активность ПАМ, но приводит к недоразвитию боковых побегов, связанную с дефектами при заложении пазушных меристем [20]. TAB1 не экспрессируется в ПАМ, но активен в меристеме соцветия и при заложении пазушных меристем [20, 21]. Функцию поддержания ПАМ у O. sativa выполняет другой ген семейства WOX – OsWOX4 (см. ниже). При формировании пазушных меристем TAB1 экспрессируется до образования видимой пазушной меристемы, в так называемой премеристематической зоне, в дальнейшем его экспрессия исчезает, но уже непосредственно в пазушных меристемах начинает экспрессироваться OsWOX4 [20].
WUS и CLV3. Наиболее известным регулятором экспрессии WUS является CLE-пептид CLAVATA3 (СLV3). Ген CLV3 экспрессируется в центральной зоне ПАМ и меристемы соцветия (над ОЦ), и потеря его функции приводит к увеличению размера ПАМ, которая приобретает более выпуклую форму вследствие эктопической экспрессии WUS [22]. Напротив, сверхэкспрессия CLV3 приводит к остановке развития ПАМ и к подавлению экспрессии WUS [23]. Рецепция CLV3 осуществляется с помощью мембранных рецепторных киназ с лейцин-богатыми повторами, в частности, CLAVATA1 (CLV1) и RECEPTOR-LIKE PROTEIN KINASE 2 (RPK2), способных формировать гомодимеры. В рецепции CLV3 также участвует комплекс из рецептороподобного мембранного белка CLV2, не имеющего киназного домена, и мембранной псевдокиназы CORYNE (CRN). Фенотипические проявления мутаций в генах, кодирующих белки CLV1, RPK2, CRN или CLV2, сходны с фенотипом растений с потерей функции CLV3. Помимо этого, гены рецепторных киназ из группы BARELY ANY MERISTEM (ВАМ1 и 2), которые в норме экспрессируются вокруг центральной зоны ПАМ и поддерживают ее стволовые клетки, в случае потери функции CLV1 начинают экспрессироваться в центральной зоне. В этом случае данные киназы способны частично выполнять функции CLV1, по-видимому, также передавая сигнал от CLV3 к WUS [24]. Путь дальнейшей передачи сигнала от CLV3 до репрессии транскрипции WUS известен не до конца, однако выяснено, что в рецепции CLV3 участвуют также киназы CLAVATA3 INSENSITIVE RECEPTOR KINASE1-4 (CIK1-4), действующие после перечисленных рецепторов [25]. Другими посредниками в передаче сигнала от CLV3 к WUS являются фосфатазы POLTERGEIST1 (POL1) и POLTERGEIST-LIKE 1 (PLL1) [26]. В норме CLV3 ингибирует работу фосфатаз POL и PLL1, активаторов экспрессии WUS [27]. Фосфатидилинозит-4-фосфат (PI(4)P) стимулирует активность POL и PLL1. Поскольку при передаче сигнала от рецепторов, связавших CLV3, активность POL и PLL1 должна подавляться, предполагается, что рецепторы могут блокировать либо синтез PI(4)Р, либо его связывание с POL и PLL1 [28]. Другие предполагаемые посредники в передаче сигнала CLV3 – G-белки [29], MAP-киназы MPK3 и 6 [30], а также ионы кальция [31]. В свою очередь, ТФ WUS активирует экспрессию гена CLV3. Так, например, эктопическая экспрессия WUS в примордиях развивающихся листьев приводит к появлению в них клеток, сходных с меристематическими, в которых экспрессируется CLV3 [22], а потеря функции WUS приводит к отсутствию или снижению активности промотора CLV3 [23]. Эти данные говорят о существовании в ПАМ системы обратной связи WUS-CLV, которая ограничивает зону экспрессии WUS в ОЦ [22] (рис. 1). Аналогичные отношения между ортологами WUS и CLV3 наблюдаются также и у других видов, например у Cucumis sativus [32]. WUS активирует транскрипцию CLV3 [14], связываясь, в частности, с пятью близко расположенными регуляторными элементами в 3′-области гена CLV3, которые образуют так называемый цис-регуляторный модуль [14, 33]. Анализ мутаций в этих элементах и соотнесение их с аффинностью к WUS, а также другие эксперименты позволили предположить, что при высокой концентрации WUS формирует гомодимеры и подавляет экспрессию CLV3, а в случае его низкой концентрации мономеры WUS, напротив, стимулируют экспрессию CLV3. Это позволяет не активировать CLV3 в ОЦ, но активировать его в центральной зоне ПАМ [33]. Однако согласно другому исследованию, основным фактором, определяющим влияние WUS на экспрессию CLV3, являются белки HAIRY MERISTEM (HAM) (см. ниже) [34]. WUS также подавляет экспрессию CLV1, связываясь с его промотором [35].
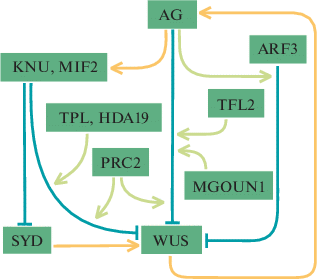
WUS и AGAMOUS. Другой регуляторный модуль, включающий WUS, связан с геном AGAMOUS (AG), кодирующим ТФ с доменом MADS, и регуляцией терминации меристемы цветка. Согласно модели развития цветка ABCDE, AG – это ген класса C, отвечающий за развитие андроцея и гинецея [36]. В растениях с мутацией в гене AG, в отличие от растений дикого типа, меристема цветка остается недетерминированной, и гены WUS и CLV3 не перестают экспрессироваться на поздних стадиях развития цветка. AG в норме подавляет транскрипцию WUS, тем самым способствуя терминации развития меристемы цветка после заложения всех цветковых органов [37]. WUS, действуя совместно с активатором цветения LFY, запускает экспрессию AG, связываясь с его локусом [37, 38]. Таким образом, гены AG и WUS, связанные петлей обратной связи (рис. 2), ответственны за прекращение активности стволовых клеток цветочной меристемы и детерминированность цветка. AG подавляет экспрессию WUS с помощью нескольких путей. В одном из них AG активирует транскрипцию гена, кодирующего ТФ KNUCKLES (KNU) с мотивом цинковых пальцев C2H2 и с доменом EAR [39], и гена ТФ MINI ZINC FINGER2 (MIF2) [40]. Белки KNU и MIF2 в составе комплекса с корепрессором TOPLESS (TPL) и деацетилазой гистонов HISTONE DEACETYLASE 19 (HDA19) подавляют экспрессию WUS, связываясь с его промотором; аналогичный регуляторный модуль обнаружен и у Solanum lycopersicum [40]. Репрессия локуса WUS под действием KNU опосредована не только деацетилированием гистонов: KNU также рекрутирует в локус WUS комплекс PRC2 (Polycomb-repressive complex 2), что приводит к накоплению репрессирующих меток H3K27me3, а также способствует снижению количества активирующих меток H3K4me3 в локусе WUS и вытеснению белка SPLAYED (SYD) – активатора WUS (см. ниже) [41].
AG также репрессирует WUS, связываясь с регуляторными последовательностями в его локусе и, вероятно, рекрутируя PRC2 [42]. Кроме того, AG взаимодействует с компонентом PRC1 – белком TERMINAL FLOWER2 (TFL2)/LIKE HETEROCHROMATIN PROTEIN1 (LHP1), также участвующим во внесении метки H3K27me3. Методами 3С и ChIP-3C показано, что в меристеме цветка AG и TFL2, связываясь друг с другом, участвуют в образовании хроматиновой петли в локусе WUS, подавляя тем самым экспрессию WUS [43]. В образование петли вовлечены так называемые CArG-боксы в 3′-регуляторной области WUS – сайты связывания AG. Интересно, что мутации в CArG-боксах в локусе ортолога WUS у S. lycopersicum – гена Locule number (Lc) – приводят к повышению уровня его экспрессии при развитии плодов и к увеличению размера плода и числа камер в нем; нуклеотидные замены в этих локусах можно обнаружить у многих культивируемых сортов томата [44, 45]. Еще один белок, вовлеченный в регуляцию локуса WUS белком AG – ДНК-топоизомераза IB MGOUN1/TOP1α, которая облегчает связывание AG с WUS посредством уменьшения количества нуклеосом в данном локусе, что в конечном счете приводит к подавлению экспрессии WUS [46].
Третий возможный путь репрессии WUS ТФ AG связан с действием ауксин-чувствительных факторов. Мутация в гене AUXIN RESPONSE FACTOR 3 (ARF3) приводит к разрастанию медиальной части гинецея, что указывает на нарушение терминации меристемы цветка. ARF3 подавляет активность WUS в меристеме цветка, а связыванию ARF3 с локусом WUS способствует AG [47]. В то же время регулятор развития цветка из группы A – ген APETALA2 (AP2), кодирующий ТФ из класса AP2/EREBP, действует как антагонист AG. AP2 опосредованно стимулирует экспрессию WUS, в том числе, вероятно, путем прямой репрессии ARF3 [47].
С помощью генетического анализа, оценки взаимодействия белков и других экспериментов показано, что AG, ген класса C, функционирует в составе тетрамерных комплексов вместе с другими белками с доменом MADS из функциональных групп B, D и E [36]. Согласно данным генетического анализа и анализа экспрессии ортолога WUS TERMINATOR (TER) у Petunia hybrida, лишь совместное действие ТФ классов C, D и E может подавить экспрессию TER в меристеме цветка [48]. Эти данные и результаты генетического анализа других видов позволяют предполагать, что AG и его ортологи подавляют экспрессию WUS или его ортологов в составе тетрамерного комплекса [49, 50].
Другие белковые регуляторы WUS. Экспрессию WUS регулируют не только AG и CLV3, но и множество других факторов. В частности, к предполагаемым прямым репрессорам WUS относятся ULTRAPETALA1 (цистеин-богатый белок с B‑боксом и ДНК-связывающим доменом SAND) [51, 52] и способный к взаимодействию с ним ULT1 INTERACTING FACTOR 1 (UIF1) с Myb- и EAR-доменами [53]. Потеря функции ULT1 и/или UIF приводит к развитию цветков с увеличенным количеством чашелистиков и лепестков [53]. UIF1 способен связываться с промотором WUS. Можно предположить, что UIF1 и ULT1 взаимодействуют друг с другом и репрессируют активность WUS в меристеме цветка [53]. Согласно данным генетического анализа, при развитии соцветий и формировании меристем цветка WUS действует в том числе через подавление ULT1 [51].
Важным регулятором развития ПАМ является также ген STM (SHOOTMERISTEMLESS). Показано, что в регуляции ПАМ STM и WUS действуют параллельно, при этом они необходимы для нормальной экспрессии друг друга [12, 54, 55]. Результаты многочисленных исследований указывают на связь между другими регуляторами развития ПАМ – ТФ HD-ZIPIII – и геном WUS. Анализ растений с мутацией cna1 в гене CORONA (CNA), кодирующем ТФ семейства HD-ZIPIII, растений с мутацией jba1D, для которых характерна сверхэкспрессия гена микроРНК (miR166G, репрессор генов HD-ZIPIII), и сочетания этих мутаций с мутацией wus-1 говорит о негативной регуляции экспрессии WUS факторами HD-ZIPIII [56, 57]. Тем не менее, показано прямое связывание ТФ HD-ZIPIII PHABULOSA (PHB), PHAVOLUTA (PHV) и REVOLUTA (REV) с локусом WUS в ходе регенерации побегов и их взаимодействие с ТФ ARABIDOPSIS RESPONSE REGULATOR (ARR) типа B (ARR1 и ARR2), прямыми активаторами WUS (см. далее). Эти ТФ HD-ZIPIII, как и ARR1 и ARR2, важны для регенерации, хотя разные сочетания мутаций в этих генах приводят как к снижению, так и к повышению способности к регенерации побегов. Предполагается, что ARR типа B и указанные HD-ZIPIII образуют комплекс, который активирует WUS в ходе регенерации, однако влияние HD-ZIPIII на развитие ПАМ и на экспрессию WUS в целом неоднозначно [11]. Также HD-ZIPIII могут, по-видимому, действовать на меристемы побега не только через WUS: одновременная потеря функции PHB, PHV и CNA приводит к частичной супрессии фенотипа мутантов wus-1 и частичному восстановлению ПАМ [58].
Изучение ортологов WUS у других видов позволило обнаружить еще несколько ТФ, регулирующих экспрессию данных ортологов путем связывания с их локусами: специфическая форма ТФ c доменом MADS CsFRUITFULL1 – активатор CsWUS у C. sativus [32], ТФ из группы AP2/ERF EXCESSIVE NUMBER OF FLORAL ORGANS (ENO) – репрессор Lc (SlWUS) у S. lycopersicum [59], PtrTALE12 – активатор PtrWUS у Populus trichocarpa [60].
WUS и фитогормоны. Взаимодействию WUS и фитогормонов, главным образом ауксинов и цитокининов, посвящено множество работ. Сигнальные пути цитокининов и WUS связаны друг с другом положительной обратной связью. WUS подавляет экспрессию генов ARR типа A (ARR5, 6, 7 и 15), кодирующих репрессоры ответа на цитокинины, также подтверждено связывание WUS с промотором гена ARR7. Таким образом, WUS действует как стимулятор передачи цитокининового сигнала [61]. Тем не менее, данные генетического анализа и математическое моделирование показывают, что WUS, вероятно, негативно влияет на синтез цитокининов (возможно, посредством репрессии гена LONELY GUY 4 (LOG4), кодирующего фермент активации цитокининов) [62].
Обработка растений цитокинином приводит к увеличению уровня экспрессии WUS, к расширению домена его экспрессии и к появлению фенотипа, сходного с фенотипом растений с мутациями в генах CLAVATA [63, 64]. Кроме того, цитокинины способны стабилизировать белок WUS, тем самым стимулируя его распространение в клетки, в которых ген WUS не экспрессируется [65]. Паттерн цитокининового ответа в ПАМ сходен с паттерном экспрессии WUS, а домены экспрессии AHK4 (ген рецептора цитокинина) и WUS значительно перекрываются между собой [63]. Промотор гена LOG4, кодирующего фермент, активирующий цитокинины, работает в слое L1 в ПАМ, что позволяет предполагать наличие источника активных форм цитокининов в этом слое. Предполагается, что место экспрессии WUS и, соответственно, позиция ОЦ в ПАМ динамически определяются комбинированным антагонистическим влиянием концентраций цитокинина и CLV3 и устанавливаются в зоне, где содержание цитокининов, поставляемых из L1, еще достаточно высоко, содержание CLV3 уже достаточно мало, а также присутствует рецептор цитокинина AHK4 [62].
ТФ ARR типа B – положительные регуляторы цитокининового ответа – в частности, ARR1, 2, 10 и 12, активируют экспрессию WUS в ходе регенерации побегов, в ПАМ [66–68] и при заложении пазушных меристем [9]. При этом цитокинин стимулирует связывание ARR с локусом WUS [68, 69]. Нарушение работы ARR B-типа приводит к подавлению этих процессов и к уменьшению размера ПАМ из-за снижения уровня экспрессии WUS [9, 11, 66, 69]. Тем не менее, эффект ARR B-типа на регенерацию не всегда однозначен. Так, растения с потерей функции ARR1 характеризуются повышенной способностью к каллусообразованию и побегообразованию корневых эксплантов, а сверхэкспрессия гена ARR1 снижает эту способность. Однако в отсутствие функционального ARR12 ARR1, напротив, стимулирует побегообразование. Его ингибирующий эффект на регенерацию связан, в частности, с тем, что он вытесняет ARR12 из локусов CLV3 и WUS – стимуляторов побегообразования – будучи при этом не таким эффективным стимулятором экспрессии, как ARR12 [70].
Взаимное влияние WUS и ауксинов не однозначно. В ПАМ ауксиновый ответ локализуется в первую очередь в примордиях листьев, а в стволовых клетках центральной зоны наблюдается его минимум. Тем не менее, подавление действия ауксина в ПАМ зачастую приводит к ее терминации. Таким образом, стволовые клетки ПАМ требуют для своего поддержания определенного уровня ауксинового ответа. WUS способен непосредственно регулировать экспрессию множества генов, связанных с ауксиновым ответом и синтезом ауксина: TIR1, MONOPTEROS (MP)/ARF5 и других ARF, TARGET OF MONOPTEROS6/TMO6, TRYPTOPHAN AMINOTRANSFERASE RELATED2 (TAR2). Таким образом, WUS поддерживает в ПАМ уровень ауксинового ответа на стабильно низком, но не нулевом уровне [71].
Влияние ауксина на экспрессию WUS изучено в различных системах регенерации. В частности, для регенерации побегов in vitro экспланты часто культивируют сначала на среде с высоким содержанием ауксина (CIM, callus-inducing medium), а затем на среде с высоким содержанием цитокинина (SIM, shoot-inducing medium). Полярный транспорт ауксина, нужный для формирования его градиента, необходим для активации промотора WUS на SIM [72]. Необходимость определенной концентрации ауксина для экспрессии WUS показана и в системе получения соматических эмбрионов. Образование ауксиновых максимумов сначала в будущем апикальном домене соматического эмбриона, а затем в развивающихся примордиях семядолей коррелирует с активацией промотора WUS в зоне, соответствующей ПАМ [73]. Нарушение ауксинового ответа приводит к изменению домена экспрессии WUS и к подавлению формирования соматических эмбрионов [74].
Эпигенетические регуляторы WUS. В регуляции WUS участвуют также многие эпигенетические факторы. Так, мутации в различных генах, кодирующих эпигенетические репрессоры, такие как метилтрансфераза гистонов KRYPTONITE, ДНК-метилтрансфераза METHYLTRANSFERASE1 (MET1) и другие, приводят к повышенному уровню экспрессии WUS в каллусах и увеличению скорости регенерации побегов из таких каллусов, что предполагает важную роль эпигенетических модификаций в активации WUS в ходе регенерации [75, 76]. В пазухах молодых листьев в локусе WUS наблюдаются высокие уровни репрессирующей метки H3K27me3 и низкие уровни активирующей метки H3ac, в то время как в пазухах зрелых листьев, в которых формируются пазушные меристемы, уровень H3ac возрастает, а H3K27me3 – снижается, что делает возможной активацию WUS и формирование пазушных меристем [9].
К числу эпигенетических активаторов экспрессии WUS можно отнести хроматин-ремоделирующий фактор SPLAYED (SYD) из семейства SNF, который связывается с промотором WUS. У растений с потерей функции гена SYD наблюдается более ранняя терминация меристемы соцветия и уменьшение уровня экспрессии WUS в ней [77]. Белок BARD1 (BRCA1-associated RING domain 1), или ROW1 (REPRESSOR OF WUS1), способен связываться с участком промотора WUS, близким к сайту связывания SYD, а также взаимодействовать с SYD, однако при этом он является репрессором WUS [78]. Влияние на паттерны и уровень экспрессии WUS выявлено при изучении множества других эпигенетических регуляторов и ТФ, таких как FASCIATA1 и 2 (субъединицы фактора сборки хроматина CAF-1 (Chromatin assembly factor-1)) [79], AtRING1a и AtRING1b (компоненты комплекса PRC1) [80], FILAMENTOUS FLOWER и YABBY3 из семейства YABBY [81], HANABA TARANU из семейства GATA [82], ТФ OBERON1-4 c PHD-доменом (plant homeodomain) [83] и другие.
Физиологические регуляторы WUS. Обнаружено влияние ряда небелковых веществ, а также факторов окружающей среды на экспрессию WUS. Так, супероксид-анион, содержание которого повышено в центральной зоне ПАМ, стимулирует экспрессию WUS, а пероксид водорода, который концентрируется в периферической зоне ПАМ – подавляет. Нарушение баланса данных веществ в ПАМ может привести к изменению ее размера или даже к терминации [84]. Кроме того, активность промотора WUS в проростках стимулируется продуктами фотосинтеза (сахароза), а также уровнем освещенности. Интересно, что активность phyB, фитохрома, отвечающего за восприятие красного света, в мезофилле, эпидермисе или проводящих тканях стимулирует активность промотора WUS в ПАМ. Таким образом, вероятно, существует некий мобильный сигнал, который переносится в ПАМ и активирует ее работу после восприятия света. Одним из передатчиков активирующего сигнала к WUS от света и продуктов фотосинтеза, является киназа TARGET OF RAPAMYCIN [85].
Иные мишени WUS. WUS поддерживает стволовые клетки ПАМ не только за счет взаимодействия с сигнальными путями цитокининов и ауксинов, но и с помощью регуляции экспрессии множества генов, кодирующих различные ТФ. Среди прямых мишеней WUS обнаружены гены ТФ, стимулирующих дифференцировку, включая KANADI1 (KAN1), KAN2, ASYMMETRIC LEAVES2 (AS2) и YABBY3. В норме WUS мигрирует в периферическую зону ПАМ, подавляя экспрессию генов, отвечающих за дифференцировку, в то время как ТФ, кодируемые этими генами, в частности KAN1, напротив, ограничивают экспрессию WUS, делая ее возможной только в ОЦ [86]. WUS также подавляет в ПАМ гены группы HECATE (HEC), кодирующие ТФ из группы bHLH, связываясь с их локусами. Анализ мутантов показывает, что HEC1, 2 и 3 действуют на ПАМ сходным с WUS образом, однако при этом оказывают противоположный эффект на гены, регулируемые WUS. Так, например, HEC1 активирует экспрессию генов ARR типа A. Таким образом, HEC1 может выполнять в ПАМ двоякую функцию, с одной стороны, стимулируя пролиферацию стволовых клеток за счет активации специфических мишеней и действуя параллельно WUS, но с другой, подавляя ее с помощью других механизмов, в частности, за счет репрессии цитокининового ответа [87]. CaWUS, ортолог WUS у Cicer arietinum, также связывается с промотором гена, кодирующего CabHLH121 – ТФ из группы bHLH, вовлеченный в контроль размеров ПАМ и растения в целом [88].
Партнеры WUS. Работа WUS в ПАМ осуществляется в том числе за счет взаимодействия с белками-партнерами. Одними из первых обраруженных кофакторов WUS были белки-корепрессоры TOPLESS (TPL) и TOPLESS-RELATED 1 (TPR1), 2 и 4 [89, 90]. Способностью к взаимодействию с ортологами TPR обладает ортолог WUS риса – белок MONOCULM3 (MOC3) [91]. Эксперименты по комплементации мутантов wus-1 показали, что для выполнения WUS своих функций требуется его взаимодействие с TPL (или, возможно, TPR1, 2 и 4) [92]. Белки группы TOPLESS осуществляют репрессию транскрипции за счет взаимодействия с деацетилазами гистонов [93]. Активация WUS приводит к изменению уровня ацетилирования гистонов в большом количестве локусов, с которыми он связывается [71], что, вероятно, и приводит к их репрессии данным ТФ. При этом WUS непосредственно активирует экспрессию TPL и подавляет экспрессию TPR1 и 2 [35]. WUS также может взаимодействовать с ТФ HAIRY MERISTEM1-4 (HAM1-4) с доменом GRAS. Растения с мутациями ham1, ham2, ham3 и ham4 имеют укороченные корни, у них наблюдается остановка развития ПАМ и нарушено развитие сосудистых тканей. Таким образом, HAM1–4 необходимы, в частности, для поддержания функционирования ПАМ. Домены активности промоторов HAM1 и 2 перекрываются с доменом активности промотора WUS. Уровень экспрессии прямых мишеней WUS изменяется сходным образом в мутантах wus-1 или ham1 ham2 ham3, и было показано, что HAM2 связывается с некоторыми из этих локусов в тех же участках, в которых с ними связывается WUS [94]. Совместное участие HAM и ортолога WUS (TERMINATOR) в поддержании ПАМ обнаружено также у P. hybrida [95]. Тем не менее, HAM1 и 2 в отличие от WUS, подавляют экспрессию CLV3. Потеря функции генов HAM или отсутствие градиента их экспрессии (что наблюдается, например, при заложении придаточных меристем) приводит к формированию паттерна экспрессии CLV3 с максимумом в зоне экспрессии WUS. Эти данные, а также математическое моделирование позволяют предположить, что гетеродимеры WUS-HAM подавляют экспрессию CLV3, а мономеры WUS – активируют [34]. К предполагаемым партнерам WUS и его ортологов можно также отнести белки из группы SQUAMOSA PROMOTER BINDING PROTEIN у Glycine max [96] и A. thaliana, ТФ из группы CmCYC2 (семейство TCP) у Chrysantemum morifolium [97] и ТФ DRINK ME с доменом bZIP у A. thaliana [98].
Иные функции WUS. WUS участвует не только в поддержании меристем побега, но и в ряде других аспектов жизни растения. Так, проростки с потерей функции WUS имеют укороченный гипокотиль из-за снижения интенсивности пролиферации. WUS является непосредственным активатором гена GRP23 (GLUTAMINE-RICH PROTEIN 23), кодирующего ядерный белок с PPR-мотивом, взаимодействующий с РНК-полимеразой II. Белок GRP23 опосредует влияние WUS на длину гипокотиля [99]. Мутация jam в промоторе гена WUS приводит к снижению уровня экспрессии WUS и к развитию растений, у которых не формируются листья ювенильного фенотипа (с меньшим количеством трихомов и более простым жилкованием) [100]. Подобный эффект обусловлен снижением у растений с мутацией в гене WUS уровня экспрессии гена микроРНК miR156 (регулятор перехода из ювенильной фазы в фазу взрослого фенотипа) и увеличением уровня экспрессии одной из мишеней этой микроРНК – гена SPL9 (SQUAMOSA PROMOTER BINDING PROTEIN-LIKE9) – в ПАМ и примордиях листьев [101]. У некоторых видов ортологи WUS еще более существенно влияют на развитие листьев. Так, у растений M. truncatula с потерей функции гена HEADLESS (HDL) – ортолога WUS, помимо прочего, уменьшена длина листьев и изменена их форма, что, возможно, связано с нарушением транспорта ауксина и паттернов ответа на ауксин [17, 18].
WUS влияет также на фенотип цветка, действуя не только как регулятор развития его меристемы, но и как непосредственный участник развития органов цветка. Так, экспрессия WUS обнаружена в развивающихся пыльниках между пыльцевыми гнездами и позднее, в ходе развития тычинки, – в периферических клетках пыльников [56, 102]. Пыльники растений с потерей функции WUS часто имеют меньшие или неправильно сформированные доли по сравнению с пыльниками дикого типа и не раскрываются [103]. ТФ PHB из семейства HD-ZIPIII непосредственно подавляет экспрессию SPOROCYTELESS(SPL)/NOZZLES(NZZ) – стимулятора развития микроспороцитов – и активирует экспрессию WUS для детерминации границ между микроспорангиями, в то время как miR165/166 ограничивает активность PHB и делает возможной экспрессию SPL в развивающихся микроспорангиях [102].
WUS также экспрессируется в нуцеллусе – дистальной части семязачатка. В растениях, у которых отсутствует экспрессия WUS в семязачатке, не формируются наружный и внутренний интегументы [104, 105]. Напротив, эктопическая экспрессия WUS в халазе достаточна для формирования в нижней части семязачатка дополнительных структур, похожих на интегументы [104–106]. Также в семязачатках растений с потерей функции WUS останавливается развитие зародышевого мешка или мегаспора вообще не формируется. Согласно генетическому анализу, WUS опосредованно активирует гены WINDHOSE 1 и 2 (WIH1 и 2), кодирующие белки с GYPP-мотивом, которые стимулируют развитие мегаспоры и зародышевого мешка [106]. Тем не менее, WUS не экспрессируется в самой материнской клетке мегаспоры, его эктопическая активация в этой клетке приводит к развитию нескольких мегаспор и, в дальнейшем, зародышевых мешков. В норме экспрессия WUS в материнской клетке мегаспоры прямо репрессируется с помощью RETINOBLASTOMA-RELATED PROTEIN 1 [107]. В свою очередь, стимулятором экспрессии WUS (опосредованным или прямым) является ТФ SPL/NZZ: потеря его функции приводит к нарушению развития нуцеллуса и снижению уровня экспрессии WUS в нем [105]. Подобная схема регуляции обнаружена также у C. sativus, при этом подавление экспрессии CsSPL приводит к снижению уровня экспрессии CsWUS как в женских, так и в мужских цветках, а тычинки и семязачатки развиваются аномально. Интересно, что CsSPL и CsWUS способны к взаимодействию [108]. Напротив, эктопической экспрессии WUS в халазе и фуникулусе препятствуют белки BEL1, SEEDSTICK, SHATTERPROOF1 и 2 (возможно, образуя тримеры BEL1 + AG/SEEDSTICK/ SHATTERPROOF1/2 + SEPALLATA1/3 [109]), а также ТФ группы HD-ZIPIII [110].
WUS также является стимулятором соматического эмбриогенеза (СЭ): in vitro его эктопическая экспрессия может приводить к формированию соматических эмбрионов на безгормональной среде [111], а подавление экспрессии WUS приводит к снижению количества соматических эмбрионов [73]. Сверхэкспрессия WUS A. thaliana в гипокотильных эксплантах Gossypium hirsutum и его индуцибельная экспрессия в Capsicum chinense стимулирует развитие соматических эмбрионов, хотя дальнейшая регенерация полноценных проростков в этих случаях не наблюдается [112–114]. Индуцибельная экспрессия WUS A. thaliana в Coffea canephora также может приводить к увеличению способности к СЭ [115]. Напротив, индукция экспрессии WUS A. thaliana негативно влияет на СЭ у Picea glauca [116]. Также показано, что эктопическая экспрессия ZmWUS2 вместе с ZmBABYBOOM значительно улучшает (а в некоторых случаях делает возможной) трансформацию различных линий Zea mays и ряда других злаков за счет стимуляции регенерации [117], а использование тканеспецифичного промотора для ZmBBM и индуцибельного для ZmWUS2 сделало возможным прямой (без стадии каллуса) СЭ после трансформации зиготических эмбрионов Z. mays [118]. Активация экспрессии ортологов WUS выявлена в ходе СЭ у Vitis vinifera [119], Cocos nucifera [120], Panax ginseng [121] и других видов. У M. truncatula MtWUS экспрессируется как в эмбриогенных, так и в неэмбриогенных каллусах [122, 123], однако в эмбриогенных каллусах экспрессируется MtCLV3, что, вероятно, ограничивает транскрипцию MtWUS в зонах формирования соматических эмбрионов [122]. HaWUS у Helianthus annuus способен связываться с локусом LEC1-подобного гена HaL1L [124], предполагаемого регулятора СЭ. В ходе развития эмбрионов уровень экспрессии HaWUS постепенно увеличивается, в то время как уровень активности HaL1L снижается, что позволяет предположить репрессию гена HaL1L HaWUS [124]. Подавление транскрипции LEC1 наблюдали и после стимуляции экспрессии WUS в эксплантах A. thaliana [111].
WOX5
Ген WOX5 и его ортологи у других видов растений – одни из основных регуляторов работы КАМ. Предполагаемая потеря функции WOX5 у растений A. thaliana с мутацией wox5-1 приводит к увеличению размера клеток ПЦ, а также к увеличению и дифференцировке стволовых клеток колумеллы, что подтверждается накоплением в них гранул крахмала [125]. При этом частота делений клеток ПЦ в мутантах wox5-1 увеличивается, а число слоев клеток колумеллы уменьшается [126]. Такой же эффект потеря функции WOX5 оказывает на КАМ боковых корней, а также снижает частоту образования боковых корней [127]. Индуцируемая сверхэкспрессия WOX5 подавляет дифференцировку клеток колумеллы и боковых клеток корневого чехлика [125], а эктопическая экспрессия при ее низком уровне стимулирует клеточные деления [128]. WOX5 обычно начинает экспрессироваться в ходе эмбриогенеза после стадии 16 клеток в гипофизе, а после ее деления – в линзовидной клетке, которая дает начало ПЦ, а затем в самом ПЦ. Экспрессия WOX5 наблюдается также в примордиях семядолей [1]. Во взрослом растении, кроме ПЦ, промотор WOX5 активируется на ранних стадиях развития боковых и придаточных корней, в дальнейшем активность остается в ПЦ корня [127, 129–131].
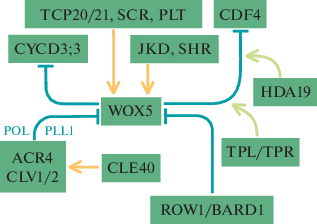
Известные на настоящий момент мишени и регуляторы WOX5 в КАМ представлены на рис. 3. Для поддержания стволовых клеток в КАМ WOX5 репрессирует ген ТФ CDF4 (CYCLING DOF FACTOR4), стимулирующего дифференцировку клеток колумеллы, путем рекрутирования факторов TPL/TPR к его промотору. В свою очередь, факторы TPL/TPR рекрутируют деацетилазу гистонов HDA19 для удаления активирующих ацетильных групп с гистонов в промоторе CDF4. При этом WOX5 перемещается из ПЦ в стволовые клетки колумеллы [129]. Тем не менее, немобильная версия WOX5, сшитая с двумя белками GFP, также способна восстановить присутствие стволовых клеток колумеллы в мутанте wox5-1 [132], что говорит о существовании неизвестного пока механизма работы WOX5 в КАМ. Как и другие белки из ветви WUS, WOX5 способен к взаимодействию с белками HAM. Домены активности промоторов WOX5 и HAM2 перекрываются в ПЦ, а растения с мутациями ham1, ham2, ham3 и ham4 характеризуются, помимо прочего, нарушениями развития КАМ и укороченными корнями, что предполагает участие белков HAM в регуляции КАМ вместе с WOX5 [94].
WOX5 подавляет дифференцировку стволовых клеток и стимулирует клеточные деления в колумелле, однако в отношении клеток ПЦ он действует частично противоположным образом, подавляя их пролиферацию. Известно, что WOX5 прямо подавляет экспрессию CYCD3;3, кодирующего циклин группы D (регулятор перехода в S‑фазу), в ПЦ [126]. Помимо WOX5, в детерминации ПЦ участвуют ТФ из семейства GRAS SHORTROOT (SHR) и SCARECROW (SCR), а также ТФ PLETHORA (PLT) из семейства AP2/ERF. Уровень экспрессии WOX5 уменьшен в растениях с мутациями по генам SHR или SCR, а потеря активности генов PLT1 и PLT2, регулирующих развитие КАМ через ауксиновый сигнальный путь, приводит к расширению домена экспрессии WOX5 [125]. В мутантах wox5-1 активность промоторов SHR, SCR, PLT1 и 2 в ПЦ и в КАМ в целом значительно снижена, а эктопическая активация WOX5 в клетках колумеллы приводит к активации промоторов PLT1 и 2 (но не SHR и SCR). Таким образом, возможно, WOX5 необходим для активации PLT1 и 2 в ПЦ [128, 130]. Согласно данным генетического анализа WOX5 вместе с SCR, SHR, PLT1 и 2 поддерживает также проксимальные стволовые клетки КАМ [125]. При этом ТФ TCP20/21 из семейства TCP (TEOSINTE BRANCHED 1, CYCLOIDEA AND PCF TRANSCRIPTION FACTOR), SCR и PLT образуют комплекс и активируют WOX5 в ПЦ [133, 134]. Также ТФ JACKDAW (JKD) с цинковыми пальцами, взаимодействуя в ПЦ с SHR, активирует экспрессию WOX5 [135].
Исследования взаимодействия WOX5 с фитогормонами посвящены главным образом ауксину. В растениях с мутациями по генам MP или BODENLOS с нарушением ауксинового ответа в эмбриогенезе экспрессия WOX5 практически никогда не детектируется [125], однако обработка ауксином ингибирует экспрессию WOX5 в главном корне. В подавлении экспрессии WOX5 ауксином участвуют ауксин-чувствительные ТФ ARF10 и 16 [130], а INDOLE-3-ACETIC ACID INDUCIBLE 17 (IAA17), ингибитор ауксинового ответа, стимулирует экспрессию WOX5 [136]. WOX5 также влияет на сигнальные пути ауксина: в норме максимальный ауксиновый ответ в КАМ выявляется в ПЦ, однако при потере функции WOX5 он смещается из ПЦ в более дистальные клетки. Кроме того, WOX5 стимулирует продукцию ауксинов в корнях, способствуя экспрессии YUCCA1, кодирующего один из ферментов синтеза ауксина [136].
Экспрессия WOX5, как и WUS, регулируется также пептидом CLE. CLE40 является близким гомологом CLV3. Этот ген экспрессируется в дифференцированных клетках колумеллы и в зоне дифференцировки в клетках стелы. Он подавляет деление стволовых клеток колумеллы и ограничивает домен экспрессии WOX5. Обработка растений синтетическим пептидом CLE40 приводит к уменьшению домена экспрессии WOX5 и к его перемещению в более проксимальные зоны корня (туда же перемещается в этом случае и сам ПЦ, согласно анализу экспрессии других его маркеров) [132]. В рецепции CLE40 и регуляции КАМ участвуют рецепторные киназы ARABIDOPSIS CRINKLY4 (ACR4) [131], CLV1 [137] и белок CLV2 [132]. Интересно, что ACR4 способна фосфорилировать WOX5 in vitro по нескольким сайтам [138]. Дальнейший путь передачи сигнала от киназ до регуляции экспрессии WOX5 не выявлен, однако на экспрессию WOX5, как и WUS, оказывают стимулирующее влияние фосфатазы POL и PLL1 [139]. Обратная связь – стимулирующее воздействие WOX5 на экспрессию CLE40 – у A. thaliana не выявлена, однако обнаружена у гомологов данных генов в КАМ у O. sativa [140]. К другим прямым репрессорам WOX5 относится ТФ ROW1/BARD1. Кодирующий его ген экспрессируется в проксимальной части КАМ, и потеря его функции приводит к развитию коротких корней, нарушению гравитропизма, к потере идентичности ПЦ и другим нарушениям КАМ, а также к расширению домена активности промотора WOX5 [141].
WOX5 и его ортологи активируются также при взаимодействии корней растения с различными симбионтами и/или при развитии нерегулярных меристематических структур. В частности, экспрессия WOX5 активируется в ходе развития корневых галлов при инфицировании нематодой Meloidogyne javanica [142]. Активность промотора WOX5 A. thaliana обнаружена в клетках, прилежащих к меристематическим очагам в генетических опухолях Raphanus sativus [143]. Экспрессия ортологов WOX5 обнаружена в азотфиксирующих клубеньках у M. truncatula, Pisum sativum [144] и Arachis hypogaea [145]. Генетический анализ показал, что количество клубеньков и экспрессия MtWOX5 и его ортолога PsWOX5 в M. truncatula и в P. sativum регулируются, как и в случае КАМ и ПАМ, с помощью генов системы CLAVATA [144]. Экспрессия ортологов WOX5 обнаружена также при прямой и непрямой регенерации корней и при каллусообразовании in vitro, в частности, у M. truncatula [122], Rosa canina [146] и A. thaliana [74]. Экспрессия WOX5 при регенерации корней активируется ТФ WOX11 и 12 (см. ниже). Интересно, что уровень экспрессии WOX5 и других корнеспецифичных генов A. thaliana возрастает при формировании каллуса на среде CIM [147, 148], но снижается при переносе на SIM. Значительную роль в активации WOX5 и других корнеспецифичных генов в каллусе играет ацетилтрансфераза гистонов HAG1 (HISTONE ACETYLTRANSFERASE of the GNAT/MYST superfamily 1), ацетилирующая H3 в локусах WOX5 и 14, SCR, PLT1 и 2 [149]; активация данных генов в каллусе важна для дальнейшей регенерации побегов.Рис. 4.
Рис. 4.
Органы и ткани A. thaliana, на развитие и функционирование которых влияют ТФ WOX. WUS регулирует функционирование ПАМ, меристемы соцветия и меристемы цветка, WOX9 участвует в функционировании ПАМ, WOX1 и WOX3 важны для разрастания боковых органов побега, WOX4 и 14 регулируют работу камбия и прокамбия, WOX5 контролирует функции КАМ, WOX5, 7 и 14 участвуют в развитии боковых корней, WOX11, 12, 5 и 7 регулируют развитие придаточных корней, WOX13 и 14 контролируют сроки цветения, WUS и WOX14 регулируют развитие тычинок, WOX6 и WUS важны для развития семязачатков, WOX13 регулирует развитие плода, WOX2, 8, 9, 1, 3, 5 участвуют в эмбриогенезе.

WOX5 необходим также для прохождения СЭ у A. thaliana: подавление его экспрессии в эксплантах значительно снижает количество формируемых соматических эмбрионов и нарушает их развитие. Для формирования нормального паттерна экспрессии WOX5 при СЭ необходим цитокининовый сигнальный путь. Возможно, цитокинин ограничивает зону экспрессии WOX5, делая возможным формирование ПЦ КАМ [74]. WOX5 также экспрессируется в ходе развития мужского гаметофита. У мутантов tepiztin1 со сниженным уровнем экспрессии WOX5 нарушено функционирование мужского гаметофита, в частности, их пыльцевые трубки растут медленнее и имеют меньшие размеры [150].
WOX1
Основная функция WOX1 и его ортологов – регуляция разрастания боковых органов. Одиночные мутанты A. thaliana с потерей функции WOX1 лишь незначительно отличаются от растений дикого типа, однако потеря функции WOX1 и другого гена WOX современной клады – WOX3/ PRESSED FLOWER (PRS) – приводит к значительным изменениям: листовые пластинки, чашелистики и лепестки таких растений значительно сужены вследствие снижения количества делений клеток; у них нарушена также спецификация клеток кромок листа [151]. WOX1 экспрессируется на стадии сердца и торпеды в закладывающихся сосудах [1], а после прорастания WOX1 и 3 экспрессируются в кромках и срединных слоях примордиев листьев (в так называемом срединном домене листа) [151]. Интересно, что потеря функции WUS усиливает эффект от потери функции WOX1 и 3, в частности, делая листья мутантов еще более узкими. Можно предположить, что WUS также принимает участие в регуляции разрастания листовой пластинки [152]. В развитии листа ТФ семейства YABBY и KANADI определяют развитие абаксиального домена, а ТФ AS2 и REV – развитие адаксиального домена. Согласно анализу множественных мутантов, WOX1 и 3 вместе с данными факторами также участвуют в формировании адаксиально-абаксиальной полярности листа [151, 153]. При этом ТФ YABBY стимулируют активность WOX1, а ТФ KANADI, напротив, подавляют [151]. Другие репрессоры WOX1 и 3 – ТФ из семейств NGATHA (NGA) и CINCINNATA-class-TCP (CIN-TCP), такие как TCP3, TCP4, NGA1 и NGA4, участвуют в подавлении роста листовой пластинки. Растения A. thaliana со сниженным уровнем экспрессии TCP1-5 и NGA1-4 имеют курчавые листья и расширенный домен активности промоторов WOX1 и 3 [154].
Уровень экспрессии WOX1 и 3 также регулируется ауксином. В абаксиальной части листовых примордиев содержание ауксина повышено, однако промоторы MP и ряда других ARF (NONPHOTOTROPIC HYPOCOTYL4 (NPH4)/ARF7, ARF6, 8 и 19) активны преимущественно в адаксиальной части примордиев. Срединный домен является единственной зоной перекрывания экспрессии MP, ARF6, 7, 8 и 19 и повышенного содержания ауксина; именно в этой зоне активны промоторы генов WOX1 и 3. При этом MP активирует WOX1 и 3, связываясь с их локусами. Напротив, гены ARF3 и, вероятно, 2 и 4 экспрессируются в абаксиальной части примордия на определенных стадиях его развития, и кодируемые ими ТФ подавляют экспрессию WOX1 и 3 [155]. WOX1 также регулирует ауксиновый ответ: cогласно транскриптомному анализу, многие из генов, на экспрессию которых влияет WOX1, участвуют в сигнальных путях, транспорте и синтезе ауксина. Так, положительная регуляция характерна для PIN1, AUX1 и MP, а отрицательная, например, для TAR2 и YUC5 [156]. Также WOX1 способен к взаимодействию с S-АМDC1 (S-adenosylmethionine decarboxylase) – ферментом синтеза полиаминов, важных регуляторных молекул растений, а в мутантах со сверхэкспрессией WOX1 уровень полиаминов снижен. Таким образом, WOX1 регулирует развитие латеральных органов, возможно, за счет регуляции синтеза полиаминов [157].
Механизмы работы генов группы WOX1 подробно изучены у ортолога WOX1 M. truncatula – гена STENOFOLIA (STF). В отличие от растений A. thaliana с потерей функции WOX1, не имеющих выраженных фенотипических особенностей, мутанты M. truncatula с потерей функции STF характеризуются узкими листовыми пластинками, нарушениями в формировании проводящей системы листа, а также нарушенным разрастанием лепестков и плодолистиков [158]. STF регулирует разрастание листовой пластинки, связываясь с промотором гена MtAS2, подавляющего пролиферацию клеток в адаксиальном домене листа, и подавляя его экспрессию путем привлечения корепрессора MtTPL [159]. STF способен взаимодействовать с корепрессором (и возможным коактиватором) MtLEUNIG (MtLUG) и коактиватором MtANGUSTIFOLIA3 (MtAN3), а генетический анализ мутаций в генах AN3, LUG, WOX1 и 3 у A. thaliana позволяет предположить, что AN3 и LUG участвуют в разрастании листовой пластинки вместе с WOX3 и WOX1 [160]. Экспрессия STF в различных видах злаков может приводить, помимо прочего, к развитию более широкой листовой пластинки. STF способен подавлять экспрессию генов цитокининоксидаз, увеличивая содержание цитокининов в трансгенных растениях [161], а также участвовать в регуляции метаболизма сахаров [162]. Гены, ортологичные WOX1 и STF – LATHYROIDES у P. sativum [163] и MAEWEST у P. hybrida – также необходимы для нормального разрастания боковых органов, в частности листьев и лепестков [164].
WOX3
WOX3, как и WOX1, участвует в регуляции разрастания боковых органов. У растений A. thaliana с мутацией в гене WOX3, или PRESSED FLOWER (PRS), не развиваются боковые чашелистики, а в абаксиальных и адаксиальных чашелистиках отсутствуют краевые ряды клеток. Число тычинок также уменьшено [165, 166]. Гораздо более серьезные дефекты развития наблюдаются у двойных мутантов A. thaliana с потерей функции WOX3 и WOX1 (см. раздел о WOX1). Интересно, что ортолог WOX3 у M. truncatula – ген LOOSE FLOWER (LFL) – в отличие от своего ортолога WOX3 и паралога STF не участвует в развитии листьев, но необходим для разрастания органов цветка. Согласно результатам генетического и комплементационного анализа, гены STF и LFL обладают функциональным сходством, однако они имеют разные домены экспрессии, что и определяет различия в их функциях в развитии растения [167].
Согласно данным филогенетического анализа, гены группы WOX1 специфичны для двудольных и не встречаются у однодольных [168]. Разрастание боковых органов у однодольных регулируется генами ветви WOX3. Так, потеря функции ортологов WOX3 NARROW SNEATH1 (NS1) и 2 у Z. mays приводит к отсутствию боковых доменов листьев и гомологичных им органов. Эти гены экспрессируются в боковых частях ПАМ (в слоях L1 и L2) и в кромках примордиев боковых органов [166, 169]. У риса ортологами NS1/2 являются гены NARROW LEAF2(NAL2)/3, или ген OsWOX3A/ OsPRS/OsNS, представленный двумя практически идентичными копиями. Cверхэкспрессия NAL2/3 приводит, помимо других эффектов, к развитию растений с более широкими листьями с большим количеством проводящих пучков [170]. У растений с одновременной потерей функции генов NAL2 и 3 наблюдается значительное недоразвитие листовых пластинок, у них отсутствуют щетинки на краях листа и нарушено развитие проводящих пучков в листьях [170, 171]. У таких мутантов нарушена экспрессия генов ARF, YUC и PIN, что предполагает изменение в сигнальных путях, транспорте и синтезе ауксина [170, 171], а согласно генетическому анализу и другим экспериментам, NAL2/3 непосредственно подавляет экспрессию OsYAB3. Как OsYAB3, так и NAL2/3 участвуют, предположительно, в дифференцировке листа, в частности, путем регуляции экспрессии генов семейства KNOX (OSH1 и OSH3) [172]. Другой эффект потери функции NAL2 и 3 – уменьшение количества боковых корней и увеличение количества корневых волосков. Эксперименты с обработкой ауксином позволяют предположить, что оба эти эффекта связаны с уменьшением уровня экспрессии генов OsPIN1 и с концентрацией ауксина не в перицикле, а в эпидермисе, что и приводит к снижению количества боковых корней, которые закладываются в перицикле, но при этом к увеличению количества и длины корневых волосков, которые развиваются из клеток эпидермиса [171, 173]. Сверхэкспрессия NAL2/3 приводит не только к изменениям фенотипа листьев, но и к увеличению количества боковых корней, а также к карликовости. Обработка гиббереллином восстанавливает нормальную высоту растения. NAL2/3 взаимодействует с гиббереллинами по принципу отрицательной обратной связи: их избыток стимулирует экспрессию NAL2/3, тогда как NAL2/3 подавляет синтез этих гормонов, в частности, связываясь с локусом KAO (ENT-KAURENE ACID OXIDASE), кодирующим фермент биосинтеза гиббереллинов, и подавляя его экспрессию [174].
Согласно данным филогенетического анализа, у однодольных (по меньшей мере у риса и кукурузы) имеется дополнительная ветвь, содержащая гомологи WOX3, отличные от генов группы NS [175]. Подробно изучены функции одного из представителей этой ветви – гена OsWOX3B/GLABROUS RICE1 (GLR1)/DEPILOUS (DPL)/NUDA/LEAF LATERAL SYMMETRY1 (LSY1) O. sativa. Подавление экспрессии этого гена приводит к отсутствию трихомов или к сильному уменьшению их количества на листьях и колосковых чешуйках [176–178]. OsWOX3B стимулирует развитие трихомов совместно с ТФ из группы AP2/ERF HAIRY LEAF6 (HL6), близким гомологом ТФ EMBRYOMAKER (его сверхэкспрессия также приводит к развитию трихомов на семядолях A. thaliana). OsWOX3B способен к взаимодействию с HL6, также он непосредственно активирует HL6 [179]. OsWOX3B и HL6 прямо или опосредованно стимулируют экспрессию ряда генов, вовлеченных в синтез, транспорт и передачу сигнала ауксина, таких как OsYUCCA5, OsPIN1a, OsARF4 и 7. HL6 способен связываться с локусом OsYUCCA5, а добавление гомеодомена OsWOX3B усиливает это связывание [179]. OsWOX3B, независимо от NAL2/3, участвует и в разрастании листовой пластинки. Один из эффектов потери его функции – формирование несимметричных листовых пластин из-за отсутствия латеральных разрастаний с одной из сторон листа и нарушение адаксиально-абаксиальной полярности. Кроме того, соцветия у таких мутантов часто не имеют колосковых чешуй, а развитие тычинок и семязачатков у них нарушено [180].
Ортологи WOX3 у голосеменных, как и у покрытосеменных, также участвуют в разрастании боковых органов. Так, подавление экспрессии гена PaWOX3 P. abies – ортолога WOX3 – нарушает развитие семядолей у соматических эмбрионов, а также влияет на морфологию иголок, делая их менее уплощенными и более цилиндрическими [181].
WOX2
WOX2 вместе с ТФ WOX8 и WOX9 регулирует эмбриональное развитие. Ген WOX2 начинает экспрессироваться в яйцеклетке и в центральной клетке. Вместе с WOX2 в этих клетках экспрессируется WOX8, член промежуточной ветви, однако после оплодотворения и деления зиготы эти гены начинают экспрессироваться в разных ее потомках: WOX8 – в базальной клетке (см. раздел о WOX8), а WOX2 – в основном в апикальном домене [1]. Экспрессия ортологов WOX2 в зиготических эмбрионах и/или в семенах в целом выявлена также у N. tabacum [182], O. sativa [183], P. abies [184] и других видов. Потеря функции WOX2 приводит к нарушению упорядоченности деления клеток в апикальном домене, что в данном случае не критично для развития растения [1]. У растений с потерей функции WOX2 в некоторых случаях развивается только одна семядоля, часто нарушен сосудистый паттерн в семядолях [185, 186]. Как уже сказано, в ходе раннего эмбриогенеза WOX2 и 8 экспрессируются в комплементарных друг другу областях, однако у части растений с мутациями в обоих генах наблюдаются различные дефекты семядолей (асимметричные семядоли, частичное их слияние), которые отсутствуют у одиночных мутантов [187]. WOX2 и 8 с помощью пока не выявленного механизма активируют транскрипцию CUP-SHAPED COTYLEDON2 (CUC2) и 3, обеспечивая симметричный характер их экспрессии. Предполагается, что потеря функции WOX2 активирует экспрессию WOX8 в апикальном домене, что, возможно, приводит к их избыточности в регуляции развития семядолей [186].
Одновременная потеря функции генов WOX2, 1 и 3 увеличивает частоту возникновения нарушений развития семядолей (по сравнению с потерей функции только WOX2). Добавление потери функции WOX5 усиливает эти дефекты. Ключевую роль в регуляции клеточных делений апикального домена играет WOX2, а активность остальных генов в этих процессах становится важной только в отсутствие этого гена [185]. Кроме того, WOX2 вместе с генами WOX1, 3 и 5, играющими второстепенные роли, участвует в инициации ПАМ в эмбриогенезе и в первичной активации экспрессии маркеров ПАМ, включая CLV3. Интересно, что промотор CLV3 на стадии сердца активируется за счет WOX1, 2, 3, 5 даже у мутантов wus-1, хотя в дальнейшем его активность пропадает. Таким образом, WOX1, 2, 3, 5, но не WUS, необходимы для запуска экспрессии CLV3 в эмбриогенезе. Нарушение инициации ПАМ у растений с мутациями в генах WOX1, 2, 3 и 5 сопровождается повышением интенсивности ауксинового ответа в зоне ПАМ, что может быть связано с нарушением паттернов экспрессии гена PIN1 в эмбрионах с таким генотипом. Также WOX2 (вместе с WOX1, 3 и 5) стимулирует развитие ПАМ, усиливая экспрессию HD-ZIPIII (PHB, PHV, REV) как на транскрипционном, так и на посттранскрипционном уровнях. PHB, PHV и REV в свою очередь активируют биосинтез цитокининов, необходимых для закладки ПАМ [188].
Ортологи WOX2 экспрессируются также в ходе СЭ у P. pinaster [189], P. abies [184], V. vinifera [119] и ряда других видов. Подавление экспрессии PaWOX2 у P. abies приводит к нарушению развития соматических эмбрионов [184], а WOX8 и WOX2 A. thaliana совместно индуцируют СЭ или регенерацию побегов N. tabacum in vitro [190].
WOX4
WOX4 и его ортологи у большинства видов растений регулируют развитие проводящих тканей. Так, снижение уровня экспрессии WOX4 A. thaliana приводит к уменьшению размера проводящих пучков и нарушению упорядоченной структуры стелы, перицикла и эндодермы. Потеря функции WOX4 у мутантов wox4-1 снижает активность пучкового и межпучкового камбия в стебле, хотя его активность исчезает не полностью [191]. WOX4 экспрессируется в основном в камбии и прокамбии. Активность ортологов WOX4 в камбии выявлена у многих видов растений, включая голосеменные [192].
Роль WOX4 в регуляции развития проводящих тканей изучена в основном в рамках исследования работы сигнального модуля TDIF-TDR-WOX4. TDIF (TRACHEARY ELEMENT DIFFRENTIATION INHIBITORY FACTOR) – CLE-пептид, кодируемый двумя разными генами A. thaliana – CLE41 и CLE44. TDIF и его рецептор TDR (TDIF RECEPTOR)/PXY(PHLOEM INTERCALATED WITH XYLEM) известны как регуляторы, подавляющие дифференцировку клеток камбия в ксилему и стимулирующие пролиферацию камбия [193]. Уровень экспрессии WOX4 повышается в камбии A. thaliana в ответ на обработку TDIF. WOX4, в отличие от TDR и TDIF, не относится к белкам, необходимым для подавления дифференцировки ксилемы, однако, как и эти регуляторы, важен для стимуляции пролиферации клеток прокамбия и камбия. При этом сверхэкспрессия WOX4 не вызывает утолщения стелы, что предполагает присутствие системы отрицательной обратной связи, нейтрализующей влияние излишней активности WOX4. Кроме того, сверхэкспрессия WOX4 не влияет на уровни экспрессии CLE41 и 44 [193]. Показано также, что ортологи WOX4 не влияют на интенсивность экспрессии ортологов CLE41 у гибридов тополя [194]. TDR-зависимое повышение уровня экспрессии WOX4 в проростках наблюдается также при обработке CLE-пептидом HsCLEB типа B, который синтезируют паразитические нематоды Heterodera schachtii, и который, по-видимому, является аналогом TDIF, мимикрирующим под него. Промотор WOX4 активируется при заражении нематодой на определенных этапах развития синцития – структуры, питающей нематоду [195].
Ауксин активирует экспрессию WOX4, что, в свою очередь, индуцирует развитие межпучкового камбия и способствует делению клеток внутри проводящих пучков. При этом TDR/PXY необходим для стабильной активации экспрессии WOX4 ауксином. Усиление камбиальной активности ауксином зависит от клеточного контекста, что, вероятно, связано со специфическими ауксин-чувствительными ТФ [191]. Так, MP ограничивает количество клеток камбия в стебле, прямо репрессируя ген WOX4. В то же время другие ARF (ARF3 и 4), наоборот, стимулируют активность камбия. При этом MP работает в самом камбии, в клетках, экспрессирующих WOX4, а ARF3 и ARF4 – и в камбии, и за его пределами – в ксилеме и флоэме [196]. Тем не менее, в другом исследовании показано положительное влияние MP на WOX4 и на развитие камбия в корне. Обнаружено, что прилегающий непосредственно к камбиальной зоне слой клеток с уже установленной ксилемной идентичностью выполняет функции ОЦ, поддерживающего работу камбия. Там сосредоточен локальный максимум индолилуксусной кислоты (ИУК), и экспрессируются гены группы HD-ZIPIII и MP, поддерживая идентичность этого слоя. Подавление экспрессии MP на фоне потери функции ARF7 и 19 снижает уровень транскрипции WOX4, ряда генов HD-ZIPIII, а также дифференцировки вторичной ксилемы. Индукция экспрессии miR-165a, ингибитора всех пяти HD-ZIPIII, снижает уровень активности камбия и также подавляет экспрессию WOX4 [197]. В регуляции пролиферации клеток проводящих тканей WOX4 действует совместно с WOX14 (см. WOX14) [198]. Кроме того, сверхэкспрессия WOX4, как и потеря его функции, снижает уровень экспрессии WOX14 [199]. WOX4 способен взаимодействовать с белками группы HAM, а домены активности промоторов HAM4 и WOX4 перекрываются в меристемах проводящих тканей [94].
У O. sativa ортолог WOX4 не только регулирует развитие проводящей системы, но и участвует в поддержании ПАМ и развитии листьев. При подавлении экспрессии OsWOX4 формируются аномальные ПАМ, которые быстро терминируются, наблюдается нарушение закладки и роста листьев, а также развития их проводящей системы [200, 201]. При этом CLE-пептиды FCP1/2 подавляют экспрессию OsWOX4 и стимулируют развитие листьев [201]. С помощью транскриптомного анализа и определения содержания гормонов показано, что OsWOX4 стимулирует синтез цитокининов путем активации экспрессии генов группы LOG [200].
Гены группы WOX4, как и многие другие гены WOX, участвуют не только в развитии проводящей системы и ПАМ, но и в регенерации: ортологи WOX4 экспрессируются в развивающихся эмбриогенных каллусах Coffea canefora [202], M. truncatula [203], в соматических эмбрионах V. vinifera [119] и других видов.
WOX6
WOX6/PRETTY FEW SEEDS2 (PFS2)/HIGH EXPRESSION OF OSMOTICALLY RESPONSIVE GENES 9 (HOS9) регулирует развитие семязачатков. PFS2 экспрессируется в ПАМ и в примордиях листьев, в меристемах цветка, примордиях органов цветка, пыльниках, пестиках и семязачатках, а также, в меньшей степени, – в плодолистиках, лепестках и чашелистиках. У растений с нарушением функции этого гена (pfs2-1) уменьшена материнская клетка мегаспор и, по-видимому, вследствие этого нарушено развитие зародышевого мешка. У мутантов pfs2-1 формируется намного меньше жизнеспособных семязачатков, чем у растений дикого типа. Также у них укорочены интегументы, нарушено развитие лепестков и листьев. Эта мутация проявляется на стадии спорофита: гетерозиготные растения PFS2/pfs2-1 формируют нормальные семязачатки. [204, 205]. PFS2, по-видимому, участвует также в подавлении экспрессии AG [205], а возможными репрессорами экспрессии самого PFS2 могут быть LRR-киназы ERECTA и ERECTA-LIKE1 и 2 [206].
Анализ другой мутации в гене WOX6 показывает, что этот ген участвует в ответе на холодовой стресс: мутанты hos9-1 со вставкой Т-ДНК в 3′-часть гена обладают большей чувствительностью к отрицательным температурам, у них повышены уровни экспрессии ряда генов, вовлеченных в ответ на холод. Эти мутанты отличаются более медленным ростом корней и побегов, а также более поздним цветением, они имеют узкие и мелкие листья и меньшее количество трихомов [207].
WOX7
WOX7 регулирует развитие боковых корней. Промотор гена WOX7 активен в кончиках корней в клетках-инициалях кортекса и эндодермы и в самой эндодерме, а также в примордиях боковых корней, преимущественно до их появления на поверхности корня. У растений с мутацией в WOX7 повышено количество боковых корней и примордиев боковых корней, а сверхэкспрессия WOX7 приводит к снижению числа боковых корней. WOX7 подавляет экспрессию гена циклина CYCD6;1, однако у растений с мутацией в CYCD6;1 количество боковых корней даже немного больше, чем у растений дикого типа. Активность промотора WOX7 при этом подавляется при обработке ауксином, что соответствует противоположным ролям этих регуляторов в контроле заложения боковых корней. Добавление в среду глюкозы или сахарозы активирует промотор WOX7. Высокие концентрации этих сахаров при повышенном соотношении содержания углерода и азота подавляют образование боковых корней, однако этот эффект отсутствует в растениях с нарушением функции WOX7. Таким образом, возможно, WOX7 опосредует ингибирующее действие сахаров на образование боковых корней [208].
ПРОМЕЖУТОЧНАЯ ВЕТВЬ (T2)
ТФ WOX промежуточной клады обычно вовлечены в регуляцию эмбриогенеза и регенерации. Функционально и филогенетически ТФ данной ветви разделены на две группы: WOX8/9 и WOX11/12. ТФ WOX11/12 A. thaliana обладают сходными функциями, поэтому они рассмотрены вместе.
WOX8
WOX8/STIMPY-LIKE (STPL), как уже упоминалось, является регулятором эмбриогенеза, он участвует в формировании апикально-базальной оси полярности зародыша. WOX8 экспрессируется в яйцеклетке и в центральной клетке, а после оплодотворения и деления зиготы – в базальной клетке и ее потомках, клетках суспензора и гипофизы, и в части эндосперма [1]. У растений с потерей функции WOX8 часто нарушен сосудистый паттерн в семядолях, а ауксиновый максимум в базальном полюсе распространяется на весь суспензор, в отличие от растений дикого типа, у которых он локализован лишь в базальной части эмбриона и в самой верхней части суспензора. Возможно, WOX8 активирует экспрессию PIN7 и обеспечивает отток ауксина из суспензора [186]. Функции WOX8 в эмбриогенезе частично выполняют также гены WOX2 и WOX9 (см. соответствующие разделы). Важный регуляторный модуль, включающий (по меньшей мере, до настоящего времени) WOX8, но не WOX2 и 9, связан с CLE8 – пептидом из группы CLE. CLE8 экспрессируется в эмбрионе и в эндосперме. У растений с потерей функции CLE8 эндосперм недоразвит, а развитие эмбрионов и суспензора нарушено. CLE8 необходим также для активации WOX8 в суспензоре и эндосперме. Сверхэкспрессия CLE8 ведет к увеличению размера семян, однако потеря функции WOX8 снимает этот эффект. Вероятно, WOX8 опосредует влияние CLE8 на развитие эмбриона и эндосперма [209].
WOX8 вместе с WOX9 участвует в формировании апикально-базальной оси полярности. Согласно данным генетического анализа, для их активации необходим ТФ WRKY2 с доменом типа “цинковые пальцы” [210]. Транскрипцию WOX8 совместно активируют ТФ WRKY2 и ТФ из семейства HD-ZIPIV HOMEODOMAIN GLABROUS11/12 (HDG11/12). Активация ТФ WRKY2 опосредуется киназой SHORT SUSPENSOR, мРНК которой поступает в зиготу из спермия. В то же время HDG11/12 экспрессируются в яйцеклетке, т.е. для активации WOX8 необходимо совместное действие и материнских, и отцовских факторов [211].
WOX9
WOX9/STIMPY A. thaliana известен прежде всего как регулятор эмбрионального развития, однако у ряда видов его ортологи являются важными участниками развития соцветий. WOX9 регулирует прохождение ранних стадий эмбриогенеза. Данные об экспрессии WOX9 в эмбриогенезе отчасти противоречивы, однако известно, что он начинает экспрессироваться после первого деления зиготы либо в базальной, либо в обеих клетках, затем в гипофизе и в самом эмбрионе, а в дальнейшем – в наружных слоях зародыша и в примордиях семядолей [1, 187]. Интересно, что в эмбрионах с нарушением функции MP или BODENLOS (гена репрессора ауксинового ответа из группы Aux/IAA) не наблюдается сдвиг экспрессии WOX9 из гипофизы в собственно эмбрион [1]. Различные мутации в гене WOX9 приводят к нарушению эмбриогенеза: недоразвитию базальной части зародыша, гипокотиля и семядолей и другим дефектам, и/или остановке развития на глобулярной стадии, или раньше, снижению частоты клеточных делений [187, 212]. Однако согласно другому исследованию, растения с мутацией в WOX9 не имеют дефектов в развитии эмбрионов [185]. WOX8 – близкий гомолог WOX9, и его экспрессия под промотором WOX9 способна комплементировать мутацию в WOX9 [187]. У части двойных мутантов с предполагаемой потерей функции генов WOX8 и 9 наблюдаются нарушения в делениях клеток апикального и/или базального домена и остановка развития до формирования семядолей. У таких эмбрионов нарушены домены активности промоторов генов-маркеров различных частей зародыша, в том числе WOX8, WOX2, WOX5 и ZWILLE [185, 187]. Кроме того, для этих эмбрионов характерно нарушение экспрессии PIN1 и, вероятно, вследствие этого отсутствие максимумов ауксина в зародыше [185, 187]. Эктопическая активность WOX2 в эмбрионе и суспензоре способна частично восстановить нормальный фенотип эмбрионов данных мутантов [185]. Также потеря функции DWARF TILLER1 (DWT1) и DWT-LIKE2 (DWL2) – двух из трех ортологов WOX8/9 у O. sativa – приводит к летальности эмбрионов [213]. Гены группы WOX9 участвуют также и в СЭ: повышенный уровень экспрессии ортологов WOX9 в ходе СЭ обнаружен у M. truncatula [214], P. abies [16] и других видов. Сверхэкспрессия MtWOX9-1 стимулирует СЭ у M. truncatula [215], а подавление экспрессии PaWOX8/9 у P. abies приводит к нарушению морфологии соматических эмбрионов и развитию на их основе вторичных соматических эмбрионов [216].
Вскоре после прорастания экспрессия WOX9 наблюдается в ПАМ и примордиях листьев, а затем в меристемах цветков и развивающихся органах цветка на начальных стадиях роста, а также в эпидермисе плаценты [1, 212], в растущей перегородке плода и в верхней части КАМ [212]. Одна из мутаций в WOX9 приводит к нарушению развития ПАМ: через несколько дней после прорастания она полностью дифференцируется. WOX9 важен для поддержания экспрессии WUS и CLV3 после прорастания. Тем не менее, функции WOX9 не ограничиваются, по-видимому, активацией WUS, поскольку после прекращения функционирования ПАМ мутанты с нарушением функции WOX9 не формируют ни листьев, ни вторичных побегов, в отличие от мутантов с потерей функции WUS. Интересно, что при проращивании на среде с сахарозой, которая стимулирует деление клеток, такие мутанты развиваются практически нормально. После перехода ПАМ в стадию репродуктивного роста сахароза перестает быть необходимой. WOX9 не экспрессируется в меристеме соцветия и, по-видимому, не требуется для ее функционирования [212]. Обнаружено, что по меньшей мере одной из непосредственных мишеней WOX9 является ген циклина CYCP2;1, регулирующего переход G2/M клеточного цикла. Сахароза также способна активировать CYCP2;1 [217].
Ортологи WOX9 у пасленовых участвуют в регуляции развития соцветия. Так, ТФ EVERGREEN (EVG) P. hybrida – гомолог WOX9 и WOX8 – отвечает за архитектуру соцветия. P. hybrida имеет цимозное соцветие: в ходе его развития апикальная меристема делится на две: латеральную и меристему цветка. Меристема цветка образует цветок, а латеральная продолжает расти и занимает место апикальной. Затем этот цикл повторяется, и развивается зигзагообразное соцветие. EVG экспрессируется в меристеме соцветия, но не в меристеме цветка. У растений с мутацией в гене EVG меристемы соцветия не разделяются и продолжают формировать единый стебель, хотя и утолщенный. Через несколько узлов такие меристемы разделяются на несколько ветвей, и цикл повторяется. Кроме того, ни одна из этих меристем не может сформировать цветок [218, 219]. Согласно результатам генетического анализа, в норме EVG тем или иным способом активирует экспрессию DOUBLE TOP (DOT), гомолога UFO A. thaliana, белковый продукт которого, взаимодействуя с гомологом LEAFY – белком ALF (ABERRANT LEAF AND FLOWER), обеспечивает формирование цветков [218]. Также ортолог WOX9 у S. lycopersicum – ген COMPOUND INFLORESCENSE (S) – участвует в регуляции ветвления соцветия; мутации в этом гене или его промоторе могут приводить к возникновению сильно разветвленного соцветия [45, 220], что связано с замедленным переходом вегетативной меристемы в меристему соцветия и замедленным переходом меристемы соцветия в меристему цветка, в результате чего рядом с ними успевает сформироваться больше новых вегетативных меристем и меристем соцветия, чем в норме [221]. Как и EVG, S активирует ортолог UFO, ген ANANTHA, в меристеме цветка [220]. Сходные взаимоотношения и функции выявлены у ортологов данных генов Capsicum annuum [222].
По меньшей мере один из ортологов WOX8 и 9 O. sativa, DWT1, участвует в регуляции баланса между ростом боковых побегов и основного побега. В норме эти побеги имеют одну высоту, однако потеря функции DWT1 (мутация dwt1), не влияя на высоту основного побега, приводит к уменьшению длины боковых побегов. DWT1 экспрессируется в различных частях растения, включая эмбрион, кончик корня и молодые метелки, но не в удлиняющихся междоузлиях, там также не детектируется его белковый продукт. Вероятно, действие этого ТФ приводит к возникновению пока не идентифицированного мобильного сигнала, влияющего на удлинение междоузлий [223]. Среди генов, экспрессия которых изменена в междоузлиях растений с потерей функции DWT1, много генов, связанных с цитоскелетом и делением клеток, а также с метаболизмом и сигнальными путями цитокининов. Также в междоузлиях мутантов dwt1 O. sativa повышен уровень экспрессии гена, кодирующего фермент OsGIBBERELLIN 2-OXIDASE 1 (OsGA2OX1), инактивирующий гиббереллины, снижен уровень экспрессии гена, участвующего в биосинтезе гиббереллинов (OsENT-COPALYL DIPHOSPHATE SYNTHETASE 1), и уменьшена интенсивность ответа на экзогенный гиббереллин [223]. DWT1 и его близкий гомолог DWT-LIKE2 (DWL2) способны к взаимодействию с OsPIP5K1, ферментом, вовлеченным в синтез важнейшего вторичного посредника – фосфатидилинозитол-4,5-бисфосфата. Потеря функции OsPIP5K1 приводит к развитию растений с укороченными основным и боковыми побегами, а сверхэкспрессия OsPIP5K1 частично компенсирует дефекты мутантов dwt1, увеличивая длину боковых побегов, возможно, за счет взаимодействия OsPIP5K1 с DWL2 [213].
WOX11 и 12
WOX11 и его близкий гомолог WOX12 известны в первую очередь как регуляторы каллусообразования и развития придаточных корней, т.е. корней, которые возникают из некорневых органов, а также при регенерации in vitro или после поранения. У растений с мутациями в гене WOX11 и/или 12 придаточные корни на листовых эксплантах образуются медленнее, а их количество снижено. Сверхэкспрессия WOX11 приводит к усиленному формированию придаточных корней и каллуса у эксплантов на среде без гормонов и к усилению каллусообразования на среде CIM [224]. При регенерации корней на безгормональной среде из листовых эксплантов активность промоторов WOX11 и 12 наблюдается на ранних сроках, в прокамбии центральной жилки листа вблизи места среза, а затем в клетках-основательницах придаточного корня. Затем в растущем примордии придаточного корня, где промоторы генов WOX11 и 12 уже не активны, начинают работать промоторы WOX5/7 [224, 225]. По своей природе и набору экспрессирующихся генов каллус сходен с примордием корня, и при формировании каллуса из листовых эксплантов промотор WOX11 активен также в центральной жилке, маркируя формирование клеток-основательниц, а в уже образующемся каллусе промотор WOX11 не активен, но начинает функционировать промотор WOX5 [224].
WOX11/12 непосредственно активируют экспрессию WOX5/7, которые, согласно анализу мутантов, также важны для формирования придаточных корней [225]. Также WOX11 запускает экспрессию стимуляторов каллусообразования и формирования придаточных и боковых корней – генов ТФ LBD (LATERAL ORGAN BOUNDARIES DOMAIN), в частности LBD16 и 29, причем показано, что LBD16 прямо активируется WOX11 [224, 226]. Ауксин и полярный транспорт ауксина важны для активации WOX11 и запуска экспрессии WOX5/7 и, в конечном счете, для регенерации корней [224, 225]. Тем не менее, сверхэкспрессия WOX11 способна частично восстанавливать способность к регенерации при подавлении синтеза ауксина [227]. Напротив, этилен подавляет регенерацию корней, а WOX11 и 5 репрессируются этилен-чувствительным ТФ ETHYLENE-INSENSITIVE3 (EIN3). Интересно, что чем старше листовой эксплант, тем сильнее в нем экспрессируется EIN3 и слабее – WOX11, и тем более затруднена регенерация корней [228]. Также обнаружен один из механизмов эпигенетической регуляции WOX11 при каллусогенезе: атипичный вариант гистона H3 – H3.15 (без остатка лизина в позиции 27) связывается с локусом WOX11 и активирует его экспрессию, вероятно, за счет уменьшения количества репрессирующих меток H3K27me3 [229]. Сходная схема взаимодействия ортологов WOX11/12 и WOX5 в каллусообразовании на среде CIM обнаружена у O. sativa [230]. Экспрессия ортологов WOX5 и 11 при каллусогенезе выявлена у Z. mays, и у гибрида P. davidiana × P. bollena [230], а участие ортологов WOX11 в образовании придаточных корней показано у различных видов и гибридов Populus [231–233]. Как и у семенных растений, у папоротника Ceratopteris richardii обнаружен путь формирования придаточных корней, при котором факторы ауксинового ответа активируют ген промежуточной клады WOX CrWOXA, который активирует экспрессию гена клады WUS – СrWUL. Таким образом, этот путь формирования придаточных корней (ауксин–ген WOX промежуточной клады–ген WOX клады WUS) консервативен у папоротников и семенных растений [234]. CrWOXA экспрессируется и при образовании боковых корней [4]. Гены группы WOX11 участвуют и в других вариантах регенерации, таких как побегообразование и СЭ. Так, показано положительное влияние PtWOX11 на регенерацию побегов у P. alba × P. glandulosa [233]. Повышенный уровень экспрессии ортолога WOX11 выявлен также в эмбриогенных каллусах M. truncatula [214] и в соматических эмбрионах и неэмбриогенных каллусах V. vinifera [119].
Помимо участия в процессах регенерации in vitro, ТФ группы WOX11/12 участвуют в развитии растения in vivo при формировании придаточных корней. У A. thaliana WOX11/12 не влияют на формирование боковых корней, которые в норме формируются за счет активации ARF7/19, и промотор WOX11 не активируется в ходе образования бокового корня [226]. Однако при выращивании проростков в почве промотор WOX11 активируется в различных участках корня, и изменение его функции влияет на количество вторичных, третичных и четвертичных корней. Вероятной причиной таких различий могут быть повреждения, которые корни получают при росте в почве и которые стимулируют образование корней, сходных с придаточными. В формировании как боковых, так и придаточных корней участвует ТФ LBD16. WOX11 за счет прямой активации LBD16 стимулирует развитие придаточных корней и корней в месте повреждения, а ARF7/19 стимулируют LBD16 в случае развития боковых корней без повреждения корня [226].
У O. sativa ортолог WOX11 выполняет более многочисленные функции. Растения с потерей функции OsWOX11 практически не формируют придаточные корни, кроме того, их первичный корень и ПАМ тоже недоразвиты, и они не доживают до взрослого состояния. OsWOX11 экспрессируется в КАМ, ПАМ и стебле, а также в примордиях придаточных корней, начиная с ранних стадий инициации, и в меристеме зрелых придаточных корней. Сверхэкспрессия OsWOX11 приводит к развитию большого количества придаточных и боковых корней. Ауксины повышают уровень экспрессии OsWOX11, а цитокинины снижают. OsWOX11 необходим для стимулируемого ауксином развития придаточных корней у риса [235]. При этом OsWOX11, вероятно, положительно влияет на передачу цитокининового сигнала, являясь прямым репрессором гена OsRR2, кодирующего репрессор цитокининового ответа – ARR типа A [236]. Также показано взаимодействие OsWOX11 с ТФ OsERF3 из семейства AP2/ERF, стимулятором развития придаточных корней и роста главного корня. Уровень экспрессии ERF3 уменьшен в растениях с потерей функции OsWOX11, однако ERF3 способен регулировать развитие корней по пути, не связанному с OsWOX11. OsERF3 также связывается с OsRR2 (но не в том регионе, в котором с ним связывается OsWOX11), однако он не подавляет, а стимулирует экспрессию OsRR2. При этом OsERF3 способствует взаимодействию OsWOX11 со своим сайтом связывания в локусе OsRR2. OsRR2 стимулирует заложение придаточных корней, однако его влияние на длину корней неоднозначно, что объясняет разнонаправленное влияние на него OsWOX11 и OsERF3 [237]. OsWOX11 также связывается с комплексом, включающим вспомогательный белок OsADA2 и гистон-ацетилтрансферазу OsGCN5 для активации экспрессии генов-мишеней (с помощью ацетилирования гистонов) и стимуляции развития придаточных корней, активируя в том числе гены, связанные с транспортом ауксина, в частности OsPIN9, а также с метаболизмом и построением клеточной стенки [238]. Ген ТФ DEGENERATED HULL 1 (DH1)/OsLOB16, близкий гомолог LBD16 A. thaliana, представляет собой мишень OsWOX11 [239]. Помимо регуляции образования придаточных корней, OsWOX11 регулирует поглощение корнями минеральных веществ и воды. Промотор OsWOX11 активен в эпидермисе, в кончиках и зоне роста корня [236], а сам OsWOX11 стимулирует образование и рост корневых волосков. Уровень экспрессии данного гена повышается в ответ на водный стресс, а растения с мутацией в OsWOX11 более чувствительны к засухе [240]. Кроме того, среди непосредственных мишеней OsWOX11 обнаружены гены, кодирующие предполагаемый ТФ OsABIOTIC STRESS RESPONSIVE3 (OsASR3), участвующий в ответе на дефицит воды и холодовой стресс, и OsFRD3-LIKE PROTEIN1 (OsFRDL1) – переносчик цитрата, необходимый для транспорта ионов железа в растении, а реакция ряда генов на дефицит воды зависит от присутствия функционального OsWOX11 [239]. Интересно, что экспрессия OsWOX11, направляемая промотором, активирующимся в корнях при дефиците калия, приводит к стимуляции развития корневой системы растений как в нормальных условиях, так и в условиях дефицита калия [241].
Как сказано выше, потеря функции OsWOX11 приводит также к замедлению роста побега, уменьшению размера ПАМ и соцветия. OsWOX11 способен к взаимодействию с деметилазой гистонов JMJ705, снимающей метку H3K27me3, которая также стимулирует рост побега и влияет на размер ПАМ. OsWOX11 и JMJ705 совместно регулируют множество генов, связанных, в частности, с энергообменом, фотосинтезом, развитием хлоропластов, ответом на стресс и формированием побега, например, OSH15 из семейства KNOX [242]. Также OsWOX11 вместе с другим геном из группы WOX11/12, OsWOX6, участвует в регуляции гравитропического ответа. Уровень их экспрессии увеличивается в ответ на гравистимуляцию проростков и в ответ на обработку ауксином, при гравистимуляции уровень их экспрессии повышен в нижней части проростка и снижен в верхней. Растения с потерей функции OsWOX6 и 11 слабее реагируют на гравистимуляцию и имеют больший угол наклона побегов. В условиях гравистимуляции гены OsWOX6 и 11 опосредованно регулируются ТФ HEAT STRESS TRANSCRIPTION FACTOR 2D (HSFA2D), который, посредством активации экспрессии гена ТФ LA1 (LAZY1) стимулирует асимметричное распределение ауксина, что приводит к асимметричному паттерну экспрессии OsWOX6 и 11 и способствует ответу на гравистимуляцию [243].
ДРЕВНЯЯ ВЕТВЬ (T1)
Представители древней ветви семейства WOX обнаружены в геноме не только сосудистых растений, но также мхов (Physcomitrella patens) и даже одноклеточных водорослей (Ostreococcus tauri) [6]. У A. thaliana в состав данной ветви входят три гена: WOX10, 13 и 14. WOX10, более сходный с WOX14, чем с WOX13, предположительно, является псевдогеном, поскольку его экспрессия не была детектирована [244], а WOX13 и 14 регулируют развитие цветков, плодов и проводящих тканей. Члены древней клады у других видов характеризуются весьма разнообразными паттернами экспрессии. Так, у P. ginseng PgWOX13a экспрессируется в ксилемных лучах, паренхиме и камбии главного корня, а PgWOX13b – в кортексе [245], ген AaWOX Araucaria angustifolia активен в зародышах и в проростках [246], у G. hirsutum GhWOX13a_Dt, GhWOX13b_At и GhWOX13b_Dt повышают уровень экспрессии во время развития семенных волокон [247], а OsWOX13 O. sativa, вероятно, участвует в ответе на засуху и в регуляции сроков цветения [248]. Мы рассмотрим наиболее изученных представителей древней клады.
WOX13
WOX13 A. thaliana изучен в первую очередь как регулятор развития плода. Потеря его функции приводит к уменьшению размеров медиальной части гинецея (перегородки), расширению кромки створок и к ускорению растрескивания плодов. Напротив, сверхэкспрессия WOX13 ведет к тому, что гинецей дольше остается открытым, перегородка расширяется, а растрескивание плода не происходит или происходит намного позднее. WOX13 регулирует развитие перегородки в том числе за счет прямого или опосредованного подавления экспрессии маркеров латеральных областей гинецея – генов ТФ семейства YABBY FILAMENTOUS FLOWER и ТФ с цинковыми пальцами JAGGED [249]. Помимо гинецея, активность промотора WOX13 детектирована в закладывающихся боковых корнях, листовых примордиях и цветках, а также в кончике корня, проводящих тканях корня и листа и в ПАМ, а мутация в WOX13 приводит также к уменьшению количества боковых корней и к более раннему цветению [244, 249].
У мха P. patens гены WOX представлены только членами древней клады, близкими к WOX13: PpWOX13LA, B и С [250]. Активность промоторов PpWOX13LA и B повышена в апикальных стволовых клетках хлоронемы и каулонемы, а также в яйцеклетке и зиготе. Потеря функции обоих генов приводит к уменьшению количества сформированных спорангиев и к остановке развития зигот, а также к нарушению регенерации вследствие сниженной интенсивности клеточных делений и подавления роста клеток [250].
Партнеры WOX13 или его ортологов in vivo еще не выявлены, однако показана способность ТФ PtrWOX13c P. trichocarpa формировать тример с ТФ PtrDRIF1 (PtrDIVARICATA AND RADIALIS INTERACTING FACTOR1) и PtrRADIALIS1 с доменом MYB/SANT; при этом гены, кодирующие PtrWOX13c и данные ТФ, экспрессируются в проводящих тканях [251].
WOX14
WOX14 регулирует развитие проводящих тканей и рост растений в целом. Потеря функции WOX14 приводит к карликовости растений, задержке цветения и гиполигнификации сосудов, однако эти дефекты компенсируются при добавлении гиббереллинов. WOX14 стимулирует экспрессию GA3ox1 и подавляет экспрессию GA2ox1 – генов, кодирующих ферменты биосинтеза и инактивации гиббереллинов соответственно [199]. WOX14 вместе с WOX4 стимулирует пролиферацию клеток проводящих тканей. Сосудистые пучки двойных мутантов с нарушением функции генов WOX4 и 14 (wox4-1 wox14-1) содержат меньше клеток, чем одиночные мутанты wox4-1 [198]. Обнаружена связь WOX14 с другими ТФ, стимулирующими деление клеток проводящих тканей – TMO6 и LBD4. WOX14, вероятно, связываясь с его локусом, стимулирует экспрессию TMO6, белковый продукт которого, в свою очередь, вместе с WOX14 стимулирует экспрессию LBD4 [252]. Экспрессия WOX14, как и WOX4, стимулируется CLE-пептидом TDIF. При этом обработка этим пептидом растений дикого типа вызывает активацию LBD4 и TMO6, тогда как у мутантов wox4-1 wox14-1 или у растений с мутацией в гене PXY этого не происходит [252]. Промотор WOX14 активен в примордиях боковых корней, в перицикле и проводящей системе корня, а также на ранних стадиях развития тычинок. Потеря функции WOX14 приводит к подавлению развития боковых корней и увеличению количества придаточных корней, а также к недоразвитию тычинок и частичной стерильности [244]. Также WOX14 важен для регенерации побегов in vitro в системе CIM–SIM, а при образовании каллусов его экспрессия активируется гистон-ацетилтрансферазой HAG1 [149].
ЗАКЛЮЧЕНИЕ
Гены WOX участвуют в самых разнообразных аспектах развития растения, включая регуляцию функционирования меристем, развитие боковых органов, размножение, эмбриогенез и многое другое (рис. 4). В табл. 1S (см. Приложение на сайте http://www.molecbio.ru/downloads/2020/3/ supp_Tvorogova_rus.pdf) приведены все описанные в обзоре мишени и партнеры WOX, а также регуляторы кодирующих их генов.
Таким образом, белки WOX, несмотря на разнообразие их функций, зачастую участвуют в сходных регуляторных модулях. Так, некоторые гены WOX, включая гены современной (WUS, WOX4 и 5) [22, 131, 193], промежуточной (WOX8) [209] и древней (WOX14) [252] ветвей, регулируются с помощью пептидов семейства CLE. Экспрессия генов WUS и WOX5 репрессируется одним и тем же белком BARD1/ROW1 [78, 141], и большая часть WOX тесно взаимодействует с ауксинами и цитокининами. В целом, ТФ WOX обычно стимулируют пролиферацию клеток и регенерацию. Например, сверхэкспрессия генов современной клады (PtoWUSa, PtoWOX4a, PtoWOX5a), как и генов промежуточной клады (PtoWOX11/12a) стимулирует регенерацию корней из черенков гибридного тополя (P. alba × P. glandulosa) [231]. Тем не менее обнаружены существенные различия между ТФ WOX различных клад, особенно выражено отличие современной клады от двух других. Так, комплементационный анализ мутантов wus-1 показывает, что функции WUS могут в той или иной степени замещать все гены WOX, входящие в кладу WUS, кроме WOX4, но не гены из промежуточной и древней клады [90]. Гены WOX A. thaliana, входящие в кладу WUS, за исключением WOX7, но не гены промежуточной или древней клады, способны частично или полностью комплементировать мутацию lam1 в ортологе WOX1 N. tabacum [253]. Изучение ТФ WOX не только представляет теоретический интерес, но и открывает новые возможности в биотехнологии, такие как разработка эффективных протоколов трансформации и регенерации злаков [118] и повышение урожайности сельскохозяйственных растений [161, 241]. Несмотря на активное изучение семейства WOX, детально определены далеко не все механизмы работы этих генов и генные сети, в которые они вовлечены. Табл. 2S (см. Приложение на сайте http://www.molecbio.ru/downloads/2020/3/ supp_Tvorogova_rus.pdf), в которой сгруппированы регуляторы, мишени и партнеры каждого из генов WOX A. thaliana, наглядно показывает, механизмы работы каких генов WOX еще предстоит подробно исследовать. Таким образом, нас, вероятно, ждет еще множество исследований, посвященных семейству WOX.
Работа выполнена при финансовой поддержке РФФИ в рамках научного проекта № 19-14-50209.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Haecker A., Gross-Hardt R., Geiges B., Sarkar A., Breuninger H., Herrmann M., Laux T. (2004) Expression dynamics of WOX genes mark cell fate decisions during early embryonic patterning in Arabidopsis thaliana. Development. 131, 657–668.
Nardmann J., Reisewitz P., Werr W. (2009) Discrete shoot and root stem cell-promoting WUS/WOX5 functions are an evolutionary innovation of angiosperms. Mol. Biol. Evol. 26, 1745–1755.
Wu C.-C., Li F.-W., Kramer E.M. (2019) Large-scale phylogenomic analysis suggests three ancient superclades of the WUSCHEL-RELATED HOMEOBOX transcription factor family in plants. PLoS One. 14, https://doi.org/10.1371/journal.pone.0223521
Nardmann J., Werr W. (2012) The invention of WUS-like stem cell-promoting functions in plants predates leptosporangiate ferns. Plant Mol. Biol. 78, 123–134.
Lian G., Ding Z., Wang Q., Zhang D., Xu J. (2014) Origins and evolution of WUSCHEL-related homeobox protein family in plant kingdom. Sci. World J. 2014, https://doi.org/10.1155/2014/534140
Segatto A.L.A., Thompson C.E., Freitas L.B. (2016) Molecular evolution analysis of WUSCHEL-related homeobox transcription factor family reveals functional divergence among clades in the homeobox region. Dev. Genes Evol. 226, 259–268.
Laux T., Mayer K.F., Berger J., Jürgens G. (1996) The WUSCHEL gene is required for shoot and floral meristem integrity in Arabidopsis. Development. 122, 87–96.
Yadav R.K., Tavakkoli M., Reddy G.V. (2010) WUSCHEL mediates stem cell homeostasis by regulating stem cell number and patterns of cell division and differentiation of stem cell progenitors. Development. 137, 3581–3589.
Wang J., Tian C., Zhang C., Shi B., Cao X., Zhang T.-Q., Zhao Z., Wang J.-W., Jiao Y. (2017) Cytokinin signaling activates WUSCHEL expression during axillary meristem initiation. Plant Cell. 29, 1373–1387.
Gallois J.-L., Nora F.R., Mizukami Y., Sablowski R. (2004) WUSCHEL induces shoot stem cell activity and developmental plasticity in the root meristem. Genes Dev. 18, 375–380.
Zhang T.-Q., Lian H., Zhou C.-M., Xu L., Jiao Y., Wang J.-W. (2017) A two-step model for de novo activation of WUSCHEL during plant shoot regeneration. Plant Cell. 29, 1073–1087.
Mayer K.F., Schoof H., Haecker A., Lenhard M., Jürgens G., Laux T. (1998) Role of WUSCHEL in regulating stem cell fate in the Arabidopsis shoot meristem. Cell. 95, 805–815.
Xin W., Wang Z., Liang Y., Wang Y., Hu Y. (2017) Dynamic expression reveals a two-step patterning of WUS and CLV3 during axillary shoot meristem formation in Arabidopsis. J. Plant Physiol. 214, 1–6.
Yadav R.K., Perales M., Gruel J., Girke T., Jönsson H., Reddy G.V. (2011) WUSCHEL protein movement mediates stem cell homeostasis in the Arabidopsis shoot apex. Genes Dev. 25, 2025–2030.
Daum G., Medzihradszky A., Suzaki T., Lohmann J.U. (2014) A mechanistic framework for noncell autonomous stem cell induction in Arabidopsis. Proc. Natl. Acad. Sci. USA. 111, 14619–14624.
Hedman H., Zhu T., von Arnold S., Sohlberg J.J. (2013) Analysis of the WUSCHEL-RELATED HOMEOBOX gene family in the conifer picea abies reveals extensive conservation as well as dynamic patterns. BMC Plant Biol. 13, 89.
Meng Y., Liu H., Wang H., Liu Y., Zhu B., Wang Z., Hou Y., Zhang P., Wen J., Yang H., Mysore K.S., Chen J., Tadege M., Niu L., Lin H. (2019) HEADLESS, a WUSCHEL homolog, uncovers novel aspects of shoot meristem regulation and leaf blade development in Medicago truncatula. J. Exp. Bot. 70, 149–163.
Wang H., Xu Y., Hong L., Zhang X., Wang X., Zhang J., Ding Z., Meng Z., Wang Z.-Y., Long R., Yang Q., Kong F., Han L., Zhou C. (2019) HEADLESS regulates auxin response and compound leaf morphogenesis in Medicago truncatula. Front. Plant Sci. 10, 1024.https://doi.org/10.3389/fpls.2019.01024
Nardmann J., Werr W. (2006) The shoot stem cell niche in angiosperms: expression patterns of WUS orthologues in rice and maize imply major modifications in the course of mono- and dicot evolution. Mol. Biol. Evol. 23, 2492–2504.
Tanaka W., Ohmori Y., Ushijima T., Matsusaka H., Matsushita T., Kumamaru T., Kawano S., Hirano H.-Y. (2015) Axillary meristem formation in rice requires the WUSCHEL ortholog TILLERS ABSENT1. Plant Cell. 27, 1173–1184.
Suzuki C., Tanaka W., Hirano H.-Y. (2019) Transcriptional corepressor ASP1 and CLV-like signaling regulate meristem maintenance in rice. Plant Physiol. 180, 1520–1534.
Schoof H., Lenhard M., Haecker A., Mayer K.F.X., Jürgens G., Laux T. (2000) The stem cell population of Arabidopsis shoot meristems is maintained by a regulatory loop between the CLAVATA and WUSCHEL Genes. Cell. 100, 635–644.
Brand U., Fletcher J.C., Hobe M., Meyerowitz E.M., Simon R. (2000) Dependence of stem cell fate in Arabidopsis on a feedback loop regulated by CLV3 activity. Science. 289, 617–619.
Полюшкевич Л.О., Ганчева М.С., Додуева И.Е., Лутова Л.А. (2020) Рецепторы пептидов CLE у растений. Физиология растений. 67, 3–19.
Hu C., Zhu Y., Cui Y., Cheng K., Liang W., Wei Z., Zhu M., Yin H., Zeng L., Xiao Y., Lv M., Yi J., Hou S., He K., Li J., Gou X. (2018) A group of receptor kinases are essential for CLAVATA signalling to maintain stem cell homeostasis. Nat. Plants. 4, 205–211.
Yu L.P., Simon E.J., Trotochaud A.E., Clark S.E. (2000) POLTERGEIST functions to regulate meristem development downstream of the CLAVATA loci. Development. 127, 1661–1670.
Song S.-K., Lee M.M., Clark S.E. (2006) POL and PLL1 phosphatases are CLAVATA1 signaling intermediates required for Arabidopsis shoot and floral stem cells. Development. 133, 4691–4698.
Gagne J.M., Gish L.A., Clark S.E. (2010) The role of the acyl modification, palmitoylation, in Arabidopsis stem cell regulation. Plant Signal. Behav. 5, 1048–1051.
Bommert P., Je B.I., Goldshmidt A., Jackson D. (2013) The maize Gα gene COMPACT PLANT2 functions in CLAVATA signalling to control shoot meristem size. Nature. 502, 555–558.
Lee H., Jun Y.S., Cha O.-K., Sheen J. (2019) Mitogen-activated protein kinases MPK3 and MPK6 are required for stem cell maintenance in the Arabidopsis shoot apical meristem. Plant Cell Rep. 38, 311–319.
Chou H., Zhu Y., Ma Y., Berkowitz G.A. (2016) The CLAVATA signaling pathway mediating stem cell fate in shoot meristems requires Ca2+ as a secondary cytosolic messenger. Plant J. Cell Mol. Biol. 85, 494–506.
Che G., Gu R., Zhao J., Liu X., Song X., Zi H., Cheng Z., Shen J., Wang Z., Liu R., Yan L., Weng Y., Zhang X. (2020) Gene regulatory network controlling carpel number variation in cucumber. Development. 147, dev184788. https://doi.org/10.1242/dev.184788
Perales M., Rodriguez K., Snipes S., Yadav R.K., Diaz-Mendoza M., Reddy G.V. (2016) Threshold-dependent transcriptional discrimination underlies stem cell homeostasis. Proc. Natl. Acad. Sci. USA. 113, E6298–E6306.
Zhou Y., Yan A., Han H., Li T., Geng Y., Liu X., Meyerowitz E.M. (2018) HAIRY MERISTEM with WUSCHEL confines CLAVATA3 expression to the outer apical meristem layers. Science. 361, 502–506.
Busch W., Miotk A., Ariel F.D., Zhao Z., Forner J., Daum G., Suzaki T., Schuster C., Schultheiss S.J., Leibfried A., Haubeiss S., Ha N., Chan R.L., Lohmann J.U. (2010) Transcriptional control of a plant stem cell niche. Dev. Cell. 18, 849–861.
Theißen G., Melzer R., Rümpler F. (2016) MADS-domain transcription factors and the floral quartet model of flower development: linking plant development and evolution. Development. 143, 3259–3271.
Lenhard M., Bohnert A., Jürgens G., Laux T. (2001) Termination of stem cell maintenance in Arabidopsis floral meristems by interactions between WUSCHEL and AGAMOUS. Cell. 105, 805–814.
Lohmann J.U., Hong R.L., Hobe M., Busch M.A., Parcy F., Simon R., Weigel D. (2001) A molecular link between stem cell regulation and floral patterning in Arabidopsis. Cell. 105, 793–803.
Sun B., Xu Y., Ng K.-H., Ito T. (2009) A timing mechanism for stem cell maintenance and differentiation in the Arabidopsis floral meristem. Genes Dev. 23, 1791–1804.
Bollier N., Sicard A., Leblond J., Latrasse D., Gonzalez N., Gévaudant F., Benhamed M., Raynaud C., Lenhard M., Chevalier C., Hernould M., Delmas F. (2018) At-MINI ZINC FINGER2 and Sl-INHIBITOR OF MERISTEM ACTIVITY, a conserved missing link in the regulation of floral meristem termination in Arabidopsis and tomato. Plant Cell. 30, 83–100.
Sun B., Zhou Y., Cai J., Shang E., Yamaguchi N., Xiao J., Looi L.-S., Wee W.-Y., Gao X., Wagner D., Ito T. (2019) Integration of transcriptional repression and Polycomb-mediated silencing of WUSCHEL in floral meristems. Plant Cell. 31, 1488–1505.
Liu X., Kim Y.J., Müller R., Yumul R.E., Liu C., Pan Y., Cao X., Goodrich J., Chen X. (2011) AGAMOUS terminates floral stem cell maintenance in Arabidopsis by directly repressing WUSCHEL through recruitment of Polycomb group proteins. Plant Cell. 23, 3654–3670.
Guo L., Cao X., Liu Y., Li J., Li Y., Li D., Zhang K., Gao C., Dong A., Liu X. (2018) A chromatin loop represses WUSCHEL expression in Arabidopsis. Plant J. Cell. Mol. Biol. 94, 1083–1097.
van der Knaap E., Chakrabarti M., Chu Y.H., Clevenger J.P., Illa-Berenguer E., Huang Z., Keyhaninejad N., Mu Q., Sun L., Wang Y., Wu S. (2014) What lies beyond the eye: the molecular mechanisms regulating tomato fruit weight and shape. Front. Plant Sci. 5, 227.
Rodríguez-Leal D., Lemmon Z.H., Man J., Bartlett M.E., Lippman Z.B. (2017) Engineering quantitative trait variation for crop improvement by genome editing. Cell. 171, 470–480.e8.
Liu X., Gao L., Dinh T.T., Shi T., Li D., Wang R., Guo L., Xiao L., Chen X. (2014) DNA topoisomerase I affects Polycomb group protein-mediated epigenetic regulation and plant development by altering nucleosome distribution in Arabidopsis. Plant Cell. 26, 2803–2817.
Liu X., Dinh T.T., Li D., Shi B., Li Y., Cao X., Guo L., Pan Y., Jiao Y., Chen X. (2014) AUXIN RESPONSE FACTOR 3 integrates the functions of AGAMOUS and APETALA2 in floral meristem determinacy. Plant J. Cell Mol. Biol. 80, 629–641.
Ferrario S., Shchennikova A.V., Franken J., Immink R.G.H., Angenent G.C. (2006) Control of floral meristem determinacy in petunia by MADS-box transcription factors. Plant Physiol. 140, 890–898.
Lu H., Klocko A.L., Brunner A.M., Ma C., Magnuson A.C., Howe G.T., An X., Strauss S.H. (2019) RNA interference suppression of AGAMOUS and SEEDSTICK alters floral organ identity and impairs floral organ determinacy, ovule differentiation, and seed-hair development in Populus. New Phytol. 222, 923–937.
Pelaz S., Ditta G.S., Baumann E., Wisman E., Yanofsky M.F. (2000) B and C floral organ identity functions require SEPALLATA MADS-box genes. Nature. 405, 200–203.
Carles C.C., Lertpiriyapong K., Reville K., Fletcher J.C. (2004) The ULTRAPETALA1 gene functions early in Arabidopsis development to restrict shoot apical meristem activity and acts through WUSCHEL to regulate floral meristem determinacy. Genetics. 167, 1893–1903.
Carles C.C., Choffnes-Inada D., Reville K., Lertpiriyapong K., Fletcher J.C. (2005) ULTRAPETALA1 encodes a SAND domain putative transcriptional regulator that controls shoot and floral meristem activity in Arabidopsis. Development. 132, 897–911.
Moreau F., Thévenon E., Blanvillain R., Lopez-Vidriero I., Franco-Zorrilla J.M., Dumas R., Parcy F., Morel P., Trehin C., Carles C.C. (2016) The Myb-domain protein ULTRAPETALA1 INTERACTING FACTOR 1 controls floral meristem activities in Arabidopsis. Development. 143, 1108–1119.
Endrizzi K., Moussian B., Haecker A., Levin J.Z., Laux T. (1996) The SHOOT MERISTEMLESS gene is required for maintenance of undifferentiated cells in Arabidopsis shoot and floral meristems and acts at a different regulatory level than the meristem genes WUSCHEL and ZWILLE. Plant J. 10, 967–979.
Scofield S., Dewitte W., Murray J.A. (2014) STM sustains stem cell function in the Arabidopsis shoot apical meristem and controls KNOX gene expression independently of the transcriptional repressor AS1. Plant Signal. Behav. 9, https://doi.org/10.4161/psb.28934
Green K.A., Prigge M.J., Katzman R.B., Clark S.E. (2005) CORONA, a member of the class III homeodomain leucine zipper gene family in Arabidopsis, regulates stem cell specification and organogenesis. Plant Cell. 17, 691–704.
Williams L., Grigg S.P., Xie M., Christensen S., Fletcher J.C. (2005) Regulation of Arabidopsis shoot apical meristem and lateral organ formation by microRNA miR166g and its AtHD-ZIP target genes. Development. 132, 3657–3668.
Lee C., Clark S.E. (2015) A WUSCHEL-independent stem cell specification pathway is repressed by PHB, PHV and CNA in Arabidopsis. PLoS One. 10, e0126006.
Yuste-Lisbona F.J., Fernández-Lozano A., Pineda B., Bretones S., Ortíz-Atienza A., García-Sogo B., Müller N.A., Angosto T., Capel J., Moreno V., Jiménez-Gómez J.M., Lozano R. (2020) ENO regulates tomato fruit size through the floral meristem development network. Proc. Natl. Acad. Sci. USA. 117, 8187–8195.
Bae S.-Y., Kim M.-H., Cho J.-S., Park E.-J., Lee H., Kim J.-H., Ko J.-H. (2020) Overexpression of Populus transcription factor PtrTALE12 increases axillary shoot development by regulating WUSCHEL expression. Tree Physiol. https://doi.org/10.1093/treephys/tpaa062
Leibfried A., To J.P.C., Busch W., Stehling S., Kehle A., Demar M., Kieber J.J., Lohmann J.U. (2005) WUSCHEL controls meristem function by direct regulation of cytokinin-inducible response regulators. Nature. 438, 1172–1175.
Chickarmane V.S., Gordon S.P., Tarr P.T., Heisler M.G., Meyerowitz E.M. (2012) Cytokinin signaling as a positional cue for patterning the apical–basal axis of the growing Arabidopsis shoot meristem. Proc. Natl. Acad. Sci. USA. 109, 4002–4007.
Gordon S.P., Chickarmane V.S., Ohno C., Meyerowitz E.M. (2009) Multiple feedback loops through cytokinin signaling control stem cell number within the Arabidopsis shoot meristem. Proc. Natl. Acad. Sci. USA. 106, 16529–16534.
Lindsay D.L., Sawhney V.K., Bonham-Smith P.C. (2006) Cytokinin-induced changes in CLAVATA1 and WUSCHEL expression temporally coincide with altered floral development in Arabidopsis. Plant Sci. 170, 1111–1117.
Snipes S.A., Rodriguez K., DeVries A.E., Miyawaki K.N., Perales M., Xie M., Reddy G.V. (2018) Cytokinin stabilizes WUSCHEL by acting on the protein domains required for nuclear enrichment and transcription. PLoS Genet. 14, e1007351.
Meng W.J., Cheng Z.J., Sang Y.L., Zhang M.M., Rong X.F., Wang Z.W., Tang Y.Y., Zhang X.S. (2017) Type-B Arabidopsis response regulators specify the shoot stem cell niche by dual regulation of WUSCHEL. Plant Cell. 29, 1357–1372.
Dai X., Liu Z., Qiao M., Li J., Li S., Xiang F. (2017) ARR12 promotes de novo shoot regeneration in Arabidopsis thaliana via activation of WUSCHEL expression. J. Integr. Plant Biol. 59, 747–758.
Xie M., Chen H., Huang L., O’Neil R.C., Shokhirev M.N., Ecker J.R. (2018) A B-ARR-mediated cytokinin transcriptional network directs hormone cross-regulation and shoot development. Nat. Commun. 9, 1604.
Zubo Y.O., Blakley I.C., Yamburenko M.V., Worthen J.M., Street I.H., Franco-Zorrilla J.M., Zhang W., Hill K., Raines T., Solano R., Kieber J.J., Loraine A.E., Schaller G.E. (2017) Cytokinin induces genome-wide binding of the type-B response regulator ARR10 to regulate growth and development in Arabidopsis. Proc. Natl. Acad. Sci. USA. 114, E5995–E6004.
Liu Z., Dai X., Li J., Liu N., Liu X., Li S., Xiang F. (2020) The type-B cytokinin response regulator ARR1 inhibits shoot regeneration in an ARR12-dependent manner in Arabidopsis. Plant Cell. 32, 2271–2291.
Ma Y., Miotk A., Šutiković Z., Ermakova O., Wenzl C., Medzihradszky A., Gaillochet C., Forner J., Utan G., Brackmann K., Galván-Ampudia C.S., Vernoux T., Greb T., Lohmann J.U. (2019) WUSCHEL acts as an auxin response rheostat to maintain apical stem cells in Arabidopsis. Nat. Commun. 10, 5093.
Cheng Z.J., Wang L., Sun W., Zhang Y., Zhou C., Su Y.H., Li W., Sun T.T., Zhao X.Y., Li X.G., Cheng Y., Zhao Y., Xie Q., Zhang X.S. (2013) Pattern of auxin and cytokinin responses for shoot meristem induction results from the regulation of cytokinin biosynthesis by auxin response factor 3. Plant Physiol. 161, 240–251.
Su Y.H., Zhao X.Y., Liu Y.B., Zhang C.L., O’Neill S.D., Zhang X.S. (2009) Auxin-induced WUS expression is essential for embryonic stem cell renewal during somatic embryogenesis in Arabidopsis. Plant J. Cell Mol. Biol. 59, 448–460.
Su Y.H., Liu Y.B., Bai B., Zhang X.S. (2015) Establishment of embryonic shoot–root axis is involved in auxin and cytokinin response during Arabidopsis somatic embryogenesis. Front. Plant Sci. 5, 792. https://doi.org/10.3389/fpls.2014.00792
Li W., Liu H., Cheng Z.J., Su Y.H., Han H.N., Zhang Y., Zhang X.S. (2011) DNA methylation and histone modifications regulate de novo shoot regeneration in Arabidopsis by modulating WUSCHEL expression and auxin signaling. PLoS Genet. 7, e1002243.
Shemer O., Landau U., Candela H., Zemach A., Eshed Williams L. (2015) Competency for shoot regeneration from Arabidopsis root explants is regulated by DNA methylation. Plant Sci. Int. J. Exp. Plant Biol. 238, 251–261.
Kwon C.S., Chen C., Wagner D. (2005) WUSCHEL is a primary target for transcriptional regulation by SPLAYED in dynamic control of stem cell fate in Arabidopsis. Genes Dev. 19, 992–1003.
Han P., Li Q., Zhu Y.-X. (2008) Mutation of Arabidopsis BARD1 causes meristem defects by failing to confine wuschel expression to the organizing center. Plant Cell. 20, 1482–1493.
Kaya H., Shibahara K.I., Taoka K.I., Iwabuchi M., Stillman B., Araki T. (2001) FASCIATA genes for chromatin assembly factor-1 in Arabidopsis maintain the cellular organization of apical meristems. Cell. 104, 131–142.
Chen D., Molitor A.M., Xu L., Shen W.-H. (2016) Arabidopsis PRC1 core component AtRING1 regulates stem cell-determining carpel development mainly through repression of class I KNOX genes. BMC Biol. 14, 112.
Goldshmidt A., Alvarez J.P., Bowman J.L., Eshed Y. (2008) Signals derived from YABBY gene activities in organ primordia regulate growth and partitioning of Arabidopsis shoot apical meristems. Plant Cell. 20, 1217–1230.
Zhao Y., Medrano L., Ohashi K., Fletcher J.C., Yu H., Sakai H., Meyerowitz E.M. (2004) HANABA TARANU is a GATA transcription factor that regulates shoot apical meristem and flower development in Arabidopsis. Plant Cell. 16, 2586–2600.
Saiga S., Furumizu C., Yokoyama R., Kurata T., Sato S., Kato T., Tabata S., Suzuki M., Komeda Y. (2008) The Arabidopsis OBERON1 and OBERON2 genes encode plant homeodomain finger proteins and are required for apical meristem maintenance. Development. 135, 1751–1759.
Zeng J., Dong Z., Wu H., Tian Z., Zhao Z. (2017) Redox regulation of plant stem cell fate. EMBO J. 36, 2844–2855.
Pfeiffer A., Janocha D., Dong Y., Medzihradszky A., Schöne S., Daum G., Suzaki T., Forner J., Langenecker T., Rempel E., Schmid M., Wirtz M., Hell R., Lohmann J.U. (2016) Integration of light and metabolic signals for stem cell activation at the shoot apical meristem. eLife. 5, e17023.
Yadav R.K., Perales M., Gruel J., Ohno C., Heisler M., Girke T., Jönsson H., Reddy G.V. (2013) Plant stem cell maintenance involves direct transcriptional repression of differentiation program. Mol. Syst. Biol. 9, 654.
Schuster C., Gaillochet C., Medzihradszky A., Busch W., Daum G., Krebs M., Kehle A., Lohmann J.U. (2014) A regulatory framework for shoot stem cell control integrating metabolic, transcriptional, and phytohormone signals. Dev. Cell. 28, 438–449.
Narnoliya L., Basu U., Bajaj D., Malik N., Thakro V., Daware A., Sharma A., Tripathi S., Hegde V.S., Upadhyaya H.D., Singh A.K., Tyagi A.K., Parida S.K. (2019) Transcriptional signatures modulating shoot apical meristem morphometric and plant architectural traits enhance yield and productivity in chickpea. Plant J. Cell Mol. Biol. 98, 864–883.
Kieffer M., Stern Y., Cook H., Clerici E., Maulbetsch C., Laux T., Davies B. (2006) Analysis of the transcription factor WUSCHEL and its functional homologue in Antirrhinum reveals a potential mechanism for their roles in meristem maintenance. Plant Cell. 18, 560–573.
Dolzblasz A., Nardmann J., Clerici E., Causier B., van der Graaff E., Chen J., Davies B., Werr W., Laux T. (2016) Stem cell regulation by Arabidopsis WOX genes. Mol. Plant. 9, 1028–1039.
Lu Z., Shao G., Xiong J., Jiao Y., Wang J., Liu G., Meng X., Liang Y., Xiong G., Wang Y., Li J. (2015) MONOCULM 3, an ortholog of WUSCHEL in rice, is required for tiller bud formation. J. Genet. Genomics. 42, 71–78.
Causier B., Ashworth M., Guo W., Davies B. (2012) The TOPLESS interactome: a framework for gene repression in Arabidopsis. Plant Physiol. 158, 423–438.
Wang L., Kim J., Somers D.E. (2013) Transcriptional corepressor TOPLESS complexes with pseudoresponse regulator proteins and histone deacetylases to regulate circadian transcription. Proc. Natl. Acad. Sci. USA. 110, 761–766.
Zhou Y., Liu X., Engstrom E.M., Nimchuk Z.L., Pruneda-Paz J.L., Tarr P.T., Yan A., Kay S.A., Meyerowitz E.M. (2015) Control of plant stem cell function by conserved interacting transcriptional regulators. Nature. 517, 377–380.
Stuurman J., Jäggi F., Kuhlemeier C. (2002) Shoot meristem maintenance is controlled by a GRAS-gene mediated signal from differentiating cells. Genes Dev. 16, 2213–2218.
Yang Y., Sun M., Yuan C., Han Y., Zheng T., Cheng T., Wang J., Zhang Q. (2019) Interactions between WUSCHEL- and CYC2-like transcription factors in regulating the development of reproductive organs in Chrysanthemum morifolium. Int. J. Mol. Sci. 20, https://doi.org/10.3390/ijms20061276
Sun Z., Su C., Yun J., Jiang Q., Wang L., Wang Y., Cao D., Zhao F., Zhao Q., Zhang M., Zhou B., Zhang L., Kong F., Liu B., Tong Y., Li X. (2019) Genetic improvement of the shoot architecture and yield in soya bean plants via the manipulation of GmmiR156b. Plant Biotechnol. J. 17, 50–62.
Lozano-Sotomayor P., Montes R.A.C., Silvestre-Vañó M., Herrera-Ubaldo H., Greco R., Pablo-Villa J., Galliani B.M., Diaz-Ramirez D., Weemen M., Boutilier K., Pereira A., Colombo L., Madueño F., Marsch-Martínez N., Folter S. (2016) Altered expression of the bZIP transcription factor DRINK ME affects growth and reproductive development in Arabidopsis thaliana. Plant J. 88, 437–451.
Zhang D., Wang X., Wang M., Li J., Guo X., Chong K., Xu Y. (2013) Ectopic expression of WUS in hypocotyl promotes cell division via GRP23 in Arabidopsis. PLoS One. 8, e75773. https://doi.org/10.1371/journal.pone.0075773
Hamada S., Onouchi H., Tanaka H., Kudo M., Liu Y.-G., Shibata D., Machida C., Machida Y. (2000) Mutations in the WUSCHEL gene of Arabidopsis thaliana result in the development of shoots without juvenile leaves. Plant J. 24, 91–101.
Fouracre J.P., Poethig R.S. (2019) Role for the shoot apical meristem in the specification of juvenile leaf identity in Arabidopsis. Proc. Natl. Acad. Sci. USA. 116, 10168–10177.
Li X., Lian H., Zhao Q., He Y. (2019) MicroRNA166 Monitors SPOROCYTELESS/NOZZLE for building of the anther internal boundary. Plant Physiol. 181, 208–220.
Deyhle F., Sarkar A.K., Tucker E.J., Laux T. (2007) WUSCHEL regulates cell differentiation during anther development. Dev. Biol. 302, 154–159.
Gross-Hardt R., Lenhard M., Laux T. (2002) WUSCHEL signaling functions in interregional communication during Arabidopsis ovule development. Genes Dev. 16, 1129–1138.
Sieber P., Gheyselinck J., Gross-Hardt R., Laux T., Grossniklaus U., Schneitz K. (2004) Pattern formation during early ovule development in Arabidopsis thaliana. Dev. Biol. 273, 321–334.
Lieber D., Lora J., Schrempp S., Lenhard M., Laux T. (2011) Arabidopsis WIH1 and WIH2 genes act in the transition from somatic to reproductive cell fate. Curr. Biol. 21, 1009–1017.
Zhao X., Bramsiepe J., Van Durme M., Komaki S., Prusicki M.A., Maruyama D., Forner J., Medzih-radszky A., Wijnker E., Harashima H., Lu Y., Schmidt A., Guthörl D., Logroño R.S., Guan Y., Pochon G., Grossniklaus U., Laux T., Higashiyama T., Lohmann J.U., Nowack M.K., Schnittger A. (2017) RETINOBLASTOMA RELATED1 mediates germline entry in Arabidopsis. Science. 356, eaaf6532. https://doi.org/10.1126/science.aaf653
Liu X., Ning K., Che G., Yan S., Han L., Gu R., Li Z., Weng Y., Zhang X. (2018) CsSPL functions as an adaptor between HD-ZIP III and CsWUS transcription factors regulating anther and ovule development in Cucumis sativus (cucumber). Plant J. 94, 535–547.
Brambilla V., Battaglia R., Colombo M., Masiero S., Bencivenga S., Kater M.M., Colombo L. (2007) Genetic and molecular interactions between BELL1 and MADS box factors support ovule development in Arabidopsis. Plant Cell. 19, 2544–2556.
Yamada T., Sasaki Y., Hashimoto K., Nakajima K., Gasser C.S. (2016) CORONA, PHABULOSA and PHAVOLUTA collaborate with BELL1 to confine WUSCHEL expression to the nucellus in Arabidopsis ovules. Development. 143, 422–426.
Zuo J., Niu Q.-W., Frugis G., Chua N.-H. (2002) The WUSCHEL gene promotes vegetative-to-embryonic transition in Arabidopsis. Plant J. Cell Mol. Biol. 30, 349–359.
Bouchabké-Coussa O., Obellianne M., Linderme D., Montes E., Maia-Grondard A., Vilaine F., Pannetier C. (2013) Wuschel overexpression promotes somatic embryogenesis and induces organogenesis in cotton (Gossypium hirsutum L.) tissues cultured in vitro. Plant Cell Rep. 32, 675–686.
Zheng W., Zhang X., Yang Z., Wu J., Li F., Duan L., Liu C., Lu L., Zhang C., Li F. (2014) AtWuschel promotes formation of the embryogenic callus in Gossypium hirsutum. PLoS One. 9, e87502.
Solís-Ramos L.Y., González-Estrada T., Nahuath-Dzib S., Zapata-Rodriguez L.C., Castaño E. (2008) Overexpression of WUSCHEL in C. chinense causes ectopic morphogenesis. Plant Cell Tissue Organ Cult. 96, 279–287.
Arroyo-Herrera A., Gonzalez A.K., Moo R.C., Quiroz-Figueroa F.R., Loyola-Vargas V.M., Rodriguez-Zapata L.C., D’Hondt C.B., Suárez-Solís V.M., Castaño E. (2008) Expression of WUSCHEL in Coffea canephora causes ectopic morphogenesis and increases somatic embryogenesis. Plant Cell Tissue Organ Cult. 94, 171–180.
Klimaszewska K., Pelletier G., Overton C., Stewart D., Rutledge R.G. (2010) Hormonally regulated overexpression of Arabidopsis WUS and conifer LEC1 (CHAP3A) in transgenic white spruce: implications for somatic embryo development and somatic seedling growth. Plant Cell Rep. 29, 723–734.
Lowe K., Wu E., Wang N., Hoerster G., Hastings C., Cho M.-J., Scelonge C., Lenderts B., Chamberlin M., Cushatt J., Wang L., Ryan L., Khan T., Chow-Yiu J., Hua W., Yu M., Banh J., Bao Z., Brink K., Igo E., Rudrappa B., Shamseer P., Bruce W., Newman L., Shen B., Zheng P., Bidney D., Falco C., Register J., Zhao Z.-Y., Xu D., Jones T., Gordon-Kamm W. (2016) Morphogenic regulators Baby boom and Wuschel improve monocot transformation. Plant Cell. 28, 1998–2015.
Lowe K., La Rota M., Hoerster G., Hastings C., Wang N., Chamberlin M., Wu E., Jones T., Gordon-Kamm W. (2018) Rapid genotype “independent” Zea mays L. (maize) transformation via direct somatic embryogenesis. In Vitro Cell. Dev. Biol. 54, 240–252.
Gambino G., Minuto M., Boccacci P., Perrone I., Vallania R., Gribaudo I. (2011) Characterization of expression dynamics of WOX homeodomain transcription factors during somatic embryogenesis in Vitis vinifera. J. Exp. Bot. 62, 1089–1101.
Rajesh M.K., Fayas T.P., Naganeeswaran S., Rachana K.E., Bhavyashree U., Sajini K.K., Karun A. (2016) De novo assembly and characterization of global transcriptome of coconut palm (Cocos nucifera L.) embryogenic calli using Illumina paired-end sequencing. Protoplasma. 253, 913–928.
Киселев К.В., Шумакова О.А. (2014) Регуляция соматического эмбриогенеза в культурах клеток женьшеня Panax ginseng с помощью гена PgCDPK2DS1. Генетика. 50, 683–691.
Chen S.-K., Kurdyukov S., Kereszt A., Wang X.-D., Gresshoff P.M., Rose R.J. (2009) The association of homeobox gene expression with stem cell formation and morphogenesis in cultured Medicago truncatula. Planta. 230, 827–840.
Orłowska A., Kępczyńska E. (2018) Identification of Polycomb repressive complex1, Trithorax group genes and their simultaneous expression with WUSCHEL, WUSCHEL-related Homeobox5 and SHOOT MERISTEMLESS during the induction phase of somatic embryogenesis in Medicago truncatula Gaertn. Plant Cell Tissue Organ Cult. 134, 345–356.
Salvini M., Fambrini M., Giorgetti L., Pugliesi C. (2016) Molecular aspects of zygotic embryogenesis in sunflower (Helianthus annuus L.): correlation of positive histone marks with HaWUS expression and putative link HaWUS/HaL1L. Planta. 243, 199–215.
Sarkar A.K., Luijten M., Miyashima S., Lenhard M., Hashimoto T., Nakajima K., Scheres B., Heidstra R., Laux T. (2007) Conserved factors regulate signalling in Arabidopsis thaliana shoot and root stem cell organizers. Nature. 446, 811–814.
Forzani C., Aichinger E., Sornay E., Willemsen V., Laux T., Dewitte W., Murray J.A.H. (2014) WOX5 suppresses CYCLIN D activity to establish quiescence at the center of the root stem cell niche. Curr. Biol. 24, 1939–1944.
Tian H., Jia Y., Niu T., Yu Q., Ding Z. (2014) The key players of the primary root growth and development also function in lateral roots in Arabidopsis. Plant Cell Rep. 33, 745–753.
Xu M., Gu X., Liang N., Bian X., Wang H., Qin Y., Pi L., Wu S. (2020) Intersected functional zone of transcriptional regulators patterns stemness within stem cell niche of root apical meristem. J. Integr. Plant Biol. 62, 897–911.
Pi L., Aichinger E., van der Graaff E., Llavata-Peris C.I., Weijers D., Hennig L., Groot E., Laux T. (2015) Organizer-derived WOX5 signal maintains root columella stem cells through chromatin-mediated repression of CDF4 expression. Dev. Cell. 33, 576–588.
Ding Z., Friml J. (2010) Auxin regulates distal stem cell differentiation in Arabidopsis roots. Proc. Natl. Acad. Sci. USA. 107, 12046–12051.
Stahl Y., Wink R.H., Ingram G.C., Simon R. (2009) A signaling module controlling the stem cell niche in Arabidopsis root meristems. Curr. Biol. CB. 19, 909–914.
Berckmans B., Kirschner G., Gerlitz N., Stadler R., Simon R. (2020) CLE40 signaling regulates root stem cell fate. Plant Physiol. 182, 1776–1792.
Shimotohno A., Heidstra R., Blilou I., Scheres B. (2018) Root stem cell niche organizer specification by molecular convergence of PLETHORA and SCARECROW transcription factor modules. Genes Dev. 32, 1085–1100.
Santuari L., Sanchez-Perez G.F., Luijten M., Rutjens B., Terpstra I., Berke L., Gorte M., Prasad K., Bao D., Timmermans-Hereijgers J.L.P.M., Maeo K., Nakamura K., Shimotohno A., Pencik A., Novak O., Ljung K., Heesch S. van, Bruijn E. de, Cuppen E., Willemsen V., Mähönen A.P., Lukowitz W., Snel B., Ridder D. de, Scheres B., Heidstra R. (2016) The PLETHORA gene regulatory network guides growth and cell differentiation in Arabidopsis roots. Plant Cell. 28, 2937–2951.
Long Y., Stahl Y., Weidtkamp-Peters S., Postma M., Zhou W., Goedhart J., Sánchez-Pérez M.-I., Ga-della T.W.J., Simon R., Scheres B., Blilou I. (2017) In vivo FRET–FLIM reveals cell-type-specific protein interactions in Arabidopsis roots. Nature. 548, 97–102.
Tian H., Wabnik K., Niu T., Li H., Yu Q., Pollmann S., Vanneste S., Govaerts W., Rolcík J., Geisler M., Friml J., Ding Z. (2014) WOX5-IAA17 feedback circuit-mediated cellular auxin response is crucial for the patterning of root stem cell niches in Arabidopsis. Mol. Plant. 7, 277–289.
Stahl Y., Grabowski S., Bleckmann A., Kühnemuth R., Weidtkamp-Peters S., Pinto K.G., Kirschner G.K., Schmid J.B., Wink R.H., Hülsewede A., Felekyan S., Seidel C.A.M., Simon R. (2013) Moderation of Arabidopsis root stemness by CLAVATA1 and ARABIDOPSIS CRINKLY4 receptor kinase complexes. Curr. Biol. 23, 362–371.
Meyer M.R., Shah S., Zhang J., Rohrs H., Rao A.G. (2015) Evidence for intermolecular interactions between the intracellular domains of the Arabidopsis receptor-like kinase ACR4, Its homologs and the Wox5 transcription factor. PLoS One. 10, e0118861.
Song S.-K., Hofhuis H., Lee M.M., Clark S.E. (2008) Key divisions in the early Arabidopsis embryo require POL and PLL1 phosphatases to establish the root stem organizer and vascular axis. Dev. Cell. 15, 98–109.
Chu H., Liang W., Li J., Hong F., Wu Y., Wang L., Wang J., Wu P., Liu C., Zhang Q., Xu J., Zhang D. (2013). A CLE-WOX signalling module regulates root meristem maintenance and vascular tissue development in rice. J. Exp. Bot. 64, 5359–5369.
Zhang Y., Jiao Y., Liu Z., Zhu Y.-X. (2015) ROW1 maintains quiescent centre identity by confining WOX5 expression to specific cells. Nat. Commun. 6, 6003.
Olmo R., Cabrera J., Díaz-Manzano F.E., Ruiz-Ferrer V., Barcala M., Ishida T., García A., Andrés M.F., Ruiz-Lara S., Verdugo I., Pernas M., Fukaki H., del Pozo J.C., Moreno-Risueno M.Á., Kyndt T., Gheysen G., Fenoll C., Sawa S., Escobar C. (2020) Root-knot nematodes induce gall formation by recruiting developmental pathways of post-embryonic organogenesis and regeneration to promote transient pluripotency. New Phytol. 227, 200–215.
Lebedeva (Osipova) M.A., Tvorogova V.E., Vinogradova A.P., Gancheva M.S., Azarakhsh M., Ilina E.L., Demchenko K.N., Dodueva I.E., Lutova L.A. (2015) Initiation of spontaneous tumors in radish (Raphanus sativus): cellular, molecular and physiological events. J. Plant Physiol. 173, 97–104.
Osipova M.A., Mortier V., Demchenko K.N., Tsyganov V.E., Tikhonovich I.A., Lutova L.A., Dolgikh E.A., Goormachtig S. (2012) Wuschel-related homeobox5 gene expression and interaction of CLE peptides with components of the systemic control add two pieces to the puzzle of autoregulation of nodulation. Plant Physiol. 158, 1329–1341.
Kundu A., DasGupta M. (2018) Silencing of putative cytokinin receptor histidine kinase1 inhibits both inception and differentiation of root nodules in Arachis hypogaea. Mol. Plant-Microbe Interact. 31, 187–199.
Gao B., Wen C., Fan L., Kou Y., Ma N., Zhao L. (2014) A Rosa canina WUSCHEL-related homeobox gene, RcWOX1, is involved in auxin-induced rhizoid formation. Plant Mol. Biol. 86, 671–679.
Liu S., Wang B., Li X., Pan J., Qian X., Yu Y., Xu P., Zhu J., Xu X. (2019) Lateral organ boundaries domain 19 (LBD19) negative regulate callus formation in Arabidopsis. Plant Cell Tissue Organ Cult. 137, 485–494.
Jiang F., Xu X., Liu H., Zhu J. (2015) DRM1 and DRM2 are involved in Arabidopsis callus formation. Plant Cell Tissue Organ Cult. 123, 221–228.
Kim J., Yang W., Forner J., Lohmann J.U., Noh B., Noh Y. (2018) Epigenetic reprogramming by histone acetyltransferase HAG1/AtGCN5 is required for pluripotency acquisition in Arabidopsis. EMBO J. 37, e98726.
Dorantes-Acosta A.E., Vielle-Calzada J.-P. (2006) The male gametophytic mutant tepitzin1 indicates a requirement of the homeobox gene WOX5 for pollen tube growth in Arabidopsis. Sex. Plant Reprod. 19, 163–173.
Nakata M., Matsumoto N., Tsugeki R., Rikirsch E., Laux T., Okada K. (2012) Roles of the middle domain-specific WUSCHEL-RELATED HOMEOBOX genes in early development of leaves in Arabidopsis. Plant Cell. 24, 519–535.
Zhang F., Tadege M. (2015) Repression of AS2 by WOX family transcription factors is required for leaf development in Medicago and Arabidopsis. Plant Signal. Behav. 10, e993291.
Nakata M., Okada K. (2012) The three-domain model: a new model for the early development of leaves in Arabidopsis thaliana. Plant Signal. Behav. 7, 1423–1427.
Alvarez J.P., Furumizu C., Efroni I., Eshed Y., Bowman J.L. (2016) Active suppression of a leaf meristem orchestrates determinate leaf growth. eLife. 5, e15023.
Guan C., Wu B., Yu T., Wang Q., Krogan N.T., Liu X., Jiao Y. (2017) Spatial auxin signaling controls leaf flattening in Arabidopsis. Curr. Biol. 27, 2940–2950. e4.
Nakata M.T., Tameshige T., Takahara M., Mitsuda N., Okada K. (2018) The functional balance between the WUSCHEL-RELATED HOMEOBOX1 gene and the phytohormone auxin is a key factor for cell proliferation in Arabidopsis seedlings. Plant Biotechnol. 35, 141–154.
Zhang Y., Wu R., Qin G., Chen Z., Gu H., Qu L.-J. (2011) Over-expression of WOX1 leads to defects in meristem development and polyamine homeostasis in Arabidopsis. J. Integr. Plant Biol. 53, 493–506.
Tadege M., Lin H., Bedair M., Berbel A., Wen J., Rojas C.M., Niu L., Tang Y., Sumner L., Ratet P., McHale N.A., Madueño F., Mysore K.S. (2011) STENOFOLIA regulates blade outgrowth and leaf vascular patterning in Medicago truncatula and Nicotiana sylvestris. Plant Cell. 23, 2125–2142.
Zhang F., Wang Y., Li G., Tang Y., Kramer E.M., Tadege M. (2014) STENOFOLIA recruits TOPLESS to repress ASYMMETRIC LEAVES2 at the leaf margin and promote leaf blade outgrowth in Medicago truncatula. Plant Cell. 26, 650–664.
Zhang F., Wang H., Kalve S., Wolabu T.W., Nakashima J., Golz J.F., Tadege M. (2019) Control of leaf blade outgrowth and floral organ development by LEUNIG, ANGUSTIFOLIA3 and WOX transcriptional regulators. New Phytol. 223, 2024–2038.
Wang H., Niu L., Fu C., Meng Y., Sang D., Yin P., Wu J., Tang Y., Lu T., Wang Z.-Y., Tadege M., Lin H. (2017) Overexpression of the WOX gene STENOFOLIA improves biomass yield and sugar release in transgenic grasses and display altered cytokinin homeostasis. PLoS Genet. 13, e1006649.
Liu M., Lei L., Miao F., Powers C., Zhang X., Deng J., Tadege M., Carver B.F., Yan L. (2018) The STENOFOLIA gene from Medicago alters leaf width, flowering time and chlorophyll content in transgenic wheat. Plant Biotechnol. J. 16, 186–196.
Zhuang L.-L., Ambrose M., Rameau C., Weng L., Yang J., Hu X.-H., Luo D., Li X. (2012) LATHYROIDES, encoding a WUSCHEL-related Homeobox1 transcription factor, controls organ lateral growth, and regulates tendril and dorsal petal identities in garden pea (Pisum sativum L.). Mol. Plant. 5, 1333–1345.
Vandenbussche M., Horstman A., Zethof J., Koes R., Rijpkema A.S., Gerats T. (2009) Differential recruitment of WOX transcription factors for lateral development and organ fusion in Petunia and Arabidopsis. Plant Cell. 21, 2269–2283.
Matsumoto N., Okada K. (2001) A homeobox gene, PRESSED FLOWER, regulates lateral axis-dependent development of Arabidopsis flowers. Genes Dev. 15, 3355–3364.
Nardmann J., Ji J., Werr W., Scanlon M.J. (2004) The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131, 2827–2839.
Niu L., Lin H., Zhang F., Watira T.W., Li G., Tang Y., Wen J., Ratet P., Mysore K.S., Tadege M. (2015) LOOSE FLOWER, a WUSCHEL-like homeobox gene, is required for lateral fusion of floral organs in Medicago truncatula. Plant J. Cell Mol. Biol. 81, 480–492.
Tadege M., Lin H., Bedair M., Berbel A., Wen J., Rojas C.M., Niu L., Tang Y., Sumner L., Ratet P., McHale N.A., Madueño F., Mysore K.S. (2011) STENOFOLIA regulates blade outgrowth and leaf vascular patterning in Medicago truncatula and Nicotiana sylvestris. Plant Cell. 23, 2125–2142.
Shimizu R., Ji J., Kelsey E., Ohtsu K., Schnable P.S., Scanlon M.J. (2009) Tissue specificity and evolution of meristematic WOX3 function. Plant Physiol. 149, 841–850.
Ishiwata A., Ozawa M., Nagasaki H., Kato M., Noda Y., Yamaguchi T., Nosaka M., Shimizu-Sato S., Nagasaki A., Maekawa M., Hirano H.-Y., Sato Y. (2013) Two WUSCHEL-related homeobox genes, narrow leaf2 and narrow leaf3, control leaf width in rice. Plant Cell Physiol. 54, 779–792.
Cho S.-H., Yoo S.-C., Zhang H., Pandeya D., Koh H.-J., Hwang J.-Y., Kim G.-T., Paek N.-C. (2013) The rice narrow leaf2 and narrow leaf3 loci encode WUSCHEL-related homeobox 3A (OsWOX3A) and function in leaf, spikelet, tiller and lateral root development. New Phytol. 198, 1071–1084.
Dai M., Hu Y., Zhao Y., Liu H., Zhou D.-X. (2007) A WUSCHEL-LIKE HOMEOBOX gene represses a YABBY gene expression required for rice leaf development. Plant Physiol. 144, 380–390.
Yoo S.-C., Cho S.-H., Paek N.-C. (2013) Rice WUSCHEL-related homeobox 3A (OsWOX3A) modulates auxin-transport gene expression in lateral root and root hair development. Plant Signal. Behav. 8, https://doi.org/10.4161/psb.25929
Cho S.-H., Kang K., Lee S.-H., Lee I.-J., Paek N.-C. (2016) OsWOX3A is involved in negative feedback regulation of the gibberellic acid biosynthetic pathway in rice (Oryza sativa). J. Exp. Bot. 67, 1677–1687.
Nardmann J., Zimmermann R., Durantini D., Kranz E., Werr W. (2007) WOX gene phylogeny in Poaceae: a comparative approach addressing leaf and embryo development. Mol. Biol. Evol. 24, 2474–2484.
Li J., Yuan Y., Lu Z., Yang L., Gao R., Lu J., Li J., Xiong G. (2012) Glabrous rice 1, encoding a homeodomain protein, regulates trichome development in rice. Rice. 5, 32.
Angeles-Shim R.B., Asano K., Takashi T., Shim J., Kuroha T., Ayano M., Ashikari M. (2012) A WUSCHEL-related homeobox 3B gene, depilous (dep), confers glabrousness of rice leaves and glumes. Rice. 5, 28.
Zhang H., Wu K., Wang Y., Peng Y., Hu F., Wen L., Han B., Qian Q., Teng S. (2012) A WUSCHEL-like homeobox gene, OsWOX3B responses to NUDA/GL-1 locus in rice. Rice. 5, 30.
Sun W., Gao D., Xiong Y., Tang X., Xiao X., Wang C., Yu S. (2017) Hairy Leaf 6, an AP2/ERF transcription factor, interacts with OsWOX3B and regulates trichome formation in rice. Mol. Plant. 10, 1417–1433.
Honda E., Yew C.-L., Yoshikawa T., Sato Y., Hibara K.-I., Itoh J.-I. (2018) LEAF LATERAL SYMMETRY1, a member of the WUSCHEL-RELATED HOMEOBOX3 gene family, regulates lateral organ development differentially from other paralogs, NARROW LEAF2 and NARROW LEAF3 in rice. Plant Cell Physiol. 59, 376–391.
Alvarez J.M., Sohlberg J., Engström P., Zhu T., Englund M., Moschou P.N., von Arnold S. (2015) The WUSCHEL-RELATED HOMEOBOX 3 gene PaWOX3 regulates lateral organ formation in Norway spruce. New Phytol. 208, 1078–1088.
Zhou X., Guo Y., Zhao P., Sun M.-X. (2018) Comparative analysis of WUSCHEL-RELATED HOMEOBOX genes revealed their parent-of-origin and cell type-specific expression pattern during early embryogenesis in tobacco. Front. Plant Sci. 9, 311.
Wang L., Yuan J., Ma Y., Jiao W., Ye W., Yang D.-L., Yi C., Chen Z.J. (2018) Rice interploidy crosses disrupt epigenetic regulation, gene expression, and seed development. Mol. Plant. 11, 300–314.
Zhu T., Moschou P.N., Alvarez J.M., Sohlberg J.J., von Arnold S. (2016) WUSCHEL-RELATED HOMEOBOX 2 is important for protoderm and suspensor development in the gymnosperm Norway spruce. BMC Plant Biol. 16, 19.
Breuninger H., Rikirsch E., Hermann M., Ueda M., Laux T. (2008) Differential expression of WOX genes mediates apical-basal axis formation in the Arabidopsis embryo. Dev. Cell. 14, 867–876.
Lie C., Kelsom C., Wu X. (2012) WOX2 and STIMPY-LIKE/WOX8 promote cotyledon boundary formation in Arabidopsis. Plant J. Cell Mol. Biol. 72, 674–682.
Wu X., Chory J., Weigel D. (2007) Combinations of WOX activities regulate tissue proliferation during Arabidopsis embryonic development. Dev. Biol. 309, 306–316.
Zhang Z., Tucker E., Hermann M., Laux T. (2017) A molecular framework for the embryonic initiation of shoot meristem stem cells. Dev. Cell. 40, 264–277. e4.
Alvarez J.M., Bueno N., Cañas R.A., Avila C., Cánovas F.M., Ordás R.J. (2018) Analysis of the WUSCHEL-RELATED HOMEOBOX gene family in Pinus pinaster: new insights into the gene family evolution. Plant Physiol. Biochem. 123, 304–318.
Kyo M., Maida K., Nishioka Y., Matsui K. (2018) Coexpression of WUSCHEL related homeobox (WOX) 2 with WOX8 or WOX9 promotes regeneration from leaf segments and free cells in Nicotiana tabacum L. Plant Biotechnol. 35, 23–30.
Suer S., Agusti J., Sanchez P., Schwarz M., Greb T. (2011) WOX4 imparts auxin responsiveness to cambium cells in Arabidopsis. Plant Cell. 23, 3247–3259.
Nardmann J., Werr W. (2013) Symplesiomorphies in the WUSCHEL clade suggest that the last common ancestor of seed plants contained at least four independent stem cell niches. New Phytol. 199, 1081–1092.
Hirakawa Y., Kondo Y., Fukuda H. (2010) TDIF peptide signaling regulates vascular stem cell proliferation via the WOX4 homeobox gene in Arabidopsis. Plant Cell. 22, 2618–2629.
Kucukoglu M., Nilsson J., Zheng B., Chaabouni S., Nilsson O. (2017) WUSCHEL-RELATED HOMEOBOX4 (WOX4)-like genes regulate cambial cell division activity and secondary growth in Populus trees. New Phytol. 215, 642–657.
Guo X., Wang J., Gardner M., Fukuda H., Kondo Y., Etchells J.P., Wang X., Mitchum M.G. (2017) Identification of cyst nematode B-type CLE peptides and modulation of the vascular stem cell pathway for feeding cell formation. PLoS Pathog. 13, e1006142.
Brackmann K., Qi J., Gebert M., Jouannet V., Schlamp T., Grünwald K., Wallner E.-S., Novikova D.D., Levitsky V.G., Agustí J., Sanchez P., Lohmann J.U., Greb T. (2018) Spatial specificity of auxin responses coordinates wood formation. Nat. Commun. 9, 875.
Smetana O., Mäkilä R., Lyu M., Amiryousefi A., Sánchez Rodríguez F., Wu M.-F., Solé-Gil A., Leal Gavarrón M., Siligato R., Miyashima S., Roszak P., Blomster T., Reed J.W., Broholm S., Mähönen A.P. (2019) High levels of auxin signalling define the stem-cell organizer of the vascular cambium. Nature. 565, 485–489.
Etchells J.P., Provost C.M., Mishra L., Turner S.R. (2013) WOX4 and WOX14 act downstream of the PXY receptor kinase to regulate plant vascular proliferation independently of any role in vascular organisation. Development. 140, 2224–2234.
Denis E., Kbiri N., Mary V., Claisse G., Silva N.C., Kreis M., Deveaux Y. (2017) WOX14 promotes bioactive gibberellin synthesis and vascular cell differentiation in Arabidopsis. Plant J. 90, 560–572.
Yasui Y., Ohmori Y., Takebayashi Y., Sakakibara H., Hirano H.-Y. (2018) WUSCHEL-RELATED HOMEOBOX4 acts as a key regulator in early leaf development in rice. PLoS Genet. 14, e1007365.
Ohmori Y., Tanaka W., Kojima M., Sakakibara H., Hirano H.-Y. (2013) WUSCHEL-RELATED HOMEOBOX4 is involved in meristem maintenance and is negatively regulated by the CLE gene FCP1 in rice. Plant Cell. 25, 229–241.
Nic-Can G.I., López-Torres A., Barredo-Pool F., Wrobel K., Loyola-Vargas V.M., Rojas-Herrera R., De-la-Peña C. (2013) New insights into somatic embryogenesis: LEAFY COTYLEDON1, BABY BOOM1 and WUSCHEL-RELATED HOMEOBOX4 are epigenetically regulated in Coffea canephora. PLoS One. 8, e72160.
Imin N., Nizamidin M., Wu T., Rolfe B.G. (2007) Factors involved in root formation in Medicago truncatula. J. Exp. Bot. 58, 439–451.
Park S.O., Hwang S., Hauser B.A. (2004) The phenotype of Arabidopsis ovule mutants mimics the morphology of primitive seed plants. Proc. R. Soc. B. Biol. Sci. 271, 311–316.
Park S.O., Zheng Z., Oppenheimer D.G., Hauser B.A. (2005) The PRETTY FEW SEEDS2 gene encodes an Arabidopsis homeodomain protein that regulates ovule. Development. 132, 841–849.
Pillitteri L.J., Bemis S.M., Shpak E.D., Torii K.U. (2007) Haploinsufficiency after successive loss of signaling reveals a role for ERECTA-family genes in Arabidopsis ovule development. Development. 134, 3099–3109.
Zhu J., Shi H., Lee B., Damsz B., Cheng S., Stirm V., Zhu J.-K., Hasegawa P.M., Bressan R.A. (2004) An Arabidopsis homeodomain transcription factor gene, HOS9, mediates cold tolerance through a CBF-independent pathway. Proc. Natl. Acad. Sci. USA. 101, 9873–9878.
Kong D., Hao Y., Cui H. (2016) The WUSCHEL related homeobox protein WOX7 regulates the sugar response of lateral root development in Arabidopsis thaliana. Mol. Plant. 9, 261–270.
Fiume E., Fletcher J.C. (2012) Regulation of Arabidopsis embryo and endosperm development by the polypeptide signaling molecule CLE8. Plant Cell. 24, 1000–1012.
Ueda M., Zhang Z., Laux T. (2011) Transcriptional activation of Arabidopsis axis patterning genes WOX8/9 links zygote polarity to embryo development. Dev. Cell. 20, 264–270.
Ueda M., Aichinger E., Gong W., Groot E., Verstraeten I., Vu L.D., De Smet I., Higashiyama T., Umeda M., Laux T. (2017) Transcriptional integration of paternal and maternal factors in the Arabidopsis zygote. Genes Dev. 31, 617–627.
Wu X., Dabi T., Weigel D. (2005) Requirement of homeobox gene STIMPY/WOX9 for Arabidopsis meristem growth and maintenance. Curr. Biol. 15, 436–440.
Fang F., Ye S., Tang J., Bennett M.J., Liang W. (2020) DWT1/DWL2 acts together with OsPIP5K1 to regulate plant uniform growth in rice. New Phytol. 225, 1234–1246.
Творогова В.Е., Лебедева М.А., Лутова Л.А. (2015) Экспрессия генов WOX и PIN в соматическом и зиготическом эмбриогенезе у Medicago truncatula. Генетика. 51, 1376–1385.
Tvorogova V.E., Fedorova Y.A., Potsenkovskaya E.A., Kudriashov A.A., Efremova E.P., Kvitkovskaya V.A., Wolabu T.W., Zhang F., Tadege M., Lutova L.A. (2019) The WUSCHEL-related homeobox transcription factor MtWOX9-1 stimulates somatic embryogenesis in Medicago truncatula. Plant Cell Tissue Organ Cult. 138, 517–527.
Zhu T., Moschou P.N., Alvarez J.M., Sohlberg J.J., von Arnold S. (2014) WUSCHEL-RELATED HOMEOBOX 8/9 is important for proper embryo patterning in the gymnosperm Norway spruce. J. Exp. Bot. 65, 6543–6552.
Peng L., Skylar A., Chang P.L., Bisova K., Wu X. (2014) CYCP2;1 integrates genetic and nutritional information to promote meristem cell division in Arabidopsis. Dev. Biol. 393, 160–170.
Rebocho A.B., Bliek M., Kusters E., Castel R., Procissi A., Roobeek I., Souer E., Koes R. (2008) Role of EVERGREEN in the development of the Cymose petunia inflorescence. Dev. Cell. 15, 437–447.
Schorderet M., Duvvuru Muni R.R., Fiebig A., Reinhardt D. (2018) Deregulation of MADS-box transcription factor genes in a mutant defective in the WUSCHEL-LIKE HOMEOBOX gene EVERGREEN of Petunia hybrida. Plant Signal. Behav. 13, e1471299.
Lippman Z.B., Cohen O., Alvarez J.P., Abu-Abied M., Pekker I., Paran I., Eshed Y., Zamir D. (2008) The making of a compound inflorescence in tomato and related nightshades. PLoS Biol. 6, e288.
Park S.J., Jiang K., Schatz M.C., Lippman Z.B. (2012) Rate of meristem maturation determines inflorescence architecture in tomato. Proc. Natl. Acad. Sci. USA. 109, 639–644.
Cohen O., Borovsky Y., David-Schwartz R., Paran I. (2014) Capsicum annuum S (CaS) promotes reproductive transition and is required for flower formation in pepper (Capsicum annuum). New Phytol. 202, 1014–1023.
Wang W., Li G., Zhao J., Chu H., Lin W., Zhang D., Wang Z., Liang W. (2014) Dwarf Tiller1, a Wuschel-related homeobox transcription factor, is required for tiller growth in rice. PLoS Genet. 10, e1004154.
Liu J., Sheng L., Xu Y., Li J., Yang Z., Huang H., Xu L. (2014) WOX11 and 12 are involved in the first-step cell fate transition during de novo root organogenesis in Arabidopsis. Plant Cell. 26, 1081–1093.
Hu X., Xu L. (2016) Transcription factors WOX11/12 directly activate WOX5/7 to promote root primordia initiation and organogenesis. Plant Physiol. 172, 2363–2373.
Sheng L., Hu X., Du Y., Zhang G., Huang H., Scheres B., Xu L. (2017) Non-canonical WOX11-mediated root branching contributes to plasticity in Arabidopsis root system architecture. Development. 144, 3126–3133.
Chen L., Tong J., Xiao L., Ruan Y., Liu J., Zeng M., Huang H., Wang J.-W., Xu L. (2016) YUCCA-mediated auxin biogenesis is required for cell fate transition occurring during de novo root organogenesis in Arabidopsis. J. Exp. Bot. 67, 4273–4284.
Li H., Yao L., Sun L., Zhu Z. (2020) ETHYLENE INSENSITIVE 3 suppresses plant de novo root regeneration from leaf explants and mediates age-regulated regeneration decline. Development. 147, dev179457.
Yan A., Borg M., Berger F., Chen Z. (2020) The atypical histone variant H3.15 promotes callus formation in Arabidopsis thaliana. Development. 147, dev184895.
Hu B., Zhang G., Liu W., Shi J., Wang H., Qi M., Li J., Qin P., Ruan Y., Huang H., Zhang Y., Xu L. (2017) Divergent regeneration-competent cells adopt a common mechanism for callus initiation in angiosperms. Regen. Oxf. Engl. 4, 132–139.
Liu B., Wang L., Zhang J., Li J., Zheng H., Chen J., Lu M. (2014) WUSCHEL-related homeobox genes in Populus tomentosa: diversified expression patterns and a functional similarity in adventitious root formation. BMC Genomics. 15, 296.
Xu M., Xie W., Huang M. (2015) Two WUSCHEL-related HOMEOBOX genes, PeWOX11a and PeWOX11b, are involved in adventitious root formation of poplar. Physiol. Plant. 155, 446–456.
Liu B., Zhang J., Yang Z., Matsui A., Seki M., Li S., Yan X., Kohnen M.V., Gu L., Prasad K., Tuskan G.A., Lu M., Oka Y. (2018) PtWOX11 acts as master regulator conducting the expression of key transcription factors to induce de novo shoot organogenesis in poplar. Plant Mol. Biol. 98, 389–406.
Yu J., Zhang Y., Liu W., Wang H., Wen S., Zhang Y., Xu L. (2020) Molecular evolution of auxin-mediated root initiation in plants. Mol. Biol. Evol. 37, 1387–1393.
Zhang T., Li R., Xing J., Yan L., Wang R., Zhao Y. (2018) The YUCCA-auxin-WOX11 module controls crown root development in rice. Front. Plant Sci. 9, 523.
Zhao Y., Hu Y., Dai M., Huang L., Zhou D.-X. (2009) The WUSCHEL-related homeobox gene WOX11 is required to activate shoot-borne crown root development in rice. Plant Cell. 21, 736–748.
Zhao Y., Cheng S., Song Y., Huang Y., Zhou S., Liu X., Zhou D.-X. (2015) The interaction between rice ERF3 and WOX11 promotes crown root development by regulating gene expression involved in cytokinin signaling. Plant Cell. 27, 2469–2483.
Zhou S., Jiang W., Long F., Cheng S., Yang W., Zhao Y., Zhou D.-X. (2017) Rice homeodomain protein WOX11 recruits a histone acetyltransferase complex to establish programs of cell proliferation of crown root meristem. Plant Cell. 29, 1088–1104.
Jiang W., Zhou S., Zhang Q., Song H., Zhou D.-X., Zhao Y. (2017) Transcriptional regulatory network of WOX11 is involved in the control of crown root development, cytokinin signals, and redox in rice. J. Exp. Bot. 68, 2787–2798.
Cheng S., Zhou D.-X., Zhao Y. (2016) WUSCHEL-related homeobox gene WOX11 increases rice drought resistance by controlling root hair formation and root system development. Plant Signal. Behav. 11, e1130198.
Chen G., Feng H., Hu Q., Qu H., Chen A., Yu L., Xu G. (2015) Improving rice tolerance to potassium deficiency by enhancing OsHAK16p:WOX11-controlled root development. Plant Biotechnol. J. 13, 833–848.
Cheng S., Tan F., Lu Y., Liu X., Li T., Yuan W., Zhao Y., Zhou D.-X. (2018) WOX11 recruits a histone H3K27me3 demethylase to promote gene expression during shoot development in rice. Nucl. Acids Res. 46, 2356–2369.
Zhang N., Yu H., Yu H., Cai Y., Huang L., Xu C., Xiong G., Meng X., Wang J., Chen H., Liu G., Jing Y., Yuan Y., Liang Y., Li S., Smith S.M., Li J., Wang Y. (2018) A core regulatory pathway controlling rice tiller angle mediated by the LAZY1-dependent asymmetric distribution of auxin. Plant Cell. 30, 1461–1475.
Deveaux Y., Toffano-Nioche C., Claisse G., Thareau V., Morin H., Laufs P., Moreau H., Kreis M., Lecharny A. (2008) Genes of the most conserved WOX clade in plants affect root and flower development in Arabidopsis. BMC Evol. Biol. 8, 291.
Liu J., Jiang C., Chen T., Zha L., Zhang J., Huang L. (2019) Identification and 3D gene expression patterns of WUSCEHEL-related homeobox (WOX) genes from Panax ginseng. Plant Physiol. Biochem. 143, 257–264.
Schlögl P.S., dos Santos A.L.W., do Nascimento Vieira L., Floh E.I.S., Guerra M.P. (2012) Gene expression during early somatic embryogenesis in Brazilian pine (Araucaria angustifolia (Bert.) O. Ktze). Plant Cell Tissue Organ Cult. 108, 173–180.
He P., Zhang Y., Liu H., Yuan Y., Wang C., Yu J., Xiao G. (2019) Comprehensive analysis of WOX genes uncovers that WOX13 is involved in phytohormone-mediated fiber development in cotton. BMC Plant Biol. 19, 312.
Minh-Thu P.-T., Kim J.S., Chae S., Jun K.M., Lee G.-S., Kim D.-E., Cheong J.-J., Song S.I., Nahm B.H., Kim Y.-K. (2018) A WUSCHEL homeobox transcription factor, OsWOX13, enhances drought tolerance and triggers early flowering in rice. Mol. Cells. 41, 781–798.
Romera-Branchat M., Ripoll J.J., Yanofsky M.F., Pelaz S. (2013) The WOX13 homeobox gene promotes replum formation in the Arabidopsis thaliana fruit. Plant J. 73, 37–49.
Sakakibara K., Reisewitz P., Aoyama T., Friedrich T., Ando S., Sato Y., Tamada Y., Nishiyama T., Hiwatashi Y., Kurata T., Ishikawa M., Deguchi H., Rensing S.A., Werr W., Murata T., Hasebe M., Laux T. (2014) WOX13-like genes are required for reprogramming of leaf and protoplast cells into stem cells in the moss Physcomitrella patens. Development. 141, 1660–1670.
Petzold H.E., Chanda B., Zhao C., Rigoulot S.B., Beers E.P., Brunner A.M. (2018) DIVARICATA AND RADIALIS INTERACTING FACTOR (DRIF) also interacts with WOX and KNOX proteins associated with wood formation in Populus trichocarpa. Plant J. 93, 1076–1087.
Smit M.E., McGregor S.R., Sun H., Gough C., Bågman A.-M., Soyars C.L., Kroon J.T., Gaudinier A., Williams C.J., Yang X., Nimchuk Z.L., Weijers D., Turner S.R., Brady S.M., Etchells J.P. (2020) A PXY-mediated transcriptional network integrates signaling mechanisms to control vascular development in Arabidopsis. Plant Cell. 32, 319–335.
Lin H., Niu L., McHale N.A., Ohme-Takagi M., Mysore K.S., Tadege M. (2013) Evolutionarily conserved repressive activity of WOX proteins mediates leaf blade outgrowth and floral organ development in plants. Proc. Natl. Acad. Sci. USA. 110, 366–371.
Дополнительные материалы
- скачать ESM_1.xlsx
- Дополнительная таблица 1. Описанные в статье регуляторы, мишени и партнеры белков WOX A. thaliana и/или их ортологов, аспекты развития, в которые они вовлечены, и методы, которыми они были обнаружены.
- скачать ESM_2.docx
- Дополнительная таблица 2. Описанные в статье регуляторы, мишени и партнеры белков WOX A. thaliana и/или их ортологов
Инструменты
Молекулярная биология