Прикладная биохимия и микробиология, 2019, T. 55, № 4, стр. 366-377
Молекулярно-генетический и функциональный анализ генома бактерий Bacillus velezensis БИМ В-439Д
А. В. Бережная 1, *, О. В. Евдокимова 1, Л. Н. Валентович 1, Н. В. Сверчкова 1, М. А. Титок 1, Э. И. Коломиец 1
1 Институт микробиологии НАН Беларуси
220141 Минск, Беларусь
* E-mail: anastasiya1981@bk.ru
Поступила в редакцию 09.10.2018
После доработки 10.01.2019
Принята к публикации 20.02.2019
Аннотация
В результате полногеномного секвенирования бактерий Bacillus velezensis БИМ В-439Д было установлено, что их геном представлен одной кольцевой хромосомой размером 3 978 954 п.н. и содержит 3969 предсказанных генов. В хромосоме выявлено 10 консервативных локусов, определяющих продукцию антимикробных соединений разной химической природы: липопептиды (сурфактин, бацилломицин, фенгицин), поликетиды (бациллен, диффицидин/оксидиффицидин, макролактин), сидерофор бациллибактин, дипептид бацилизин, белок/поликетид неопределенного состава и бактериоцин амилоциклицин. Методом направленного мутагенеза получены мутанты с нарушенным синтезом сурфактина, отличающиеся повышенным образованием бацилломицина и фенгицина. При этом мутанты демонстрировали высокую антимикробную активность в отношении ряда бактериальных и грибных патогенов (Penicillium expansum, Alternaria tenuis, Botrytis cinerea, Bipolaris sorokiniana), но в меньшей степени подавляли рост грибов рода Fusarium.
Отличительной особенностью филогенетически близких бактерий Bacillus amyloliquefaciens subsp. plantarum и Bacillus velezensis является способность вступать в плотный контакт с корневой системой растений и продуцировать широкий спектр биологически активных соединений, обеспечивающих подавление патогенов и стимулирующих рост и развитие растения [1]. В геноме микроорганизмов данной таксономической группы присутствует большое число хромосомных локусов, определяющих синтез антимикробных соединений разной химической природы, образование которых в клетке происходит без участия рибосом. Это липопептиды (сурфактин, фенгицин и бацилломицин), поликетиды (макролактин, бациллен, диффицидин/оксидиффицидин), дипептид бацилизин и сидерофор бациллибактин. Помимо этого, отдельные представители B. amyloliquefaciens subsp. plantarum и Bacillus velezensis способны синтезировать полипептид/поликетид с неизвестным химическим составом, а также бактериоцины [2]. Благодаря способности подавлять широкий круг патогенов (бактерии, грибы, вирусы) данные бактерии применяются в качестве основы безопасных для окружающей среды биопрепаратов при получении сельскохозяйственной продукции. В хозяйствах Pеспублики Беларусь широко используется созданный на основе бактерий B. velezensis БИМ В-439Д зарегистрированный биопестицид Бетапротектин, эффективно снижающий поражение сахарной и столовой свеклы кагатной гнилью, огурцов и томатов прикорневой гнилью, сосны обыкновенной диплодиозом (http://mbio.bas-net.by/prod/betaprotectin/).
В настоящее время для детального изучения практически важных микроорганизмов широко используется метод полногеномного секвенирования. Данная технология позволяет в полной мере оценить генетический потенциал организма, научно обосновать его биологическую безопасность и создать исчерпывающий генетический паспорт для коммерческого использования, а также заложить основу для изучения тонких молекулярно-генетических механизмов, определяющих практически важные свойства.
Имея полногеномную последовательность, можно с помощью сравнения с уже аннотированными геномами хорошо изученных штаммов отобрать ряд генетических детерминант, предположительно участвующих в исследуемом процессе и оценить их роль. Одним из методов функциональной геномики является направленный мутагенез и анализ фенотипа получившихся мутантов. Инсерционные мутанты, у которых нарушен синтез определенных метаболитов, позволяют продемонстрировать роль этих соединений в формировании антагонистической активности штамма.
Цель работы – молекулярно-генетический анализ генома бактерий B. velezensis БИМ В-439Д, обладающих выраженным антагонизмом к широкому спектру патогенных грибов и бактерий и стимулирующих рост культурных растений, а также оценка роли сурфактина в проявлении антагонистических свойств штамма.
МЕТОДИКА
Штаммы микроорганизмов. В работе использовали штамм спорообразующих бактерий B. velezensis БИМ В-439Д (ранее определен как B. amyloliquefaciens subsp. plantarum), выделенный из образца дерново-подзолистой почвы на территории Минской области и использующийся в качестве основы зарегистрированного биопрепарата Бетапротектин; коммерческие штаммы Escherichia coli XL1-Blue [3], E. coli GM2163 [4], и коллекционные плазмиды pUC19 [5], pMTL21E [6], а также штаммы фитопатогенных бактерий Pseudomonas syringae pv. syringae БИМ В-695, Xanthomonas campestris pv. campestris БИМ В-634, Agrobacterium tumefaciens 8628 и фитопатогенных грибов Penicillium expansum БИМ F-564, Alternaria tenuis БИМ F-460, Fusarium graminearum БИМ F-601, Fusarium oxysporum БИМ F-381, Fusarium culmorum БИМ F-459 и Fusarium avenaсeum БИМ F-458, Botrytis cinerea БИМ F-383, Bipolaris sorokiniana БИМ F-464, Phoma betae БИМ F-461 из Белорусской коллекции непатогенных микроорганизмов Института микробиологии НАН Беларуси.
Среды и растворы. Для выращивания бактерий использовали среды LB и Мейнелла [7], фитопатогенных грибов – картофельно-глюкозную среду [8]. Агаризованные среды содержали 1.2 или 2% агар-агара. Коммерческие препараты антибиотиков ампициллина и/или эритромицина применяли в концентрации 100 мкг/мл.
Методы исследований. Тотальную ДНК выделяли с помощью набора реактивов Bacteria DNA Preparation Kit “Jena Bioscience GmbH” (Германия). Плазмидную ДНК выделяли с использованием набора реактивов GeneJET Plasmid Miniprep “Thermo Fisher Scientific” (США).
Для полимеразной цепной реакции (ПЦР) использовали реактивы фирмы “Thermo Fisher Scientific” (США). Реакционная смесь (50 мкл), содержала 100 нг ДНК матрицы, дНТФ (0.2 ммоль/л каждого нуклеотида), 1.5 ммоль/л MgCl2, 0.2 мкмоль/л каждого праймера, 0.5 ед. Pfu-полимеразы и соответствующий буфер (1х). Амплификацию фрагмента гена srfAA проводили в режиме: 95°С – 5 мин (1 цикл); 95°С – 30 с, 57°С – 30 с, 72°С – 2.5 мин (30 циклов); 72°С – 10 мин (1 цикл). Для амплификации фрагментов генов baeB, dfnA, mlnA, fenA, bmyD, dhbA проводили ПЦР по аналогичному протоколу, за исключением температуры отжига праймеров (54–56°С). Список используемых праймеров производства “Праймтех” (Беларусь) приведен в табл. 1. Продукты ПЦР очищали из геля с использованием набора реактивов MinElute PCR Purification Kit “Qiagen” (Германия).
Таблица 1.
Праймеры, использованные в работе
| Ген, продукт, синтез которого детерминируется локусом, содержащим данный ген | Нуклеотидная последовательность праймеров | Длина ПЦР-продукта, п. н. |
|---|---|---|
| baeB, бациллен | Fbae TGGTGTCATCTGGTAAAGTGG Rbae ACTCAGCCATTCATCACCAG |
860 |
| dhbA, бациллибактин | Fbaci TGACTGCGTGTTTTGATTTGG Rbaci AACGCATAAATCATGCATCG |
846 |
| bmyD, бацилломицин | Fbac GCAGGAAGGTGTAACAATGG Rbac TCCTTGTTGCGGAAATGTCC |
723 |
| dfnA, диффицидин/оксидиффицидин | Fdif AGATGCTTACGTTTGTGTTCC RdifAGCAGTATCAGACTCTTCTTTCG |
887 |
| fenA, фенгицин | Ffen AACTCACGCACAAAGAAGAG Rfen ACAAAGGACAGGAACTCTGC |
903 |
| mlnA, макролактин | FmacACATAAAACCAATCGTTGTTTCG Rmac TGTCCCATTAGATACCTGATGC |
1077 |
| srfAA, сурфактин | FsrfTTGAGAAAGCGATACAGGAATTCG RsrfTCGCATCTTCAATCAGCGTGATC |
1131 |
Рестриктазы SmaI, EcoRI и ДНК-лигазу фага Т4 производства “Thermo Fisher Scientific” (США) использовали в соответствии с рекомендациями фирмы изготовителя.
Трансформацию бактерий E. coli и B. velezensis БИМ В-439Д осуществляли согласно методикам, изложенным в публикациях [9, 10].
Для постановки секвенирующей реакции по Сэнгеру (определение последовательностей отдельных генетических детерминант) использовали набор DNA Cycle Sequensing Kit “Jena Bioscience GmbH” (Германия) и стандартные праймеры M13 Forward, M13 Reverse “Праймтех” (Беларусь). Продукты секвенирующей реакции детектировали с помощью автоматического секвенатора 4300 DNA Analyzer, “Li-COR” (США). Полученные результаты анализировали с помощью программ eSeq Version 3.1 “Li-COR” (США), BLASTN2.2.1 (http://www.ncbi.nlm.nih.gov), Primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/ primer-blast/) и базы данных ГенБанк (https:// www.ncbi.nlm.nih.gov/ genbank/).
Подготовку библиотеки ДНК для полногеномного секвенирования осуществляли с помощью набора Nextera XT “Illumina” (США). Определение нуклеотидных последовательностей проводили на приборе MiSeq “Illimina” (США), используя комплект реактивов MiSeq Reagent Kit v3 (600-cycle) (США). Адаптерные и недостоверные последовательности удаляли с помощью программы Trimmomatic-0.38 (https://academic.oup.com/bioinformatics/ article/30/15/2114/2390096).
Отобранные парноконцевые последовательности размером от 50 и до 300 п.н. собирали в контиги с помощью программы SPAdes-3.11.1 (http:// spades.bioinf.spbau.ru/release3.11.1/manual.html). Полученные контиги объединяли в хромосомную последовательность используя для сравнения доступные геномы наиболее близкородственных штаммов – Bs-916 (CP009611) и TrigoCor1448 (CP007244). Предварительную аннотацию генома проводили с помощью системы RAST 2.0 и PROKKA 1.12. Поиск профаговых последовательностей осуществлялся с помощью веб-сервиса PHASTER (http://phaster.ca/). Подсчет средней нуклеотидной идентичности (ANI – Average Nucleotide Identity) осуществлялся с помощью веб-сервиса JSpeciesWS (http://jspecies.ribohost.com/ jspeciesws/) [11].
Направленный мутагенез бактерий B. velezensis БИМ В-439Д проводили с использованием суицидальной плазмиды pMTL21E, в EcoRI-сайт которой был встроен EcoRI-фрагмент гена srfAA размером 774 п.н., вырезанный из плазмиды pUC19:: srfAA (ПЦР-продукт гена srfAA размером 1131 п.н., встроенный в SmaI-сайт плазмиды pUC19, содержал два сайта для рестриктазы EcoRI, что было установлено в результате секвенирования). Полученную гибридную плазмиду pMTL21E:: srfAA клонировали в клетках штамма E. coli GM2163 для деметилирования, затем выделяли и внедряли в клетки бактерий B. velezensis БИМ В-439Д путем химической трансформации. Трансформантов отбирали на селективной среде с эритромицином, выделяли тотальную ДНК и проверяли наличие вставки в гене srfAA методом ПЦР.
Липопептиды из культуральной жидкости (КЖ) исходных и мутантных бактерий экстрагировали по методике, описанной в работе [12]. Полученный экстракт выпаривали, осадок растворяли в ацетонитриле.
Анализ содержания липопептидов проводили методом высокоэффективной жидкостной хроматографии (ВЭЖХ) на приборе Agilent 1260 Infinity “Agilent Technologies” (США) c гибридным квадрупольно-времяпролетным масс-спектрометром высокого разрешения Accurate Mass Q-TOF 6530 “Agilent Technologies” (США). Разделение компонентов осуществляли на колонке Zorbax Eclipse Plus C18 50 × 2.1 мм “Agilent Technologies” (США). Анализ проводили в режиме градиентного элюирования, в качестве подвижной фазы использовали подкисленную с помощью муравьиной кислоты (0.1%) воду (раствор А) и ацетонитрил с 0.1%‑ной муравьиной кислотой (раствор В). Линейный градиент включал: 0 мин – 30%-ный раствор В; 0.01–0.75 мин повышение концентрации раствора В до 65%; 0.75–10.0 мин повышение концентрации раствора В до 100%, 10–14 мин 100% раствор В; 14.0–15.1 мин снижение концентрации раствора В до 30%; 15.1–18.0 мин уравновешивание колонки 30%-ным раствором В. Объем инъекции 1 мкл, скорость потока 0.4 мл/мин. Сурфактин в экстракте КЖ определяли относительно по времени удерживания и масс-спектру коммерческого стандарта сурфактина S3523 “Sigma-Aldrich” (Германия). Для определения фенгицина и бацилломицина использовали данные масс-спектров, приведенные в работе [13].
Антагонистическую активность определяли методом лунок [14], для анализа использовали супернатант КЖ бактерий после фильтрования через фильтр с диаметром пор 0.2 мкм, в контрольных вариантах − стерильную воду.
Для сравнения отдельных генетических локусов использовали веб-ресурс antiSMASH (https:// antismash.secondarymetabolites.org). Для сравнения геномов использовали программу Mauve (v. 20150226) [15]. Рисунки генетических локусов создавали с помощью веб-сервиса Gene Graphics (https://katlabs.cc/genegraphics/).
Статистическую обработку данных проводили по стандартным методикам в программе LibreOffice Calc (версия: 6.1.3.2).
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Анализ общей структуры генома бактерий B. velezensis БИМ В-439Д в сравнении с изученными в этом отношении микроорганизмами позволил установить характерные особенности, свойственные микроорганизмам данной систематической группы, а также выявить отличительные особенности исследуемого штамма. Следует отметить, что на декабрь 2018 г. в свободном доступе ГенБанка NCBI депонированы полные нуклеотидные последовательности 56 геномов B. velezensis, 22 генома B. amyloliquefaciens и значительное число близких к ним представителей рода Bacillus. Такой объем доступной научной информации позволяет детально проанализировать полученные в результате секвенирования данные, а также свидетельствует о большом научном интересе, проявляемом в мире к бактериям данной систематической группы.
До проведения полногеномного секвенирования был осуществлен частичный сиквенс-анализ отдельных генетических детерминант (определены фрагменты генов 16S рРНК и гена gyrA), позволившие идентифицировать исследуемые бактерии как B. amyloliquefaciens subsp. plantarum. Следует отметить, что точную таксономическую принадлежность данного штамма (до уровня подвида) удалось установить только на основании нуклеотидной последовательности гена gyrA, поскольку гены 16S рРНК у целого ряда представителей рода Bacillus практически идентичны (в частности, B. subtilis subsp. subtilis, B. siamensis, B. vanillea, B. velezensis, B. methylotrophicus, B. amyloliquefaciens subsp. amyloliquefaciens и B. amyloliquefaciens subsp. plantarum) [16]. Следовательно, при отсутствии полной геномной последовательности, ген gyrA может использоваться в качестве молекулярно-генетического маркера для идентификации данных микроорганизмов. Согласно литературным данным, подвиды amyloliquefaciens и plantarum вида B. amyloliquefaciens отличаются между собой по ряду свойств. Например, только в геномах бактерий подвида plantarum выявлены локусы, определяющие синтез поликетидных антибиотиков макролактина и диффицидина/оскидиффицидина [17–19]. Кроме того, характерной особенностью бактерий B. amyloliquefaciens subsp. plantarum является способность колонизировать корни растений. Вступая в плотный контакт с растением, данные микроорганизмы способны обеспечивать его эффективную защиту от патогенов и активно влиять на его рост и развитие [20]. Подобные свойства характерны и для бактерий вида B. velezensis. Представителей этих двух таксономических групп в англоязычной литературе обозначают как “связанные с растением бактерии” (plant-associated bacterium). На основании анализа геномов и физиолого-биохимических особенностей бактерии B. amyloliquefaciens subsp. plantarum и B. velezensis не так давно были объединены в один вид B. velezensis [21]. Тем не менее, в литературе до сих пор ряд штаммов обозначают как B. amyloliquefaciens subsp. plantarum. При этом необходимо подразумевать их филогенетическое единство с бактериями B. velezensis.
Сравнительный анализ геномов типичных представителей вида B. velezensis и исследуемых бактерий позволил установить сходство в структурной организации их генетического материала в целом и отдельных генетических детерминант (рис. 1), что указывало на их близкое филогенетическое родство и принадлежность к одной систематической группе. Средняя нуклеотидная идентичность (ANIb) со штаммами G341 (CP011686), FZB42 (CP000560.1), AS43.3 (CP003838.1) составляла соответственно 99.19, 98.74 и 98.69%. Геном бактерий B. velezensis БИМ В-439Д по результатам сиквенс-анализа был депонирован в базу GenBank NCBI под номером CP032144. Определено, что геном представлен одной кольцевой хромосомой размером 3 978 954 п.н., содержащей 46.5% Г/Ц-пар и включающей 3 969 предсказанных генов, в том числе – 3769 белок-кодирующих последовательностей, 27 рРНК, 86 тРНК и 82 псевдогена (https://www.ncbi.nlm.nih.gov/nuccore/CP032144). При этом гены, кодирующие рибосомные РНК, как правило, расположены кластерами (5S, 16S, 23S). В хромосоме бактерий B. velezensis (например, штамм G341) и B. amyloliquefaciens subsp. plantarum (например, штамм UCMB5033, HG328253) встречается до 10 кластеров генов, кодирующих рРНК. В хромосоме исследуемых бактерий присутствовало 9 генетических локусов, определяющих синтез рРНК. Практически для всех бактерий этой таксономической группы характерно большое число генов, кодирующих синтез тРНК. Их количество варьирует от 51 у штамма B. velezensis SB1216 (CP015417) до 95 у штамма B. velezensis G341 (CP011686). Анализируемый штамм БИМ-439 содержал 86 генов, детерминирующих синтез тРНК. Следует отметить, что такое большое содержание в клетке компонентов аппарата трансляции является характерной особенностью целого ряда бактерий, природной средой обитания которых являются почвенные экосистемы [22]. Данные RAST-анализа генома B. velezensis БИМ В-439Д в сравнении с известными представителями этой группы бактерий показали, что в составе хромосомы исследуемых бактерий содержалось большое количество генетических детерминант, определяющих метаболизм углеводов, аминокислот, белков, жирных кислот, липидов, изопреноидов, а также синтез кофакторов, витаминов, простетических групп и пигментов (табл. 2). Это вполне закономерно, поскольку данные бактерии продуцируют без участия рибосом широкий спектр антибиотикоподобных соединений, для синтеза которых требуется активный углеводный обмен, в ходе которого образуется ключевые субстраты. Среди множества синтезируемых белков важную роль играют ферменты, определяющие поступление в клетку сахаров и этапы их окисления (пермеазы, гидролазы). В результате этих процессов образуются, в частности, пируват, 2-оксо-глутарат, оксалоацетат и ацетил-КоА, которые являются предшественниками синтеза аминокислот, жирных кислот, поликетидов и целого ряда других жизненно важных метаболитов. Следует отметить, что присутствующий в геноме исследуемых бактерий ген sacA, кодирующий фермент сахарозо-6-фосфатгидролазу, участвующий в первичном расщеплении сахаров, полностью идентичен таковым представителей B. amyloliquefaciens subsp. plantarum и B. velezensis.
Рис. 1.
Сравнительный анализ геномов бактерий B. velezensis БИМ В-439Д и B. velezensis. Идентичные локусы обозначены одинаковым цветом. Пики внутри локусов показывают степень идентичности расположенных в них генов. Пробелами обозначены уникальные локусы.
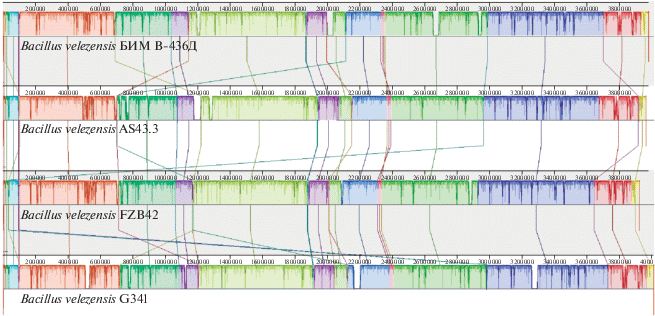
Таблица 2.
Сравнительный анализ систем клеточного метаболизма бактерий B. velezensis БИМ-439Д
| Системы метаболизма | Содержание генов в детерминантах штаммов | |||||
|---|---|---|---|---|---|---|
| БИМ В-439Д | FZB42* | GH1-13* | ||||
| всего | % | всего | % | всего | % | |
| Метаболизм белка | 182 | 6.79 | 166 | 6.07 | 196 | 7.18 |
| Метаболизм углеводов | 414 | 15.45 | 423 | 15.46 | 413 | 15.13 |
| Метаболизм аминокислот и их производных | 438 | 16.34 | 437 | 15.97 | 439 | 16.10 |
| Метаболизм нуклеозидов и нуклеотидов | 117 | 4.37 | 122 | 4.46 | 121 | 4.43 |
| Метаболизм жирных кислот, липидов, изопреноидов | 138 | 5.15 | 141 | 5.15 | 141 | 5.16 |
| Метаболизм ароматических соединений | 12 | 0.45 | 13 | 0.48 | 12 | 0.44 |
| Метаболизм РНК | 158 | 5.90 | 159 | 5.81 | 155 | 5.68 |
| Метаболизм ДНК | 79 | 2.95 | 112 | 4.09 | 98 | 3.59 |
| Метаболизм серы | 39 | 1.45 | 39 | 1.42 | 43 | 1.57 |
| Метаболизм фосфора | 29 | 1.08 | 31 | 1.13 | 31 | 1.13 |
| Метаболизм азота | 31 | 1.16 | 31 | 1.13 | 29 | 1.06 |
| Системы, определяющие баланс калия | 10 | 0.38 | 10 | 0.37 | 9 | 0.33 |
| Поступление железа в клетку | 29 | 1.08 | 31 | 1.13 | 29 | 1.06 |
| Синтез кофакторов, витаминов, простетических групп, пигментов | 233 | 8.69 | 241 | 8.81 | 230 | 8.42 |
| Клеточное деление и клеточный цикл | 53 | 1.98 | 52 | 1.90 | 54 | 1.98 |
| Подвижность и хемотаксис | 88 | 3.28 | 87 | 3.18 | 87 | 3.19 |
| Регуляция и сигнальные системы | 61 | 2.28 | 58 | 1.12 | 63 | 2.31 |
| Вторичный метаболизм | 5 | 0.19 | 12 | 0.44 | 5 | 0.18 |
| Состояние покоя и споруляции | 116 | 4.33 | 126 | 4.61 | 119 | 4.36 |
| Процессы дыхания | 75 | 2.80 | 73 | 2.67 | 74 | 2.71 |
| Ответ на стрессовые факторы | 107 | 3.99 | 106 | 3.87 | 112 | 4.10 |
| Системы транспорта через клеточную мембрану | 72 | 2.69 | 70 | 2.56 | 69 | 2.53 |
| Клеточная стенка, синтез капсулы | 133 | 4.96 | 131 | 4.81 | 136 | 4.98 |
| Клеточная защита | 61 | 2.28 | 65 | 2.38 | 65 | 2.38 |
| Всего | 2 680 | 100 | 2 736 | 100 | 2 730 | 100 |
Отличительные особенности организации генетического аппарата исследуемых бактерий были выявлены при анализе мобильных генетических элементов (IS-элементы и профаги). В их хромосоме присутствовали два варианта инсерционных элементов семейства IS3: 6 копий варианта IS150A (1285 п.н.) и 6 копий IS150B (1284 п.н.), отличающихся между собой заменами, вставками и делециями отдельных нуклеотидов, затрагивающими порядка 130 позиций. При этом, все обнаруженные IS-последовательности локализуются между генами, не нарушая открытых рамок считывания. Следует отметить, что подобного типа мобильные элементы встречаются в геномах бактерий B. amyloliquefaciens и B. velezensis [18, 19]. В частности, в хромосоме штамма B. velezensis G341 (выделен из 4-летних корней женьшеня в Корее) присутствует 10 копий IS150A и 4 копии IS150B, а в хромосоме B. velezensis FZB42 (выделен из зараженной патогенами почвы в Германии) обнаружено 3 копии только IS150A. В тоже время штамм B. amyloliquefaciens Y2, изолированный из ризосферы пшеницы в Китае, вообще их не содержит. Помимо IS-элементов в геноме бактерий B. velezensis БИМ В-439Д обнаружено 3 профага: два полноразмерных (интактных) и один дефектный. Один из полноразмерных профагов размером 37 558 п.н. (координаты в геноме 1 995 476–2 033 033), предположительно относящийся к п-орядку Caudovirales, не выявлялся в геномах близкородственных B. velezensis и B. amyloliquefaciens subsp. plantarum, однако обнаруживается в хромосомах некоторых других представителей подвида B. amyloliquefaciens subsp. amyloliquefaciens [17]. Последовательность встроенного фага содержала 37% Г/Ц-пар, что практически на 10% меньше, чем в хромосоме клетки хозяина – это свидетельствует в пользу того, что его интеграция в хромосому произошла относительно недавно. Второй интактный вирус размером 37 592 п.н. (координаты 2 647 315–2 684 906) по Г/Ц-составу практически не отличался от такового клетки хозяина и отдельные участки его генома встречались в хромосомах бактерий рода Bacillus. Нуклеотидная последовательность еще одного дефектного профага размером 31 632 п.н. (координаты 1 183 850–1 215 481) также присутствовала в хромосомах бактерий B. amyloliquefaciens KHG19, B. velezensis (штаммы CMT-6 и NJN-6) и B. subtilis B-1. Следует отметить, что в хромосомах практически всех бактерий содержатся интактные или дефектные вирусные геномы, наличие которых свидетельствует о происходящих процессах горизонтального переноса. В тоже время присутствие фагов в составе бактериальной хромосомы может иметь и функциональное значение, в частности, защищать клетку от проникновения других подобных вирусов, свободно циркулирующих во внешней среде.
В хромосоме бактерий B. velezensis БИМ В-439Д выявлено присутствие характерных для представителей этой таксономической группы протяженных локусов, определяющих синтез полифункциональных ферментных комплексов, ключевыми из которых являются, нерибосомные пептидсинтазы (NRPS) и поликетидсинтазы (PKS). Данные сложно организованные белковые молекулы обеспечивают без участия рибосом образование в клетке циклических липопептидов (сурфактин, бацилломицин, фенгицин), поликетидов (бациллен, диффицидин, макролактин), а также дипептида бацилизина и сидерофора бациллибактина, обладающих антимикробными свойствами (рис. 2, табл. 3). Выявленные генетические кластеры являются высоко консервативными и, несмотря на большие размеры, сходны по нуклеотидному составу таковым бактерий B. velezensis и B. amyloliquefaciens subsp. plantarum (идентичность составляла не менее 99%) [20, 23]. Кроме того, данные локусы имеют одинаковое генетическое окружение в хромосомах разных штаммов, что указывает на единство их происхождения и отсутствие рекомбинационных процессов, приводящих к изменению их локализации в геномах. Исключение составил ген bmyB, детерминирующий синтез В-субъединицы нерибосомной пептидсинтазы, участвующей в образовании бацилломицина. Нуклеотидная последовательность данной генетической детерминанты размером 16 122 п.н., кодирует фермент, значительно отличающийся от многих представителей B. velezensis аминокислотным составом функционального домена (3267–3777 аминокислотные остатки), определяющего аденилирование субстрата (сходство не превышает 48%).
Рис. 2.
Генетические карты хромосомных локусов бактерий B. velezensis БИМ В-439Д, определяющих синтез антимикробных метаболитов. Рядом с названием метаболита указан размер локуса. Светлые стрелки − гены, входящие в состав локуса, серые стрелки − гены, примыкающие к локусам. Направление стрелок соответствует направлению транскрипции. Внутри стрелок указаны названия генов. Цифры по краям локусов обозначают их координаты в геноме.
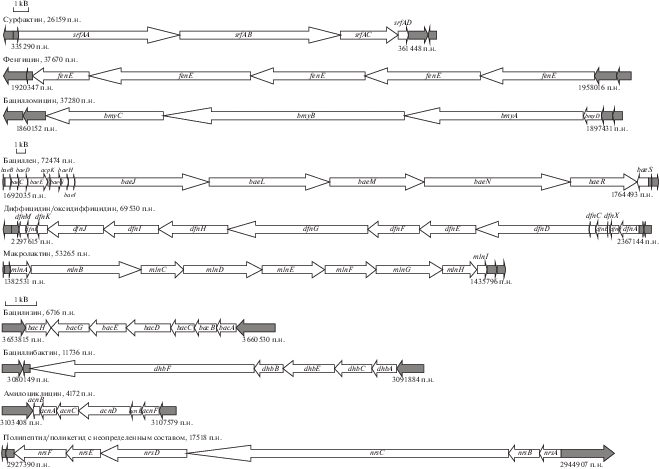
Таблица 3.
Характеристика хромосомных локусов бактерий B. velezensis БИМ В-439Д, определяющих синтез антимикробных метаболитов
| Метаболит | Гены, входящие в состав локуса | Координаты локуса в геноме |
Размер локуса, п.н. |
Сходство с гомологичными локусами бактерий* | Химическая природа метаболита | Уникальность локуса для бактерий B. velezensis | Подавляет развитие | ||
|---|---|---|---|---|---|---|---|---|---|
| штамм | № в ГенБанке NCBI | % | |||||||
| Сурфактин | srfABCD | 335 290–361 448 | 26 159 | B. velezensis G341 | CP011686.1 | 99 | Липопротеин | Характерен для бактерий рода Bacillus | Грибы, бактерии |
| Бацилломицин | bmyDABC | 1 860 152–1 897 431 | 37 280 | B. velezensis OSY-S3 | CP024706.1 | 99 | Липопротеин | Уникален | Грибы |
| Фенгицин | fenABCDE | 1 920 347–1 958 016 | 37 670 | B. velezensis BS-37 | CP023414.1 | 99 | Липопротеин | Уникален | Грибы |
| Бациллен | baeBCDE, acpk, baeGHIJLMNRS | 1 692 035–1 764 493 | 72 474 | B. velezensis G341 | CP011686.1 | 99 | Поликетид | Характерен для рода Bacillus | Бактерии |
| Диффицидин | dfnAYXBCDEFGHIJKLM | 2 297 615–2 367 144 | 69 530 | B. velezensis BS-37 | CP023414.1 | 99 | Поликетид | Уникален | Бактерии |
| Макролактин | mlnABCDEFGHI | 1 382 531–1 435 796 | 53 265 | B. velezensis BS-37 | CP023414.1 | 99 | Поликетид | Уникален | Бактерии |
| Соединение с неопределенным составом, NRS | nrsABCDEF | 2 927 390–2 944 907 | 17 518 | B. velezensis BS-37 B. velezensis FZB42 B amyloliquefaciens subsp. plantarum CC178 |
CP023414.1 CP000560.1 CP006845.1 |
99 | Предпологаемый пептид/поликетид, сидерофор | Уникален | Грибы, бактерии |
| Бациллибактин | dhbACEBF | 3 080 149–3 091 884 | 11 736 | B. velezensis G341 | CP011686.1 | 99 | Сидерофор | Характерен для рода Bacillus | Грибы, бактерии |
| Бацилизин | bacABCDEGH | 3 653 815–3 660 530 | 6716 | B. velezensis G341 | CP011686.1 | 99 | Дипептид | Характерен для рода Bacillus | бактерии |
| Амилоциклицин | acnBACDEF | 3 103 408–3 107 579 | 4172 | B amyloliquefaciens subsp. plantarum UCMB5113 | HG328254.1 | 99 | Циклический пептид, бактериоцин | Уникален | Грам+ бактерии |
Локусы, определяющие синтез сурфактина, бациллена, бацилизина и бациллибактина, присутствуют не только в геномах бактерий B. velezensis и B. amyloliquefaciens subsp. plantarum, но и в хромосомах других видов рода Bacillus (например, B. amyloliquefaciens subsp. amyloliquefaciens и B. subtilis). В геноме бактерий B. subtilis 168 присутствует также сходным образом организованный генетический кластер, определяющий синтез фенгицина (ppsABCDE), однако данный метаболит не синтезируется в клетках этих бактерий, в связи с отсутствием гена sfp, продукт которого необходим для экспрессии всех вышеуказанных соединений за исключением бацилизина [19]. Бактерии B. amyloliquefaciens subsp. amyloliquefaciens (к примеру, штаммы DSM7; S23; ATCC 15841) также не синтезируют фенгицин, поскольку данный кластер является неполным и представлен в хромосоме только двумя генетическими детерминантами (fenDE). В тоже время, хромосомные локусы, определяющие синтез макролактина и диффицидина являются видоспецифичными и характерны только для бактерий B. velezensis и B. amyloliquefaciens subsp. plantarum.
Как указывалось выше, для экспрессии генов, определяющих синтез липопептидов, поликетидов, сидерофора бациллибактина, необходимо присутствие 4'-фосфопантетеинилтрансферазы, продукта гена sfp. Данный фермент, обладая несколькими функциональными активностями, необходим на стадии инициации синтеза вышеуказанных метаболитов, а также обеспечивает их превращение в активную форму после завершения синтеза [19]. Этот ген (675 п.н.) присутствует в геноме бактерий B. velezensis БИМ В-439Д и имеет сходную с другими представителями этой таксономической группы локализацию (за srf-опероном, координаты 365 837–366 511). Данная детерминанта полностью идентична нуклеотидным последовательностям генов sfp бактерий B. velezensis G341 (CP011686.1), Bacillus sp. Bs-916 (CP009611.1) и B. amyloliquefaciens subsp. plantarum UCMB5033 (HG328253.1) и определяет синтез белка размером 25.5 кДа, идентичного на 100% с таковым бактерий рода Bacillus (WP_007410244.1).
Кроме вышеуказанных генетических детерминант, характерных практически для всех представителей B. velezensis и B. amyloliquefaciens subsp. plantarum, в хромосоме исследуемого штамма выявлен локус размером 17 518 п.н. (рис. 2, табл. 3), состоящий из шести генов, два из которых детерминируют синтез поликетидсинтазы, а один определяет синтез нерибосомной пептидсинтазы. Поскольку химический состав метаболита (обозначен как nrs) не определен, считается, что он может иметь пептидную/поликетидную природу и синтезируется без участия рибосом. Сравнение данного локуса с известными, депонированными в ГенБанк NCBI, показал, что сходным образом организованная нуклеотидная последовательность встречается только в геноме трех штаммов (B. velezensis BS-37, B. velezensis FZB42 и B. amyloliquefaciens subsp. plantarum CC178). В состав данного локуса входят гены, в значительной степени отличающиеся по Г/Ц-составу от остальных хромосомных детерминант. Доля Г/Ц-пар в данном локусе варьирует от 27 до 32%, тогда как среднее число Г/Ц-пар в хромосоме исследуемого штамма и у других представителей B. velezensis и B. amyloliquefaciens subsp. plantarum соответствует значению 46.5%. Данный факт свидетельствует о чужеродном происхождении данного генетического кластера, попавшего в геном в результате горизонтального переноса [24]. Установлено, что гены, входящие в этот локус экспрессируются и обеспечивают образование в клетке сложного соединения, обладающего функцией сидерофора и отвечают за транспорт железа в клетку-хозяина, отсутствие которого в ризосфере препятствует развитию грибов и бактерий [19].
В отличие от всех вышеописанных метаболитов, только бактериоцин амилоциклицин, представляющий собой циклический пептид, синтезируется с участием рибосом и его образование в клетке зависит от экспрессии шести генов (локус размером 4172 п.н. (рис. 2)), присутствие которых также характерно для большинства представителей B. velezensis и B. amyloliquefaciens subsp. plantarum [25].
Таким образом, в геноме бактерий B. velezensis БИМ В-439Д было обнаружено 10 генетических локусов общей протяженностью 336 520 п.н. (8.5% от всего генома), детерминирующих синтез антимикробных соединений. Выявленные детерминанты являются высоко консервативными и присутствуют в геномах многих представителей B. velezensis и B. amyloliquefaciens subsp. plantarum, что дает основание предположить их функциональную активность в клетках исследуемого штамма.
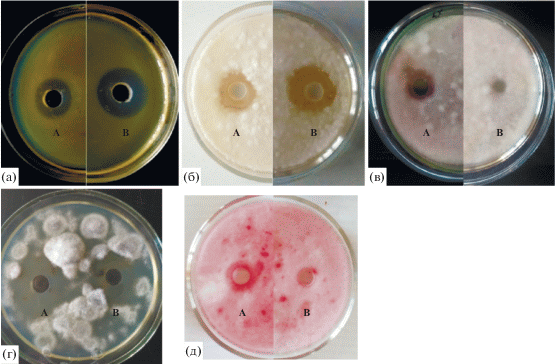
В ходе выполнения данной работы была изучена способность бактерий B. velezensis БИМ В-439Д синтезировать полифункциональные липопептиды. Ранее для штамма B. velezensis FZB42 было установлено, что его антифунгальная активность зависит от бациллибактина и фенгицина, тогда как сурфактин не влиял на их способность подавлять развитие патогенного гриба Fusarium oxysporum. Подобные результаты были получены и для штамма B. subtilis 916 [11, 23]. В научных публикациях сурфактину отводится ключевая роль в образовании биопленок и адгезии, способствующей взаимодействию микроорганизмов с растением [23]. Полагают, что сурфактин, обеспечивая образование биопленки при поверхностной колонизации корней растений, защищает синтезирующих его бактерий от других микроорганизмов, в том числе, патогенных. Для установления роли сурфактина в антифунгальной активности исследуемого штамма методом направленного мутагенеза были отобраны варианты с нарушенным синтезом этого метаболита (нарушен srf-оперон в результате инсерции в ген srfAA). Посредством масс-спектрометрического анализа было установлено, что в КЖ штамма дикого типа после его культивирования в течение 24 ч выявляются две формы сурфактина (с разной длиной углеродной цепи – С14 и С15), небольшое количество фенгицина (С16) и следовые количества бацилломицина (С14). После 48 ч культивирования в КЖ бактерий возрастало количество фенгицина С16, появлялся фенгицин С17, бацилломицин не обнаруживался. В тоже время в КЖ мутантного штамма srf– вне зависимости от времени культивирования отсутствовал сурфактин, но после 48 ч выращивания достоверно выявлялись фенгицин С16 и С17 и бацилломицин С14 (табл. 4, рис. 3). Полученные данные свидетельствуют об отсутствии синтеза сурфактина у мутантных бактерий и его повышенной способности синтезировать фенгицин и бацилломицин. Анализ антагонистической активности позволил установить, что у мутантных бактерий увеличилась антибактериальная активность в отношении фитопатогенных бактерий Pseudomonas. syringae pv. syringae, Xanthomonas. campestris pv. campestris, Agrobacterium tumefaciens, сохранилась антифунгальная активность по отношению к грибам Penicillium expansum, Alternaria tenuis, Botrytis cinerea, Bipolaris sorokiniana и заметно снизилась способность подавлять рост грибов рода Fusarium (табл. 5, рис. 4). Таким образом, при отсутствии сурфактина в КЖ штамма B. velezensis БИМ В-439Д наблюдались специфичные изменения антимикробной активности бактерий, что свидетельствует о влиянии этого метаболита на проявление антагонизма. Полученные результаты могут указывать на различия в регуляции синтеза липопептидов у исследуемых бактерий по сравнению с близкородственными штаммами B. velezensis FZB42 и B. subtilis 916.
Таблица 4.
Продукция липопептидов исходным и мутантным вариантами бактерий B. velezensis БИМ В-439Д
| Липопептид | Масса/заряд | Изоформа | Время удержания, мин | Интенсивность* | |||
|---|---|---|---|---|---|---|---|
| исходный штамм | мутант srf – | ||||||
| 24 ч | 48 ч | 24 ч | 48 ч | ||||
| Сурфактин | 1021.66749 | С14[M+H]+ | 11.146 | 12.597 | 13.680 | – | – |
| 1035.68314 | С15[M+H]+ | 11.478 | 22.862 | 21.822 | – | – | |
| Фенгицин | 1462.79593 | С16[M+H]+ | 6.389 | 0.850 | 1.210 | 0.676 | 2.137 |
| 1476.81158 | С17[M+H]+ | 6.622 | – | 0.403 | – | 0.577 | |
| Бацилломицин | 1020.51278 | С14[M+H]+ | 4.725 | 0.324 | – | – | 1.232 |
Рис. 3.
Хроматограммы липопептидов. продуцируемых диким (а) и мутантным штаммами (б) бактерий B. velezensis БИМ В-439Д за 24 и 48 ч культивирования.
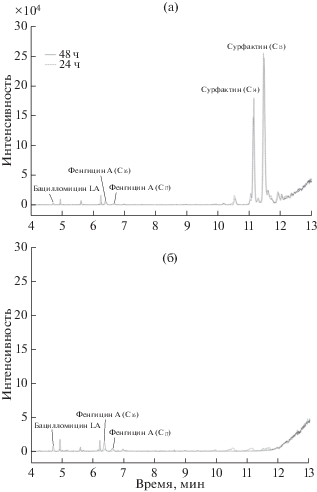
Таблица 5.
Антимикробная активность бактерий B. velezensis БИМ В-439Д
| Патогенный микроорганизм | Зоны задержки роста патогена под действием КЖ, мм | |
|---|---|---|
| исходный штамм | инсерционный мутант srf – | |
| Pseudomonas. syringae var. syringae | 38 ± 0.3 | 39 ± 0.5 |
| Xanthomonas campestris pv. campestris | 24 ± 0.8 | 27 ± 0.7 |
| Agrobacterium tumefaciens | 23 ± 0.9 | 25 ± 0.6 |
| Fusarium graminearum | 30 ± 0.9 | 27 ± 1.0 |
| Fusarium oxysporum | 28 ± 1.0 | 19 ± 0.7 |
| Fusarium culmorum | 25 ± 0.7 | 17 ± 0.6 |
| Fusarium avenaсeum | 23 ± 0.9 | 15 ± 0.9 |
| Botrytis cinerea | 27 ± 0.9 | 24 ± 0.7 |
| Alternaria tenuis | 24 ± 0.3 | 23 ± 1.0 |
| Penicillium expansum | 23 ± 0.4 | 25 ± 0.8 |
| Bipolaris sorokiniana | 33 ± 0.6 | 31 ± 0.6 |
| Phoma betae | 31 ± 0.5 | 27 ± 0.4 |
Рис. 4.
Тест на антимикробную активность КЖ штаммов дикого типа слева на чашке Петри и мутантных B. velezensis БИМ В-439Д (справа). Зоны задержки роста патогенных бактерий X. campestris pv. сampestris (а) и патогенных грибов: P. expansum (б), F. сulmorum (в); Ph. betae (г); F. avenaceum (д).
Наличие полной нуклеотидной последовательности хромосомы бактерий B. velezensis БИМ В-439Д и выявленные особенности в организации и локализации мобильных генетических элементов и отдельных генетических детерминант могут служить надежным генетическим паспортом штамма при его коммерческом использовании.
Список литературы
Borriss R. // Bacteria in Agrobiology: Plant Growth Responses / Ed. D.K. Maheshwari. Heidelberg: Springer, 2011. P. 41–76.
Chen X.H., Koumoutsi A., Scholz R., Borriss R. // J. Mol. Microbiol. Biotechnol. 2009. V. 16. № 1–2. P. 14–24. https://doi.org/10.1159/000142891
Bullock W.O., Fernandez J.M., Short J.M. // Biotechniques. 1987. V. 5. № 3. P. 376–379.
Palmer B.R., Marinus M.G. // Gene. 1994. V. 143. № 1. P. 1–12. https://doi.org/https://doi.org/10.1016/0378-1119(94)90597-5
Vieira J., Messing J. // Gene. 1982. V. 19. № 3. P. 259–268.
Steve P. Cbambers, Sue E. Prior, David A. Barstow, Nigel P. Minton // Gene. 1988. V. 68. № 1. P. 139–149.
Миллер Дж. Эксперименты в молекулярной генетике / Ред. С.И. Алиханяна. М.: Мир, 1976. 436 с.
Дудка И.А., Вассер С.П., Элланская И.А., Коваль Э.3., Горбик Л.Т., Никольская Е.А. и др. // Методы экспериментальной микологии: справочник / Ред. В.И. Билай. Киев: Наукова думка, 1982. 429 с.
Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии: молекулярное клонирование / Pед. А.А. Баев, К.Г. Скрябина. М.: Мир, 1984. 479 с.
Koumoutsi A., Chen X.H., Henne A., Liesegang H., Hitzeroth G., Franke P., Vater J., Borriss R. // J. Bacteriol. 2004. V. 186. № 4. P. 1084–1096.
Richter M., Rosselló-Móra R., Oliver Glöckner F., Peplies J. // Bioinformatics. 2016. V. 32. № 6. P. 929–931.
Yokota K., Yatsuda M., Miwa E., Higuchi K.J. // J. ISSAAS. 2012. V. 18 № 1. P. 70–75.
Yang H., Li X., Li X., Yu H., Shen Z. // Anal. Bioanal. Chem. 2015. V. 407. № 9. P. 2529–2542. https:// doi.org/https://doi.org/10.1007/s00216-015-8486-8
Сэги Й. Методы почвенной микробиологии. М.: Колос, 1983. 253 с.
Darling A.E., Mau B., Perna N.T. // PLoS ONE. 2010. V. 5. № 6. art. no. e11147. https://doi.org/.https://doi.org/10.1371/ journal.pone.0011147
Fan B., Blom J., Klenk Hans-P., Borriss R. // Frontiers in Microbiol. 2017. V. 8. № 22. P. 1–15.
Niazi A., Manzoor Sh., Asari S., Bejai S., Meijer J., Bongcam-Rudloff E. // PLOS ONE. 2014. V. 9. № 8. art. no. e104651. https://doi.org/.https://doi.org/10.1371/journal.pone.0104651
Rückert Ch., Blom J., Chen X.H., Revac O., Borriss R.J. // Biotechnol. 2011. № 155. P. 78–85.
Chen X.H., Koumoutsi A., Scholz R., Eisenreich A., Schneider K., Heinemeyer I. et al. // Nature Biotechnology. 2007. V. 25. P. 1007–1014. Published online 19 August 2007; https://doi.org/10.1038/nbt1325
Borriss R., Chen X.H., Rückert Ch., Blom J., Becker A., Baumgarth B., Fan B., Pukall R., Schumann P., Spröer C., Junge H., Vater J., Pühler A., Klenk Hans-P. // International J. Systematic and Evolutionary Microbiology. 2011. P. 1786–1801.
Christopher A. Dunlap, Soo-Jin Kim, Soon-Wo Kwon, Alejandro P. Rooney // International J. Systematic and Evolutionary Microbiology. 2016. V. 66. P. 1212–1217.
Klappenbach J.A., Dunbar J.M., Schmidt T.M. // Appl. Environ. Microbiol. 2000. V.66. P. 1328–1333.
Luo C., Liu X., Zhou H., Wang Z., Chen X.H. // Appl. Environ Microbiol. 2015. V. 81. № 1. P. 422–431.
Reva O.N, Tümmler B. // BMC Bioinformatics. 2005. V. 6. № 251. P. 1–12.
Scholz R., Vater J., Budiharjo A., Wang Z., He Y., Dietel K., Schwecke T., Herfort S., Lasch P., Borriss R. // J. Bacteriol. 2014. V. 196. № 10. P. 1842–1852.
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


