Прикладная биохимия и микробиология, 2021, T. 57, № 5, стр. 450-457
Получение и свойства рекомбинантного протеолитически активного гомолога сериновой пептидазы SerPH122 Tenebrio molitor
В. Ф. Терещенкова 1, Н. И. Жиганов 2, Ф. И. Акентьев 3, 4, И. И. Губайдуллин 3, 4, Д. Г. Козлов 4, Н. В. Беляева 2, И. Ю. Филиппова 1, Е. Н. Элпидина 5, *
1 Московский государственный университет им. М.В. Ломоносова, химический факультет
119991 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова, биологический факультет
119991 Москва, Россия
3 НИЦ “Курчатовский институт” – ГосНИИгенетика, Курчатовский геномный центр
117545 Москва, Россия
4 Национальный исследовательский центр “Курчатовский институт”
123182 Москва, Россия
5 Московский государственный университет им. М.В. Ломоносова, Научно-исследовательский институт
физико-химической биологии им. А.Н. Белозерского
119991 Москва, Россия
* E-mail: elp@belozersky.msu.ru
Поступила в редакцию 02.04.2021
После доработки 15.04.2021
Принята к публикации 22.04.2021
Аннотация
Псевдоферменты – гомологи активных ферментов, имеют аминокислотные замены в активном центре и в связи с этим обычно не обладают ферментативной активностью. В данной работе получен рекомбинантный пробелок (proSerPH122) гомолога сериновых пептидаз семейства S1 мучного хрущака (Tenebrio molitor) в штамме-продуценте дрожжей Komagataella kurtzmanii. В процессе секреции в дрожжах целевой белок, содержащий His6-tag, продуцировался в гликозилированной форме. Изучение свойств проводили параллельно для гликозилированной и дегликозилированной форм. Для изучения энзиматических свойств полученный в виде пробелка гомолог proSerPH122 с заменой Ser активного центра на Thr процессировали предварительно трипсином. Показано, что процессированный зрелый гомолог SerPH122 обладал невысокой, но достоверно детектируемой активностью по хромогенному субстрату Suc-Ala-Ala-Pro-Phe-pNA, и эта активность не зависела от уровня его гликозилирования.
Сериновые пептидазы семейства S1 химотрипсина по классификации MEROPS [1] представляют обширную группу протеолитических ферментов. Родоначальником семейства является химотрипсин, активный центр которого составляет каталитическая триада аминокислотных остатков His57-Asp102-Ser195 (нумерация во введении дана по химотрипсину А Bos taurus, NCBI ID XP_003587247 [2]). Субстратная специфичность сериновых пептидаз обусловлена аминокислотными остатками S1 субсайта связывания, сформированного 189, 216 и 226 остатками [3, 4]. В зависимости от состава S1-субсайта выделяют химотрипсины A/B (Ser-Gly-Gly), химотрипсины С (Ser-Gly-Val), трипсины (Asp-Gly-Gly), сериновые эластазы (Gly-Val-Ser/Thr), коллагеназы (Gly-Gly-Asp).
Подробный биоинформатический анализ последовательностей, широко используемый в последние годы, показал, что наряду с активными ферментами, способными осуществлять катализ, существует целый ряд псевдоферментов, или псевдопептидаз, которые являются структурными гомологами ферментов, но содержат замены аминокислотных остатков активного центра, что приводит в большинстве случаев к полному отсутствию каталитической активности [5] или значительному еe снижению [6]. Данных о функциях многочисленной группы псевдоферментов в настоящее время очень мало, и они касаются, в основном, их биоинформатической характеристики [7, 8]. Псевдоферменты функционируют часто как компоненты сложных комплексов. Считается, что они могут принимать участие в регуляции своих каталитически-активных гомологов, выступая в качестве белков-партнеров, участвовать в регуляции метаболических и сигнальных путей, часто ассоциированных с патологическими состояниями, что делает их потенциальными мишенями для разработки терапевтических агентов наряду с активными ферментами [9].
Растущая важность и актуальность изучения псевдоферментов подчеркивается как широким спектром процессов, в которых они участвуют, так и их многочисленностью. По мере выявления большего числа псевдоферментов возрастает необходимость в понимании их структуры, филогенетических связей, функциональной роли.
Исследования последних лет свидетельствуют о том, что большое количество псевдоферментов характерно для насекомых. Так, в частности, у Anopheles gambiae и Drosophila melanogaster, а также и у многих других родственных видов было найдено большое количество как неактивных, так и активных трипсиноподобных сериновых пептидаз, причем эволюция неактивных пептидаз и их новые функции предположительно оказались выгодными для насекомых [7]. Примером такого взаимодействия может служить белок CASPS18, найденный у комара Aedes aegypti и являющийся регулятором своего активного паралога каспазы CASPS19 [10]. Это предположение можно распространить на другие организмы, в том числе и человека. В связи с этим, большой интерес для изучения спектра и свойств псевдопептидаз могут представлять собой насекомые семейства Tenebrionidae, в частности, Tenebrio molitor, являющееся хорошо изученной биохимической моделью благодаря своим крупным размерам.
Ранее в лаборатории был получен обширный транскриптом из кишечника личинок T. molitor [11], анализ которого позволил выявить SerPH122 (Serine Peptidase Homolog) – белок-гомолог сериновых пептидаз из семейства S1 химотрипсина. Для детальной характеристики свойств гомолога SerPH122, а также для определения природных молекул или субстратов, способных к специфическому взаимодействию с SerPH122, необходимо получить рекомбинантный препарат исследуемого белка. Ранее было показано [12, 13], что метилотрофные дрожжи Komagataella kurtzmanii представляют собой основу системы экспрессии, альтернативную K. phaffii (ранее Pichia pastoris) и могут осуществлять эффективный биосинтез и секрецию рекомбинантных ферментов.
Цель работы – конструирование штамма-продуцента рекомбинантного пробелка-гомолога proSerPH122 T. molitor, получение белка proSerPH122, очистка профермента, его дегликозилирование, процессинг пробелка и тестирование наличия протеолитической активности у зрелого белка.
МЕТОДИКА
Конструирование плазмид и штаммов. Фрагмент ДНК, кодирующий рекомбинантный пробелок-гомолог proSerPH122, получали с помощью ПЦР-амплификации. Матрицей для амплификации служила плазмида pAL-TA-31-1 (“Евроген”, Россия), содержавшая кДНК пробелка-гомолога proSerPH122 (GenBank MW882981). ПЦР проводили с использованием ДНК-полимеразы Phusion (“Thermo Fisher Scientific”, США) и праймеров pNcoI (5'-tataccatggaaaagagatctaagcctggagctcgtataatt-3') и pXhoI (5'- taactcgagttaatggtgatggtgatgatggggattgatgatggttctgat-3'). Помимо сайтов рестрикции NcoI и XhoI (подчеркнуты в составе соответствующих праймеров) в ходе ПЦР в состав амплифицированного фрагмента ДНК были введены дополнительные последовательности, удлинившие N-конец рекомбинантного пробелка proSerPH122 на остаток серина и С-конец – на пептид His6-tag.
Амплифицированный фрагмент ДНК клонировали в вектор pPH727-AOX727 [14] по сайтам рестрикции NcoI/XhoI как описано ранее [13], в результате получили плазмиду pPH727-proSerPH122. Корректность последовательности структурного гена proSerPH122 в составе плазмиды pPH727-proSerPH122 подтверждена секвенированием. Регуляция экспрессии целевого гена находилась под контролем индуцируемого метанолом промотора гена АОХ1 штамма ВКПМ Y-727 K. kurtzmanii (pAOX727) [14], секрецию целевого белка направляла лидерная область artHH, как описано ранее [13]. В процессе клонирования и амплификации плазмиды pPH727-proSerPH122 использовали клетки Top10 Escherichia coli (“Invitrogen”, США). Культивировали при температуре 37°С на среде LB с добавлением ампициллина в концентрации 50 мкг/мл.
Трансформацию клеток реципиентного штамма дрожжей K. kurtzmanii Y727his4Δ проводили по методике, описанной в работе [14]. Перед трансформацией интегрируемый фрагмент ДНК линеаризовали путем гидролиза плазмиды pPH727-proSerPH122 по рестрикционным сайтам MluI.
Для культивирования полученных трансформантов использовали среду YPGM следующего состава (%): дрожжевой экстракт – 1.0 (0207; “BioSpringer”, Франция), пептон соевый – 2.0 (P140; “Amresco”, США), глицерин – 0.5 (“Panreac”, Испания), метанол – 1.0 (техническая марка “А” ГОСТ 2222-95, Россия). Метанол вносили в среду до конечной концентрации 1% при засеве дрожжей и далее с периодичностью в 24 ч. Наработку образцов культуральной жидкости (КЖ), содержащих белок proSerPH122, проводили в течение 72 ч при температуре 29°С с использованием ротационного шейкера со скоростью вращения 250 об./мин.
Электрофорез белков. Электрофорез белков КЖ трансформантов pPH727-proSerPH122 проводили в 15%-ном ПААГ в восстанавливающих условиях (ДДС-ПААГ) как описано ранее [13]. Использовали предокрашенные белковые маркеры “Thermo Fisher Scientific” (США). При подготовке образцов перед нанесением на гель белки концентрировали как описано ранее [15]. На дорожки наносили препараты, сконцентрированные из 100 мкл КЖ.
Очистка рекомбинантного пробелка proSerPH122 T. molitor. Очистку рекомбинантного пробелка из КЖ дрожжей проводили с использованием металл-хелатной аффинной хроматографии, как описано ранее [13], с тем отличием, что перевод очищенного белка из буфера А в воду mQ осуществляли методом ультрафильтрации с использованием ультрафильтрационной ячейки (“Amicon”, США) и мембраны с порогом отсечения 3 кДа (Millipore, США). Непосредственно после обессоливания раствора белка чистоту препарата оценивали с помощью электрофореза в ПААГ с Na-ДДС. Концентрацию измеряли фотометрически при длине волны 280 нм на спектрофотометре NanoDrop 1000 (“Thermo Fisher Scientific”, США). Препарат замораживали в жидком азоте и лиофилизовали на приборе FreeZone (“Labconco”, США).
Дегликозилирование рекомбинантного пробелка proSerPH122 T. molitor с использованием эндогликозидазы-Н. Для реакции дегликозилирования использовали лиофилизованный препарат, полученный на предыдущем этапе. Дегикозилирование проводили с помощью эндогликозидазы Endo H (“New England Biolabs”, США) в соответствии с рекомендациями производителя. Обессоливание и перевод белка в воду mQ осуществляли методом ультрафильтрации, как описано выше, концентрацию измеряли фотометрически при длине волны 280 нм на спектрофотометре NanoDrop 1000. Препарат замораживали в жидком азоте и лиофилизовали на приборе FreeZone (“Labconco”, США).
Масс-спектрометрический анализ. Масс-спектрометрический анализ очищенного белка проводили на MALDI-времяпролетном масс-спектрометре UltrafleXtreme (“Bruker Daltonics”, Германия), оснащенном УФ-лазером (Nd). Масс-спектры получены в линейной моде режима положительных ионов с использованием рефлектрона. Точность измеренных средних масс составляла 10 Да. Для расчета значений масс белков использовали программный пакет Vector NTI (“Thermo Fisher Scientific”, США).
Биоинформатический анализ последовательности SerPH122 T. molitor. Полную аминокислотную последовательность белка SerPH122 T. molitor, полученную в результате трансляции кодирующей ее последовательности GenBank MW882981, выравнивали с родоначальником семейства S1 химотрипсином быка NCBI ID XP_003587247 с использованием сервера для множественного выравнивания последовательностей Clustal Omega [16]. Анализ и визуализацию активного центра и субстратсвязывающего субсайта проводили в GeneDoc [17]. Сигнальный пептид предсказывали на сервере SignalP-5.0 [18].
Процессинг пробелка proSerPH122 T. molitor. Для активации proSerPH122 лиофилизированные гликозилированный и дегликозилированный препараты пробелка растворяли до концентрации 15 мкг/мл в 0.1 М ацетатно-фосфатно-боратном универсальном буфере pH 7.9, добавляли раствор трипсина до конечной концентрации 0.25 мкг/мл и инкубировали в течение 60 мин при температуре 37°C в термостатируемом шкафу (“Binder”, Германия).
Определение ферментативной активности рекомбинантного гомолога SerPH122 T. molitor. Для тестирования ферментативной активности исходного пробелка и процессированного гомолога SerPH122 в качестве хромогенного субстрата использовали N-сукцинил-Ala-Ala-Pro-Phe-(п-нитроанилид) – Suc-Ala-Ala-Pro-Phe-pNA (“Bachem”, Швейцария). Реакцию и расчет ферментативной активности проводили по опубликованной ранее методике [19]. Измерение активности проводили при рН 7.9, и 5.6 в 0.1 М ацетатно-фосфатно-боратном универсальном буфере [20].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Получение, очистка и структурное исследование proSerPH122. Биосинтез рекомбинантного пробелка proSerPH122 проводили с использованием экспрессионной системы K. kurtzmanii, зарекомендовавшей себя в качестве эффективной альтернативы широко распространенной системе экспрессии на основе K. phaffii, ранее P. pastoris (“Invitrogen”, США) [13, 14 ].
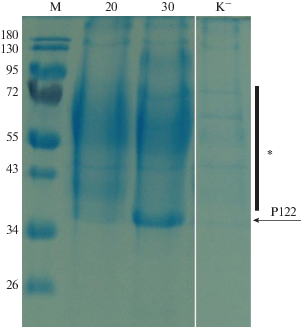
С целью получения штамма-продуцента рекомбинантного пробелка proSerPH122 клетки реципиентного штамма дрожжей K. kurtzmanii Y727his4Δ трансформировали экспрессионной конструкцией pPH727-proSerPH122. Трансформанты выращивали в стеклянных колбах в условиях индукции метанолом при температуре 20 и 30°С в течение 4 и 3 сут соответственно. Полученные образцы КЖ анализировали с использованием электрофореза в ПААГ с Na-ДДС. Результат представлен на рис. 1. В образцах КЖ были обнаружены белковые продукты, представленные на электрофореграмме в виде отдельной полосы, расположенной в области 36 кДа, и в виде диффузного шмера, распределённого в области мол. масс от 40 до 80 кДа. Окраска этих продуктов была существенно более интенсивной в образце с индукцией при 30°С, в образце реципиентного штамма K. kurtzmanii Y727his4Δ (отрицательный контроль) такие продукты отсутствовали (рис. 1). Вероятно, несоответствие полученных молекулярных масс и расчетной (26.6 кДа) обусловлено гликозилированием.
Рис. 1.
Электрофорез белков КЖ трансформантов pPH727-proSerPH122. М – маркеры молекулярной массы; К– – отрицательный контроль (Y727his4Δ); 20 и 30 – образцы целевого белка proSerPH122, полученные при культивировании трансформантов pPH727-proSerPH122 при температуре 20 и 30°С соответственно. Стрелкой показано расположение мажорной полосы целевого гликозилированного белка (Р122). *Обозначена область распределения гипергликозилированных форм целевого белка.
Последовательность рекомбинантного белка proSerPH122 содержала два потенциальных сайта N-гликозилирования: N48AT и N118ЕT (нумерация остатков соответствует зрелому активированному ферменту). Обработка белка эндогликозидазой Endo H и последующий анализ продуктов реакции методом электрофореза в ПААГ с Na-ДДС (рис. 2) и масс-спектрометрии (рис. 3 и табл. 1) показал, что секреция целевого белка действительно осуществлялась в форме N-гликозилированного продукта. В частности, согласно данным электрофореза, в результате обработки продукта секреции Endo H молекулярная масса мажорной полосы белка снижалась с 36 до 28 кДа (рис. 2). При этом интенсивность мажорной полосы значительно увеличивалась, а в области гипергликозилирования окраска практически исчезала.
Рис. 2.
Электрофоретический контроль дегликозилирования очищенного препарата proSerPH122 с использованием эндогликозидазы Endo H. М – маркеры молекулярной массы; 1 – препарат белка proSerPH122 после очистки методом металл-хелатной аффинной хроматографии; 2 – препарат очищенного белка proSerPH122 после дегликозилирования. Стрелками показано расположение мажорных полос гликозилированного (Р122) и дегликозилированного (P122dg) белков.
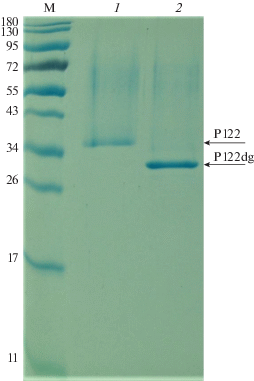
Рис. 3.
Масс-спектры образцов очищенного белка proSerPH122 перед дегликозилированием (а) и после дегликозилирования с помощью эндогликозидазы Endo H (б).
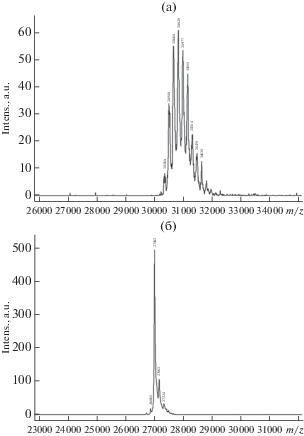
Таблица 1.
Расчетная и экспериментальная молекулярные массы (мол. массы) различных производных дегликозилированного белка proSerPH122dg
| Экспериментальная мол. масса | Расчетная мол. масса различных модификаций | ||||
|---|---|---|---|---|---|
| – | (NAG)2 | (NAG)2 desHisCend | (NAG)2 Man | (NAG)2 Man2 | |
| 26591 | +(221 – 18) × 2 | +(221 – 18) × 2 – ‒ (155 – 18) | +(221 – 18) × 2 + + (180 – 18) | +(221 – 18) × 2 + + (180 – 18) × 2 | |
| 26 865 | 26 860 | ||||
| 27 003 | 26 997 | ||||
| 27 163 | 27 159 | ||||
| 27 324 | 27 321 | ||||
По данным масс-спектрометрии средняя молекулярная масса спектра белковых продуктов снижалась с примерно 31 до 27 кДа (рис. 3). Анализ производных целевого белка (табл. 1) позволил предположить, что мажорным дегликозилированным продуктом является белок, содержащий 2 остатка N-ацетиглюкозамина (MМ = 27 003, табл. 1). Появление такого продукта ожидалось в качестве результата полного дегликозилирования белка по двум сайтам, содержащим углеводные компоненты, связанные с остатками аспарагина N-гликозидной связью. Наиболее вероятно, что другими вариантами были производные мажорного дегликозилированного продукта, содержащие 1 или 2 остатка маннозы Man и Man2 (табл. 1), являющиеся потенциальными продуктами О-гликозилирования белка, и продукт деградации белка, у которого отсутствовал концевой гистидин (desHisCend).
Энзиматические свойства SerPH122. Полная последовательность белка-гомолога SerPH122 (GenBank MW882981) содержит сигнальный пептид (рис. 4, выделен синей скобкой), который по современным представлениям отщепляется в процессе секреции, а пропептид (рис. 4, выделен зеленой скобкой), заканчивающийся остатком аргинина (рис. 4, отмечен красной стрелкой), обычно подвергается дальше процессингу с помощью трипсина [21, 22]. По сравнению с родоначальником семейства S1 химотрипсином быка (NCBI ID XP_003587247), имеющим каталитическую триаду His-Asp-Ser, гомолог SerPH122 содержит в активном центре аминокислотные остатки His-Asp-Thr (рис. 4, отмечены красным). Замена существенного для катализа аминокислотного остатка Ser на синонимичный остаток Thr у гомолога все же позволила предположить наличие у него протеолитической активности.
Рис. 4.
Множественное выравнивание последовательностей SerPH122 T. molitor и родоначальника семейства S1 химотрипсина быка (chtr_Btaur, NCBI ID XP_003587247). Сигнальный пептид SerPH122 выделен синей скобкой, пропептид – зеленой скобкой, предположительный сайт процессинга трипсином отмечен красной стрелкой. Аминокислотные остатки активного центра выделены красным, субстрат связывающего субсайта S1 – желтым.

Для поиска возможных энзиматических свойств полученный в виде пробелка гомолог proSerPH122 процессировали предварительно трипсином. Оказалось, что протеолитическая активность зрелого процессированного гомолога SerPH122 проявлялась по хромогенному пептидному субстрату Suc-Ala-Ala-Pro-Phe-pNA, который широко используется для тестирования активности химотрипсина. Обе процессированные формы (гликозилированная, SerPH122g и дегликозилированная, SerPH122dg) были протеолитически активны и гидролизовали субстрат Suc-Ala-Ala-Pro-Phe-pNA (рис. 5). Интересно, что обе непроцессированные формы пробелка proSerPH122 (гликозилированная и дегликозилированная) также обладали протеолитической активностью, однако она была в 1.5 раза ниже, чем у процессированных зрелых форм (рис. 5). Полученный результат может свидетельствовать о том, что гликозилирование не влияет на проявление активности гомолога. По сравнению с пептидазой T. molitor из семейства S1 SerP38 (NCBI ID QRE01764) [13], удельная активность по тому же субстрату Suc-Ala-Ala-Pro-Phe-pNA у SerPH122 была в 600 раз ниже. Протеолитическая активность недавно была описана у гомолога сериновой пептидазы из яда эктопаразитоида Scleroderma guani. Активный центр белка содержал аминокислотные остатки Ser-Asp-Ser вместо типичной каталитической триады сериновых пептидаз His-Asp-Ser. Гомолог проявлял трипсиноподобную активность при гидролизе субстрата Bz-Arg-pNA (Nα-бензоил-Arg-(п-нитроанилида)) [6].
Рис. 5.
Протеолитическая активность гомолога SerPH122 в гликозилированной (SerPH122g) и дегликозилированной (SerPH122dg) формах по хромогенному субстрату Suc-Ala-Ala-Pro-Phe-pNA. 1 – исходный пробелок (proSerPH122), 2 – процессированный зрелый белок после обработки трипсином (SerPH122).

Значение рН, оптимальное для работы гомолога SerPH122, определяли в соответствии с физиологическими уровнями кислотности в кишечнике личинок T. molitor: рН 5.6 в передней части и рН 7.9 в задней части средней кишки [23]. На рис. 6 показано, что активность SerPH122 выше при значении рН 7.9.
Рис. 6.
Зависимость удельной активности (нмоль/мин мг) процессированного гомолога SerPH122 от значения рН: 1 – рН 5.6; 2 – рН 7.9.

Таким образом, разработан метод получения рекомбинантного белка-гомолога сериновых пептидаз proSerPH122 T. molitor, подобраны условия его очистки с использованием металл-хелатной аффинной хроматографии, проведены дегликозилирование эндогликозидазой Endo H и процессинг пробелка до зрелой формы. Показано, что: (1) полученный гомолог подвергался процессингу под действием трипсина, (2) проявлял невысокую, но достоверно детектируемую химотрипсин-подобную активность при использовании в качестве субстрата Suc-Ala-Ala-Pro-Phe-pNA, причем при рН 7.9, в слабощелочных условиях, характерных для содержимого задней части средней кишки личинок T. molitor, активность SerPH122 значительно выше, чем при рН 5.6, в условиях содержимого передней части средней кишки, (3) гликозилирование не оказывало влияния на изученные свойства. Настоящая работа вносит вклад в исследование малоизученной области функционирования псевдопептидаз и может послужить основой для выяснения связи между структурой и функцией сериновых пептидаз и их гомологов.
Авторы выражают благодарность к. х. н. М.В. Серебряковой за проведение масс-спектрометрического анализа препарата рекомбинантного гомолога proSerPH122.
Исследование выполнено при финансовой поддержке гранта Российского научного фонда (проект № 19-74-00080).
Список литературы
Rawlings N.D., Barrett A.J., Thomas P.D., Huang X., Bateman A., Finn R.D. // Nucleic Acids Res. 2018. V. 46. № D1. P. D624–D632. https://doi.org/10.1093/nar/gkx1134
Brown J.R., Hartley B.S. // Biochem. J. 1966. V. 101. № 1. P. 214–228. https://doi.org/10.1042/bj1010214
Perona J.J., Hedstrom L., Rutter W.J., Fletterick R.J. // Biochemistry. 1995. V. 34. № 5. P. 1489–1499. https://doi.org/10.1021/bi00005a004
Perona J.J., Craik C.S. // Protein Sci. 1995. V. 4. № 3. P. 337–360. https://doi.org/10.1002/pro.5560040301
Wex T., Lipyansky A., Brömme N.C., Wex H., Guan X.Q., Brömme D. // Biochemistry. 2001 V. 40. № 5. P. 1350–1357. https://doi.org/10.1021/bi002266o
Wu C.Y., Huang J.M., Zhao Y.J., Xu Z.W., Zhu J.Y. // Toxicon. 2020. V. 183. P. 29–35. https://doi.org/10.1016/j.toxicon.2020.05.011
Pils B., Schultz J. // J. Mol. Biol. 2004. V. 340. № 3. P. 399–404. https://doi.org/10.1016/j.jmb.2004.04.063
Yang L., Lin Z., Fang Q., Wang J., Yan Z., Zou Z., Song Q., Ye G. // Dev. Comp. Immunol. 2017. V. 77. P. 56–68. https://doi.org/10.1016/j.dci.2017.07.014
Reynolds S.L., Fischer K. // Biochem. J. 2015. V. 468. № 1. P. 17–24. https://doi.org/10.1042/BJ20141506
Bryant B., Ungerer M.C., Liu Q., Waterhouse R.M., Clem R.J. // Insect Biochem. Mol. Biol. 2010. V. 40. № 7. P. 516–523. https://doi.org/10.1016/j.ibmb.2010.04.011
Martynov A.G., Elpidina E.N., Perkin L., Oppert B. // BMC Genomics. 2015. V. 16. № 75. https://doi.org/10.1186/s12864-015-1306-x
Naumov G.I., Naumova E.S., Tyurin O.V., Kozlov D.G. // Antonie Van Leeuwenhoek. 2013. V. 104. № 3. P. 339–347. https://doi.org/10.1007/s10482-013-9956-7
Горбунов А.А., Акентьев Ф.И., Губайдуллин И.И., Жиганов Н.И., Терещенкова В.Ф., Элпидина Е.Н., Козлов Д.Г. // Биотехнология. 2020. Т. 36. № 6. С. 78–86. https://doi.org/10.21519/0234-2758-2020-36-6-78-86
Матвеева А.Ю., Губайдуллин И.И., Федоров А.С., Козлов Д.Г. // Биотехнология. 2019. Т. 35. № 5. С. 3–11. https://doi.org/10.21519/0234-2758-2019-35-5-3-11
Kozlov D.G., Yagudin T.A. // Biotechnol. Lett. 2008. V. 30. № 9. P. 1661–1663. https://doi.org/10.1007/s10529-008-9739-9
Sievers F., Wilm A., Dineen D., Gibson T.J., Karplus K., Li W., Lopez R., McWilliam H., Remmert M., Söding J., Thompson J.D., Higgins D.G. // Mol. Syst. Biol. 2011. V. 7. P. 539. https://doi.org/10.1038/msb.2011.75
Nicholas K.B., Nicholas H.B. Jr., Deerfield D.W. II. // Embnew. News. 1997. V. 4. P. 14. http://www.psc.edu/biomed/genedoc
Almagro Armenteros J.J., Tsirigos K.D., Sonderby C.K., Petersen T.N., Winther O., Brunak S., von Heijne G., Nielsen H. // Nat. Biotechnol. 2019. V. 37. № 4. P. 420–423. https://doi.org/10.1038/s41587-019-0036-z
Терещенкова В.Ф., Клячко Е.В., Беневоленский С.В., Белозерский М.А., Дунаевский Я.Е., Филиппова И.Ю., Элпидина Е.Н. // Прикл. биохимия и микробиология. 2019. Т. 55. № 3. С. 231–236.
Frugoni J.A.C. // Gazz. Chem. Ital. 1957. V. 87. P. 403–407.
Parry M.A., Fernandez-Catalan C., Bergner A., Huber R., Hopfner K.P., Schlott B., Gührs K.H., Bode W. // Nat. Struct. Biol. 1998. V. 5. № 10. P. 917–923. https://doi.org/10.1038/2359
Hedstrom L. //Chem. Rev. 2002. V. 102. № 12. P. 4501–4524. https://doi.org/10.1021/cr000033x
Vinokurov K.S., Elpidina E.N., Oppert B., Prabhakar S., Zhuzhikov D.P., Dunaevsky Y.E., Belozersky M.A. // Comp. Biochem. Physiol. B. Biochem. Mol. Biol. 2006. V. 145. № 2. P. 126–137. https://doi.org/10.1016/j.cbpb.2006.05.005
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


